Degronを探せ
Grüezi,
今日は勉強したことのメモ書きを少し。
PROTACsのようなdegraderの文献を読んでいると、時々Molecular Glueというのを見かけます。PROTACがbifunctional moleculeであるのに対して、Glueは、例えばCereblonに結合したIMiDsの先にPOIのPPIを誘起します。PROTACのデザインはあくまで鍵穴モデルの延長であるのに対して、GlueはまだLogical designが難しいと言われています。というわけで、どれくらい難しそうかを見てみましょう。
今回はこちらの文献を参考にしてます。
ACS Med. Chem. Lett. 2019, 10, 1592-1602.
PDB ID: 5HXB 5FQD 6H0F
これらのCereblon, IMiDs, それとPOI (CK1a, GSPT1, and Ikaros/Aiolos)の部分構造を重ね合わせたのがこちら。

リガンドのIMiDsとCereblonの結合部位が割とよく重なっていると思いますが、POIはバラバラです。次に少し拡大してpolar contactを表示したのがこちら。

リガンドはPOIと相互作用してませんね...。Cereblonを消してもう少し近寄ります。

IMiDsの左側にGlyを含むループ構造がそれぞれ見てとれるかと思います。Cartoonだとよく重なって見えますが、側鎖を表示するとGly以外は全く違う配列です。

このように、「構造的かつ機能的に一見関係ないが、鍵となる位置にGlyを含む特定のループ構造」をDegronと呼びます。Molecular GlueはSubstrate Receptorに対して、このDegronを持つ基質タンパクのPPIを誘起するそうです。
「Backbornの特徴が似ているだけのPOIとPPIを引き起こす分子を設計」することなんて出来るんでしょうか...。これがMolecular Glueの発見がSerendipityに依存すると言われる所以です。
Molecular GlueはPROTACと比較して圧倒的にDrug-likeと言えますが、PROTACの方がこれまでの創薬経験の延長で戦えるので、参入障壁が低いと言えると思います。
ただし、Zinc Finger Degronのように、Ubiquitinationを受けることが知られているドメインもいくつか報告されています。
KNIMEでText miningでも勉強した方がいいかもですね。
(訂正)今回紹介した3つのPOIがそもそもzinc fingerを持ってるっぽいですね。素人丸出しでごめんなさい。次までに勉強しておきます。
alias を作って快適PyMOL生活
どこかで一度話題にした気もしますが、PyMOL(python?)のコマンドにはデフォでaliasという機能が組み込まれてます。簡単に言うと、コマンドラインに2,3文字入れてtabキーを叩くと、該当するコマンドを補完するか、候補のコマンドを表示してくれます。
実はこれ、自分でも作れます。
今回はこちらのページを参考にしました。
今回の内容はざっとこんな感じです。
Alias example
alias xxx, yyyみたいなコマンドを打つと、次からxxxと言うコマンドでyyyのFunctionになります。
何言ってんだよって感じなので、実際にやってみましょう。
今回参考にした文献は、たまたま読んだこちらです。
(2019) Nat Chem Biol 15: 672-680
とりあえずPDB ID: 6HR2 と6HAX, 6HAYを読み込んでおきます。

続いて、次のコマンドを実行します。
alias nice, as cartoon; show sticks, organic; show spheres, inorganic
ええ、まんまサイトからパクってます。
次にコマンド niceを実行すると…

と言う具合にalias nice, 以降のコマンドをまとめて実行してくれるわけです。
今回のコマンドは水が消えただけですね。
へぇ、それで?
ルーチンのコマンドをPyMOL>File>Edit pymolrcに書き込んで、デフォルトでエイリアス化してしまいましょう。こんな感じです。(#keetane should be replaced with your own user name)
Alias example for default setting at .pymolrc.pml
cd /Users/username/Desktop
set cartoon_transparency, 0.5
set transparency, 0.6
set surface_color, white
set sphere_scale, 0.2
set label_color, gray
alias al, alignto; zoom
alias sp, split_chains enabled; alignto; zoom
alias cc, util.color_chains("(enabled and name CA)",_self=cmd)
alias see, zoom byres organic around 8 and chain A
alias sol, as cartoon; show sticks, organic; show spheres, inorganic organic
alias del, as cartoon; show sticks, organic; show spheres, inorganic organic solvent
alias line, show lines, byres organic around 4
alias hb, select ligand, organic and enabled; cmd.dist("ligand_polar_conts","(ligand)","(byobj (ligand)) and (not (ligand))",quiet=1,mode=2,label=0,reset=1);cmd.enable("ligand_polar_conts")
alias well, show surface, byres organic expand 4
alias 5, create a5, byres organic expand 5 and enabled; disable !a5
alias 4, create a4, byres organic expand 4 and enabled; disable !a4
alias tag, label enabled and name ca, "%s%s" % (resn,resi)
alias untag, hide label
alias pick, cmd.mouse('forward')
alias ch, capture; save image.png
Practice
実際に使ってみましょう。
新たに作ったalを実行するとこんな感じです。

タンパクを重ね合わせてzoomしました。
次にccを実行して主鎖ごとに色を変えます。

さらにseeを実行してligandにzoomします。

del/solの実行で溶媒の表示を切り替えます。

今回のaliasは基本的にアクティブになっているオブジェクトに適応されるので、今は3つのオブジェクトの水が全部表示されててごちゃごちゃしてます。
disable !6HAY
で6HAY以外を非表示にします。

lineを実行して周辺残基を確認します。

hbを実行してリガンドからのpolar contactを表示します。これは、Action>find>polar contacts>to other atoms in objectと同じに設定してあります。

tag/untagの実行で側鎖のラベルの表示を切り替えます。
例えばここの水酸基はS111とH115が相互作用してそうです。

wellを実行してポケットのくぼみを観察してみます。

pickを実行してマウスモードを切り替えます。3-bottun Editingに切り替えて原子をPickしていくと、原子間距離、角度、二面角の順番に表示してくれます。



今まであんなにコマンド打ってたのはなんだったの…というくらいにはサクッと出来ました。
Hint: 今回のコマンドはsol, del, line, wellは全てのオブジェクトに適用されます。
前回記事で水分子を上手く扱えないかなぁとモヤっとしてたので、自己解決出来ました。
なるほど。
Pymol上の溶媒分子をどうするか
Grüezi,
最近湿度が高いですね。電車に乗るたびにもっとPhysical distanceが欲しいと感じて止みません。
そんな訳で今回のお題は溶媒、つまり水分子です。
Pymol上でいくつかのPDBファイルを重ねて表示することがあると思います。その時に、溶媒の水分子ってごちゃついて鬱陶しいことがあります。しかし彼らは、こちらから指定してあげないと表示の切り替えは出来ません。という訳で水分子をどう扱うか、特にコマンドを使って出来る方法を少しまとめてみます。
今回参考にした文献は、たまたま読んだこちらです。
J. Med. Chem. 2015, 58, 20, 8182
PDB ID: 6CEP と6CEOを読み込んでおきます。

それぞれ表示したら水の位置が違いますよね。


溶媒を一括で削除する
delete solvent (and enabled)
and enabledがないと、セッション全体に適用されます。復元できないので、水を確認したい時には手遅れになります。
溶媒単独のオブジェクトを作る
create solvent, solvent (and enabled)
extract solvent, solvent (and enabled)
前者は溶媒のオブジェクトを別個に作成しますが、表示のon/offには使えません。後者は抜き出すので、オブジェクトのon/offで表示を切り替えられます。表示のon/offはenable/disable コマンドです。

溶媒単独の選択オブジェクトを作る
select sol, solvent
hide everything, sol
show spheres, sol
選択オブジェクトを作成し、hideとshowで切り替えます。
対象をsession全体にしておけば、objectごとに切り替える必要はないかと思います。コマンド打つのめんどいですが。


MaestroのSplit風に管理する
(disable !5cep)
extract s5cep, solvent and 5cep
extract l5cep, organic and 5cep
group g5cep, enabled
だから何って話ですが、PDBごとに溶媒を管理しようと思うとこんな感じです。


色が変わったのはPyMOLが落ちただけなので気にしないでください…
Presetを使う(番外編)
ligand site > cartoon
コマンド使ってませんが、すっきり見える方法の一つです。


インハウスで複合体共結晶がたくさん取れてきた時とか、皆さんどうしてるんですかね?
いい方法があったら教えてください。
PDBファイルを検索結果のターゲット分子ごとに割り振ってダウンロードするKNIME WorkFlow
Grüezi,
すっかり投稿するのを忘れてた記事を一つ。
これまで紹介したNodeを使うと、 PDB ConnetcorのReport機能を使ってターゲット分子ごとにPDBを割り振ってダウンロードすることも簡単にできます。
今回はスクショメモしか残してなかったのでかなりざっくりですが、 内容はこんな感じです。
WorkFlow 全体
今回新たに紹介する nodeは多分ありません。上段にはいつも通りリガンドの画像付きExcelにまとめレポートを、下段にはターゲット分子ごとにPDBを振り分けるフローを示してます。

レポートはこんな感じでExportされます。ごめんなさい今回のキーワードは何にしたか忘れました。。。

Table Row To Variant
レポート中のRowを変数に設定します。今回はMacromolecule Nameを選択することで、Excel上ではシートに、PDBファイルはフォルダにそれぞれ割り振りできます。

String Manipulation (Variable)
このNodeを使うことで変数をStringに組み込めます。PDBファイルをダウンロードしたいDirectory名を変数を使って生成します。

Excel Sheet Appender
これもいつも通りSheet nameを変数設定することで、ターゲット分子名ごとにシートに情報を振り分けます。

Export結果
以下、こんな感じです。ソースが違っても分子名が同じであれば同じDirectoryに保管されます。

今回は自分用のWorkFlowの覚書でした。
あ、そういえば日本に帰ってきました。
苦労話はしても疲れるだけなのでしませんが、あーあ帰ってきちゃったなって感じです。
もう少しMacBookAir2012Midで頑張ることになったけど、勉強続くかな…
Dear Pymol Beginners x KNIME 1.4 -リガンドをクラスタリングして,レポートとPDBファイルのダウンロード先の振り分けに使ってみたり-
Grüezi,
早いもので,帰国まで1ヶ月を切ってしまいました.慣れてしまえばもう少し滞在したいような,でも物価の高さには相変わらず目眩もするし,正直多分虫歯が出来たっぽいしで帰国したいような,そんなどっち付かずの心持ちです.計画してた旅行の残りはコロナウィルスのせいでオールキャンセルなので,やっぱり心残りだなぁ.
さて,最近もっぱらVirtual Screeningなんかをやってるんですが,Libraryが大きくてなかなか終わりません.という訳で,KNIMEを使って合間に出来るPDBの情報収集方法の備忘録です.
今回はざっくりこんな感じのことが出来ます.
- RSC PDB Bankのサイトから取得したPDBの情報からリガンドをClusteringする
- Clusterごとの情報をExcelにExportする
- PDBをClusterごとにフォルダーにダウンロードする
今回紹介するNodeはこちら
- Workflowの全体像
- PDB connector
- Row Filter
- Fingerprints
- Distance Matrix Calculate
- k-Medolds
- Group Loop Start
- Value Counter
- TableRow To Variable
- String Manipulation (Variable)
- Excel Sheet Appender
- Variable Loop End
- Create Directory
- String Manipulation (Variable)
- PDB Saver
- Loop End
- Excel Reportからリガンドの確認
今回も,す(@sumtat_)さんのところのブログを大いに参考に(パクって)しています.いつも勉強になりますありがとうございます.
そんな訳で,今回も行ってみましょう.
Workflowの全体像
Workflowの左側でPDB情報を取得,Filtering,Clusteringを行なっています.分岐した上段では,クラスターごとに情報をグループ化してExcelにシートごとにExportしています.下段では,クラスターごとにDirectoryを作ってPDBファイルをダウンローとしています.
青で囲ったClusteringの部分が今回の肝ですが,思いっきりパクって使わせてもらってます.緑で囲った部分はClusterの命名に便利だなと思ったので採用(つまりパクリ)しましたが,他にいいアイディアがあれば代用していいと思います.つまり何が言いたいかというと,詳細な説明は先達に譲ります(!).

PDB connector
今回はIDs and KeywordsタブのText Searchに"SARS-CoV-2 main protease"と入力しています.Report OptionsはStructure Summary, Ligands, Binding Affinity, Primary Citationをチェックしています.
Row Filter
MW<100のリガンドをFilter Outしています.目的としては溶媒や添加剤,apo体を除くことです.なので,分子量をもう少し大きくしてもいいかもです.

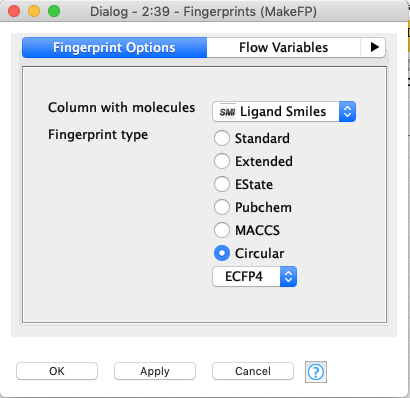
Fingerprints
LigandのいわゆるFingerprintを作ってくれます.設定はパクったので,ぶっちゃけ詳細は説明できません...
Distance Matrix Calculate
同じく詳細は割愛します.
ざっくり言うと,前項で作ったFingerprintの距離を総当たりで計算して吐き出す,みたいなNodeらしいです.かの有名なTanimoto法でお願いします.

k-Medolds
このNodeでClusteringしてくれます.Partition Countで発生させるクラスターの数を選べます.今回はまんまパクって5個にしてます.

Group Loop Start
発生させたClusterを元にGroup化を繰り返すLoop Nodeです.上段も下段も設定は同じです.
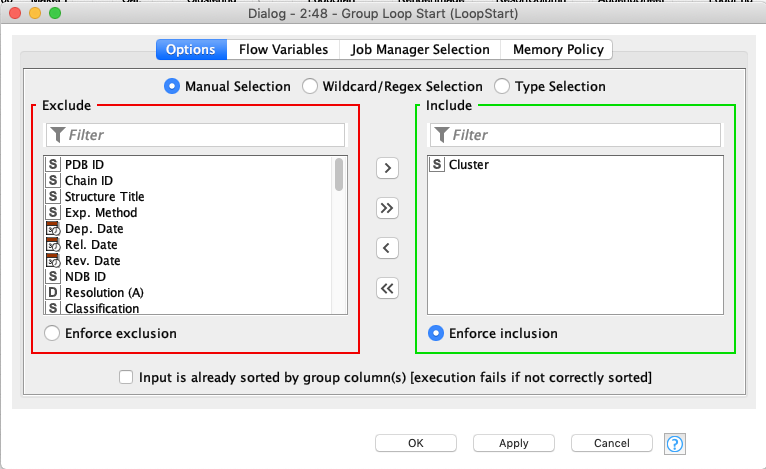
Value Counter
Group化したClusterの中にあるLigandの数を数えます.

TableRow To Variable
Countした数を変数に変換します.特に設定不要です.
String Manipulation (Variable)
前項で発生させた変数を受けてStringを発生させます.今回は数えた数をそのままExcelのシート名にします.はい,まんまパクってます.
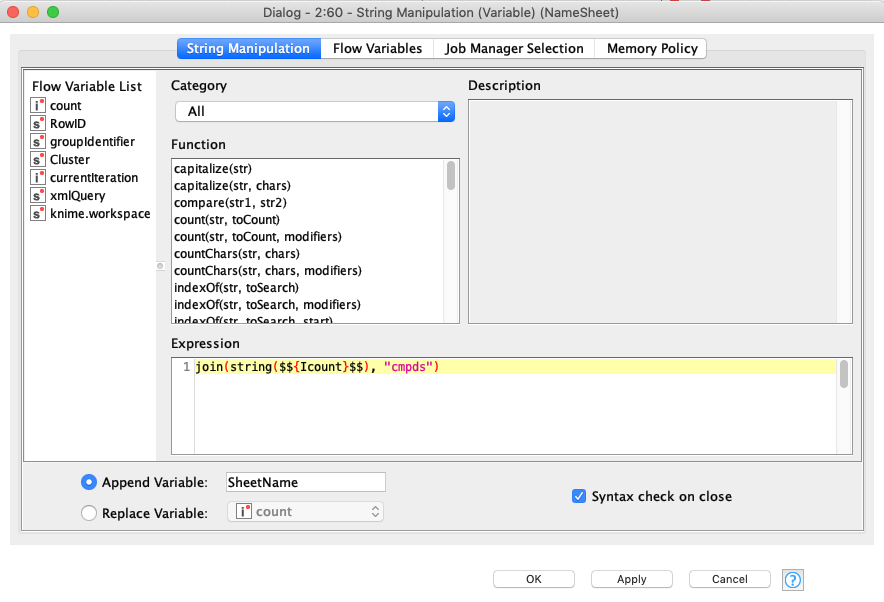
Excel Sheet Appender
ExcelにPDB情報をシートごとにまとめます.File名は自由に設定して,シート名を前項までに作った変数にします.add column headersにチェックしておいた方がいいです.

Variable Loop End
Loopの終わりを指定してあげます.
Create Directory
ClusterごとにPDBファイルを振り分けるDirectory(フォルダ)を作りたいのですが,ちょっとTrickyです.
Locationは前述のExcel Reportと同じ場所を指定しておきます.フォルダ名に変数を指定したいのですが,なぜかここに変数指定ボタンがないので,適当なダミー名(今回はa)を入力した上でFlow Variableのタブから改めて変数を指定します.
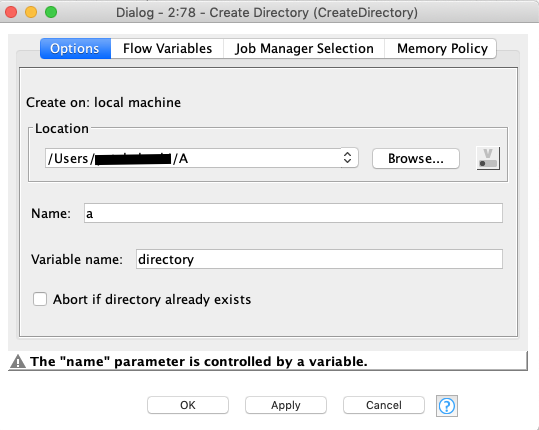
Directory名をTable To Variableで発生させたカラム名に指定します.

実行結果はこんな感じになればOKです.directory名に指定した変数(今回はクラスター内のLigandの数+cmpds)が反映されています.

String Manipulation (Variable)
前項で発生させたdirectoryのValueをPDBの保存先に指定できればよかったのですが,残念ながら"file:"という文字列が先頭に付いてるのでPDB Saverの保存先に使えません.そこで,いつものString Manipulationで保存先を発生させます.Sheetnameを変数に使えばOKです.

PDB Saver
保存先として,Parent Directoryの変数に先ほど発生させたdirectoryを指定します.

保存に成功すると,実行結果の右端にtrueと表示されます.何かしらエラーがあった場合はFailsになります.
Loop End
Loop処理の終了を宣言します.
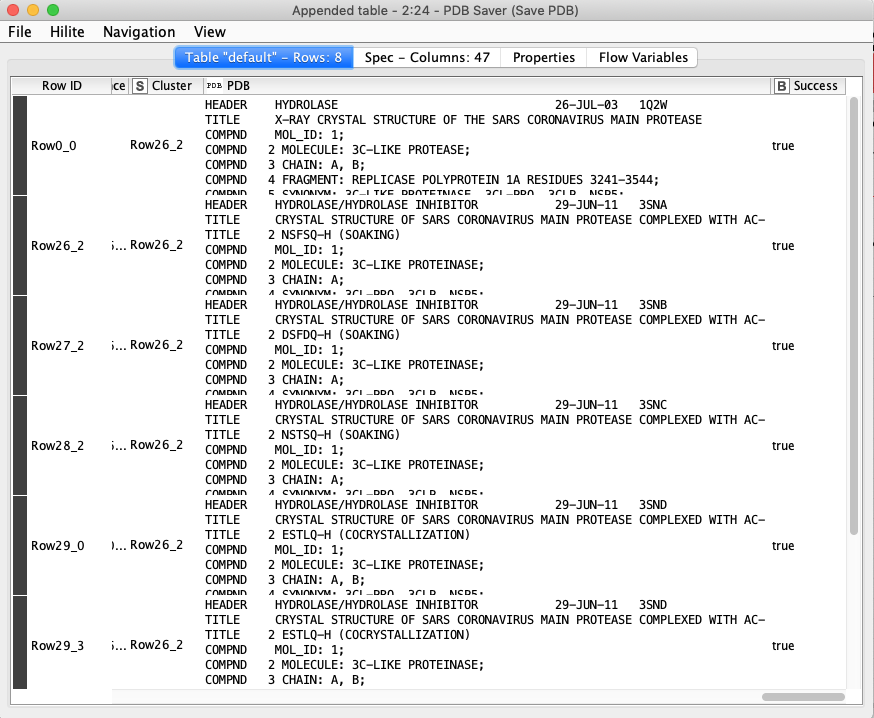
実行結果
2020/04/21現在で,実行結果はこんな感じになります.ちゃんと5つのClusterに分かれてPDBファイルが保存されました.あれ?11化合物あるはずなのにファイルは8つ?これは一つのファイルの中に,例えばA鎖とB鎖がある場合,リガンド数が2個とカウントされるためです.実際には一つのPDBファイルの中に格納されています.

PDB Saverの実行結果を見ると納得かと思います.
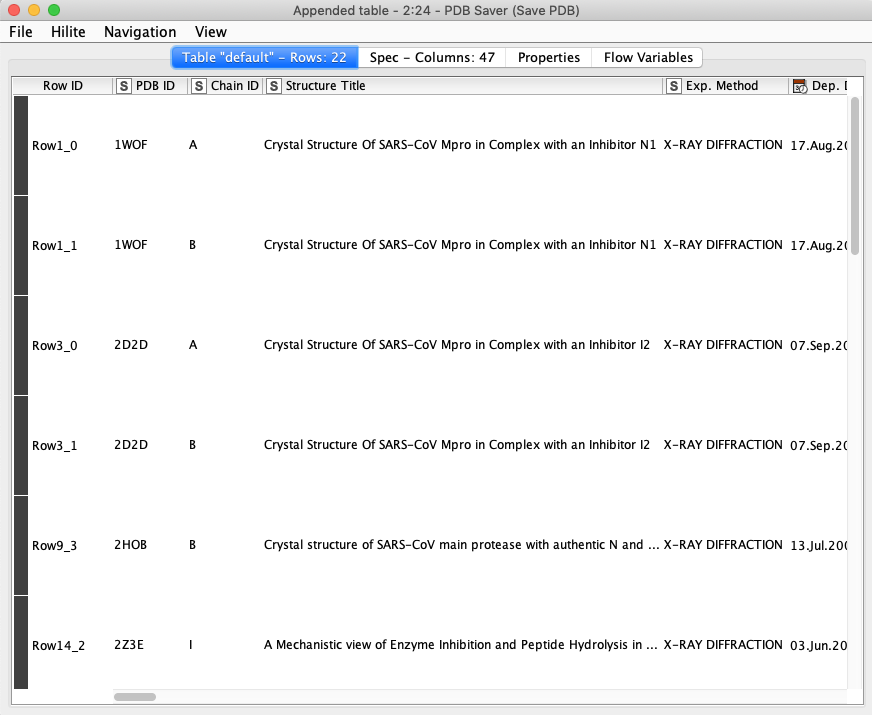
Excel Reportからリガンドの確認
さて,Excelを開いてみましょう.
ちゃんと5つのClusterごとにシートが作られてますね.数が一番多い60cmpdsのシートには,先日紹介したX-Chemのフラグメントスクリーニングの結果が含まれています.ExcelのFilter機能を使って絞り込むと,他にもリガンドが見つかりますね.前々回の記事で紹介したリガンドは,こんな感じで見つけてきました.

ついでに22cmpdsのシートも開いてみると,ここにはペプチドミミックのリガンドがまとまってました.中身がさっぱりわかってなくても,Clusteringが結構うまくいってるんだなぁと関心です.
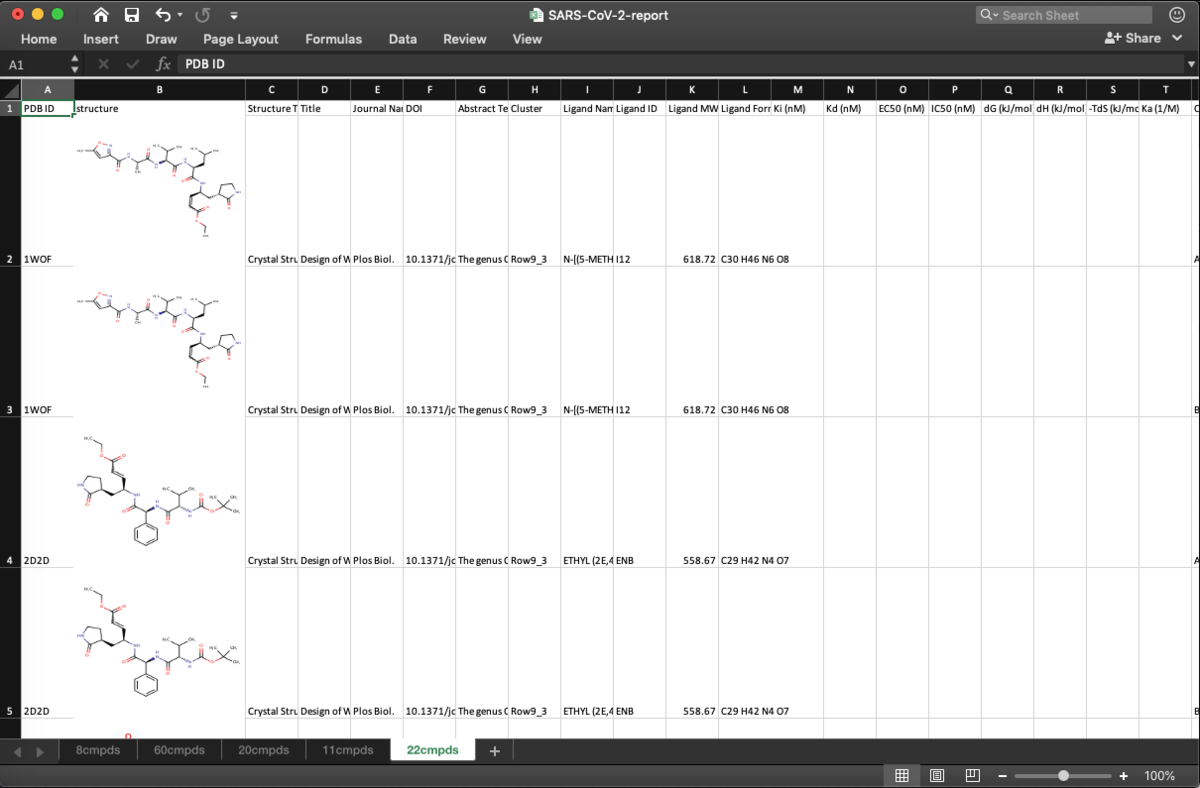
こんな感じでClusterごとに振り分けたPDBファイルを必要な分だけPymolに読み込んでGroup化してやると,プロジェクト初期には有用だったりしないかなと思います.
今回みたいに取得した情報の振り分けを自動化出来ると,特許解析とかに応用出来ないのかなとか妄想中です.
ご参考までに.
追記)後から気が付いたんですが,Creat DirectoryとかしなくてもString Manipulationで保存Directoryを指定してやれば勝手にDirectoryが作成されました.まぁ,Variableの練習をしたってことでご愛嬌.
Dear Pymol Beginner 5 -Pymolのデフォルトを変更してみたり-
Grüezi,
何てことない話なんですが,Pymolのデフォルトセッティングを紹介します.
cd /tmp
set sphere_scale, 0.2
set cartoon_transparency, 0.5
set transparency, 0.6
set surface_color, white
File > Edit pymolrcを開いて上記のTextをコピペ,閉じる時にSaveするだけです.
以下,具体的に比較するとこんな感じです.左がpymolrcなし,右がありです.
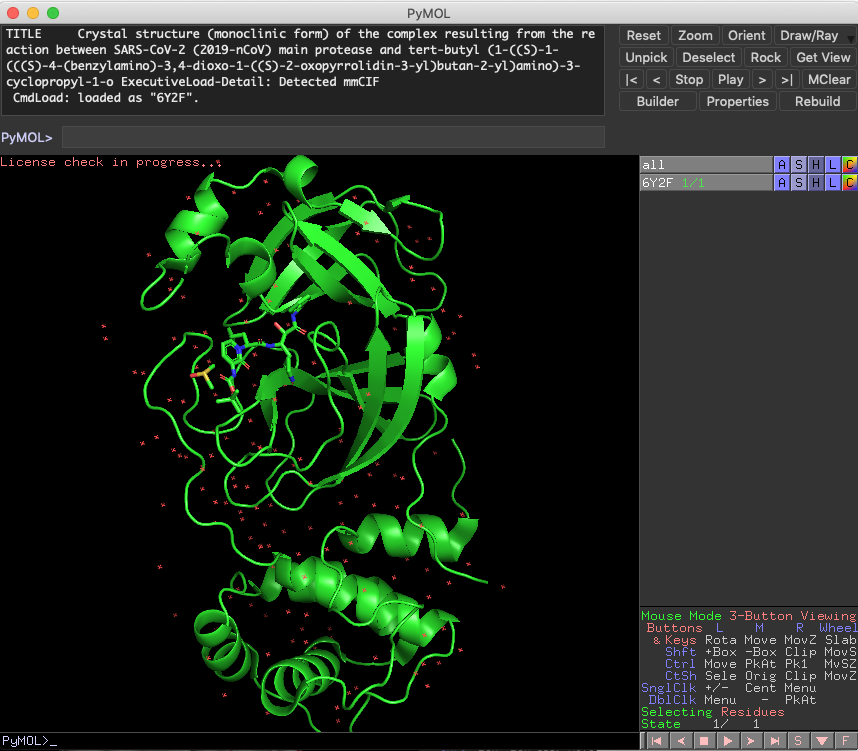
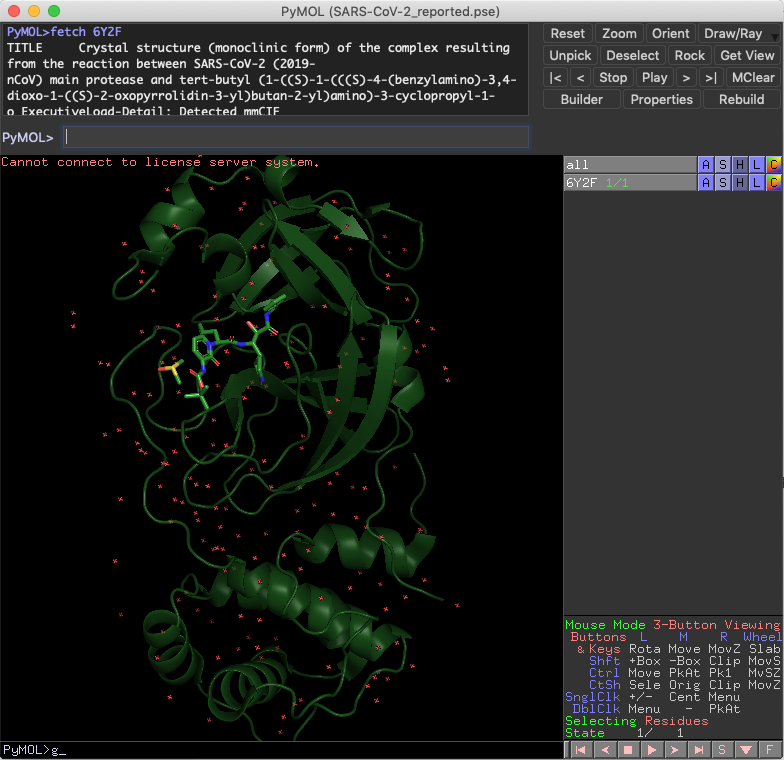
show surfaceすると思いっきり見た目は違います.左は毒々しいですね...
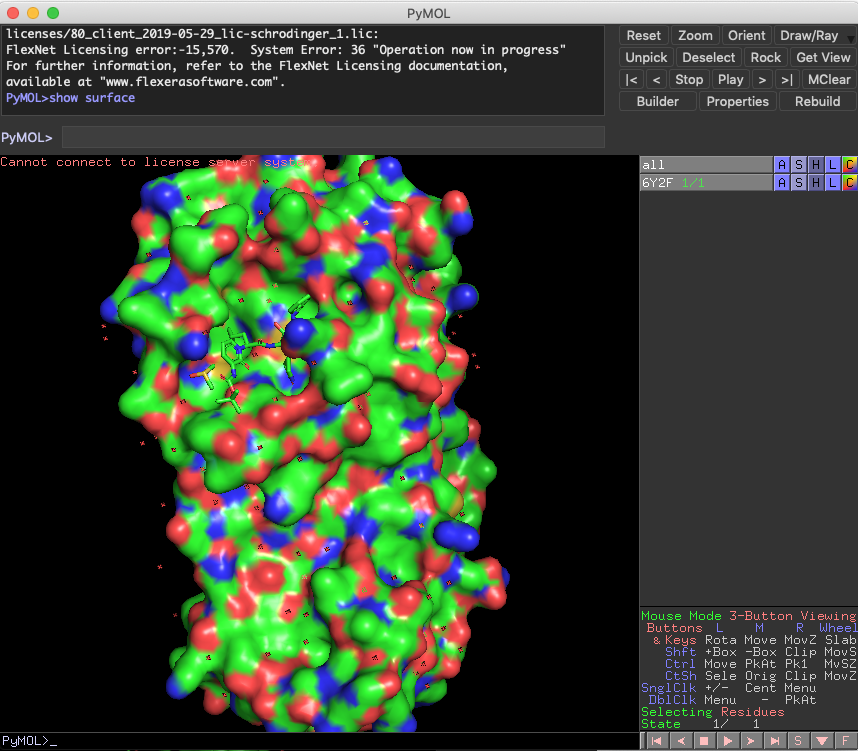
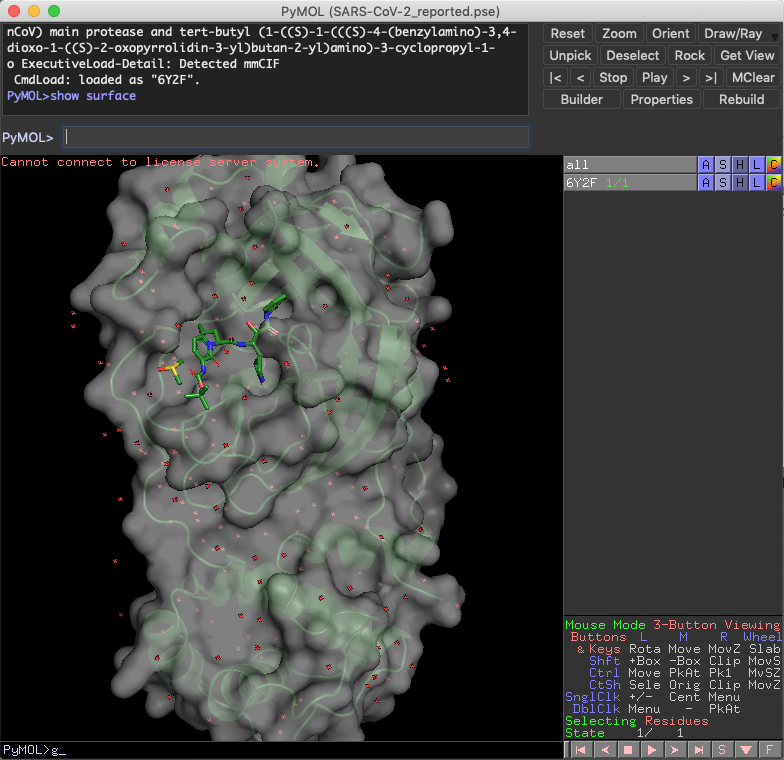
ポケット周辺も具合はだいぶ違います.

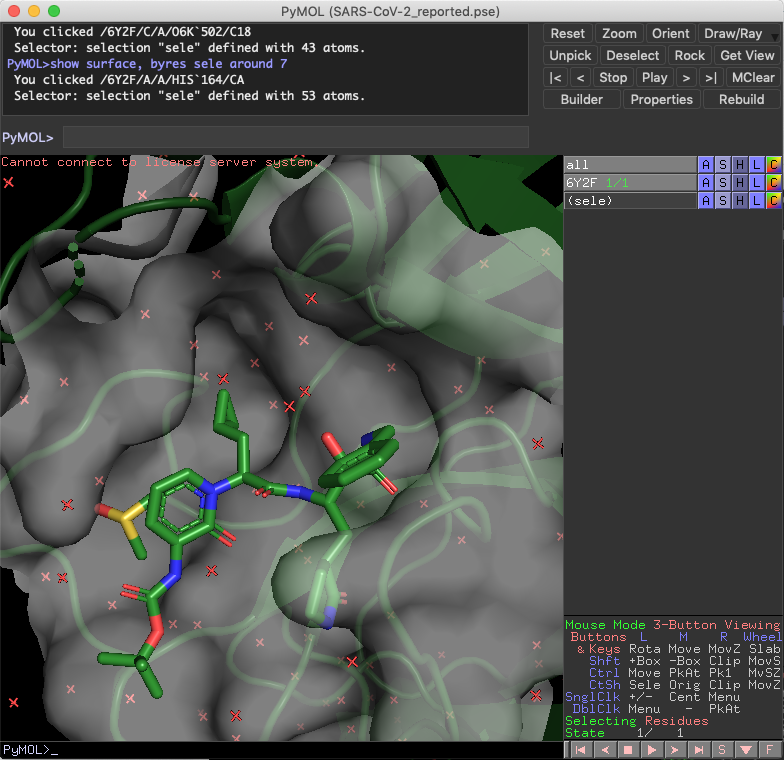
ポケット周辺のアミノ酸残基をshow linesしておくと,相互作用が取れそうな残基が見えるかもしれません.影があった方が隙間が見えて変換の余地を把握出来そうな気がします.
最後にcd /tmp ですが,fetchなどで取ってたきたcif fileの保存先を一時フォルダに変更しています.何もしないとWorking Directoryはユーザー直下になっていて,ふとした時にmessyになってることに気がつきます.
ご参考までに
Dear Pymol Beginners 4 -オブジェクトをグループ化してみたり,リガンドの結合位置や様式を比べてみたり-
Grüezi,
世界中でロックダウンが行われていますが,日本も複数の都市で行われ始めましたね.経済的な影響も心配ですが,何よりもまずは生き残ることだと思います.株の格言でも似たようなのがありましたよね.
さて,SARS-CoV-2の研究だけは活発に行われているわけですが,私も例に漏れず今回をきっかけにターゲットとしてどんなもんなの?と勉強を始めたクチです.折角なので,前回までに使用したKNIMEの実行結果も参考に,解析方法を紹介したいと思います.
今回の覚書はこちら.
- 複数の結晶構造が報告されている同一ターゲットについて,Pymol上でグループ化する.
- それらを纏めて重ね合わせる.
追記
- リガンドの結合部位を俯瞰する
今回使用するコマンドの一覧はこちら
- group covalent, enabled
- disable covalent
- group non-covalent, enabled
- disable non-covalent
- group others, enabled
- enable all
- Fetch 2Q6D 6Y2F 2GZ7
- Zoom
- Alignto 2GZ7
- disable !2GZ7
- zoom byres organic around 5
- disable all enable 6Y2F
- disable all
- enable 6Y2F 2GZ7
- set grid_mode, 1
- show surface, byres organic around 5
- set surface_color, gray
- hide surface
- set grid_mode, 0
- disable 6Y2F
- disable 2GZ7
- enable 6Y2F
- enable all
- hide cartoon
今回はついでに,XChemから公開されているFragment ScreeningのPDBファイルも見てみたいと思います.こちらからダウンロード出来るので,covalent, non-covalent, all othersのカテゴリごとにダウンロードしておくといいと思います.SummaryもExcelファイルにまとまってます.構造情報がSMILESだなんて,infomatician friendlyですね!Chemist FriendlyになるようなKNIME WorkFlowも簡単なので紹介すればよかったな...
ファイルの準備が出来たらPymolを開いておきましょう.そしてまずはCovalentのPDBファイルをがっつりDrag&DropでImportしてください.

さすが!なんの処理もなしにタンパクが重ねわせしてあるので,とっても綺麗に見えますね.
オブジェクトパネルに注目すると,こんな感じです.

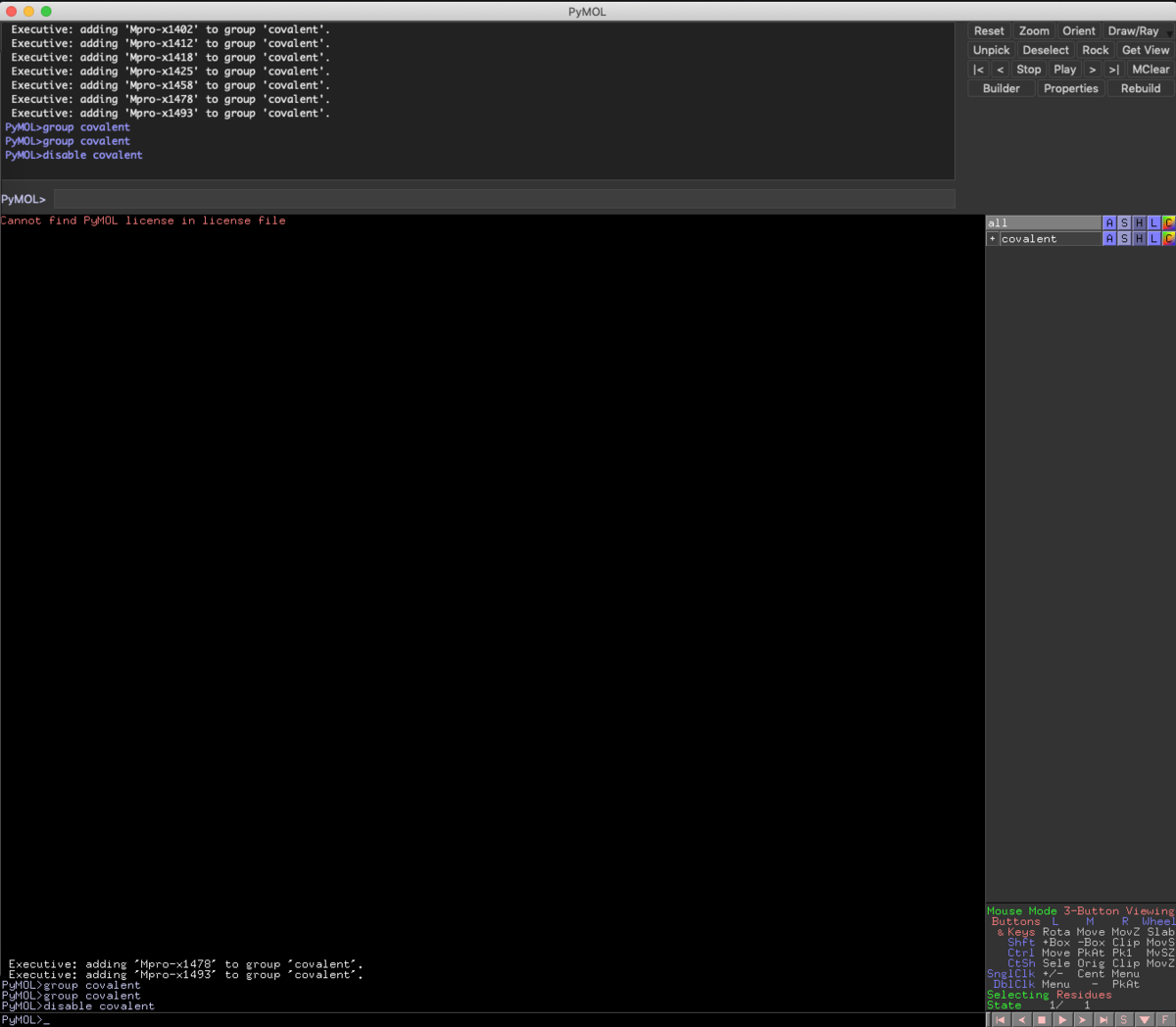
さて,これをコマンド一発でグループ化しましょう.
group covalent, enabled
こんな感じでグループ化されました.

ちなみにグループ名の左にある+をクリックするとツリーを開閉出来ますが,group covalentとコマンドを入力しても同様にツリーの開閉が出来ます.
disable covalent
disableコマンドでグループの非表示化も出来ます.
次にnon-covalentのPDBファイルをがっつりimportするとこんな感じです.

group non-covalent, enabled
今度は表示されているオフジェクトをgroupコマンドでnon-covalentとしてグループ化します.
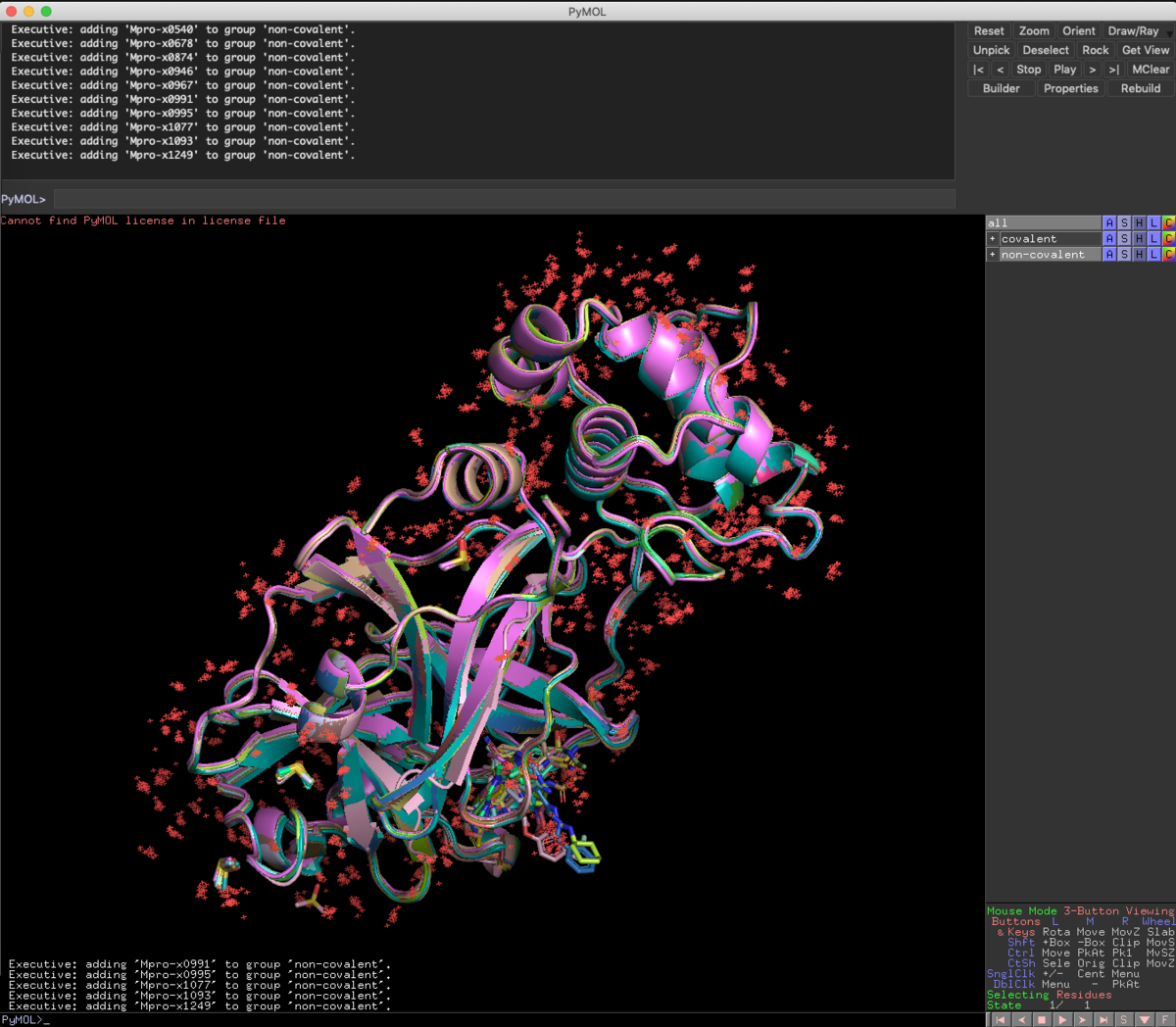
disable non-covalent
先ほどと同様に,一旦全て非表示にします.
続いて,othersのPDBをimportしてきます.

group others, enabled
othersもグループ化しておきます.
enable all
試しに全オブジェクトを表示にすると,全てのPDBファイルが重ね合わせてありますね.ありがたやー.
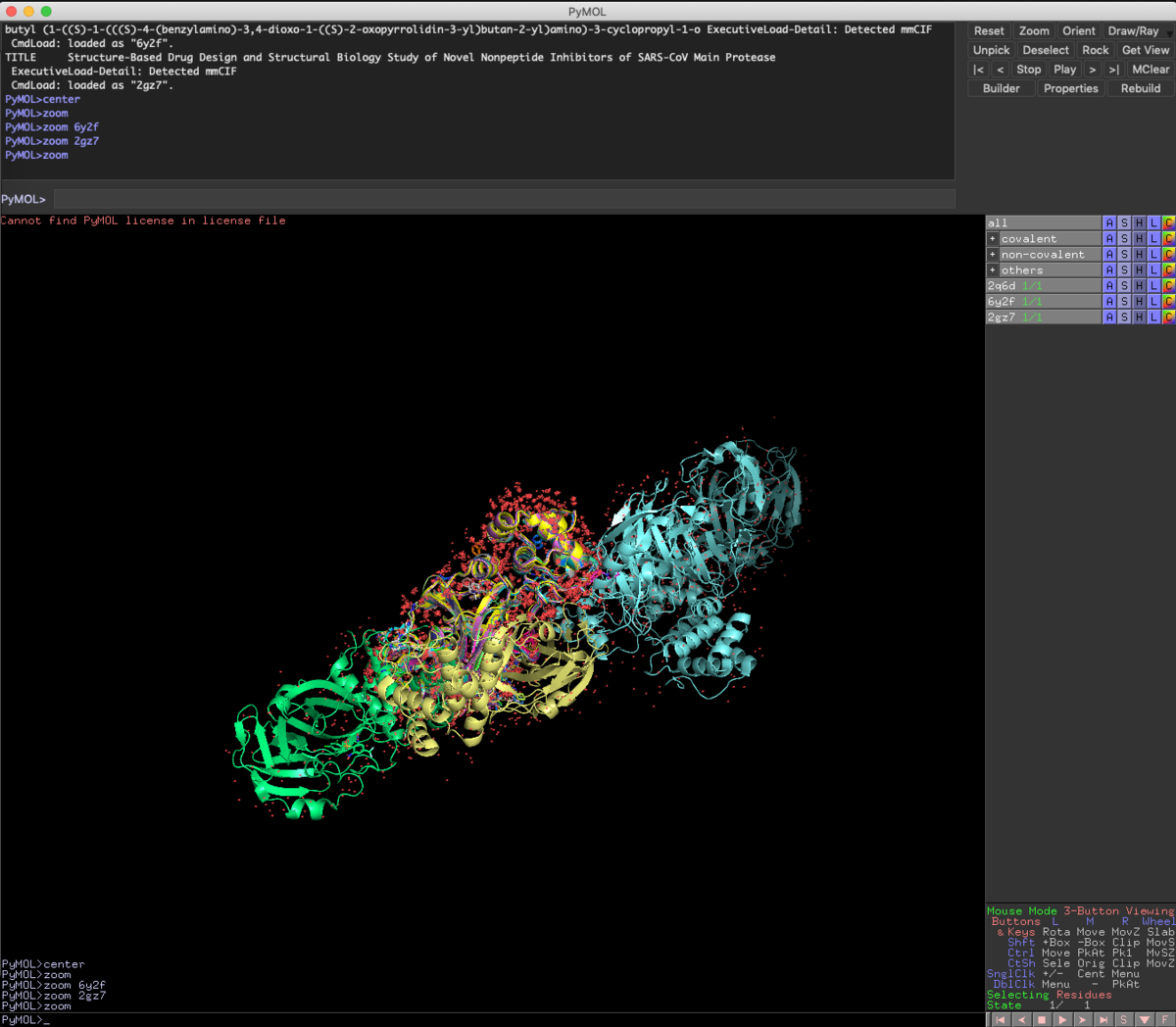
Fetch 2Q6D 6Y2F 2GZ7
ついでにScienceに報告された共有結合性阻害剤のPDBファイル(6Y2F), 前回紹介したKNIMEの実行結果から見つけた非ペプチドアナログリガンド(2GZ7), 変異プロテアーゼの基質ペプチドとの共結晶構造のPDBファイル(2Q6D)をFetchしてきました.
今回のFetchコマンドのように,複数のファイルやオブジェクトを指定したい時はコンマ(,)は要りません.
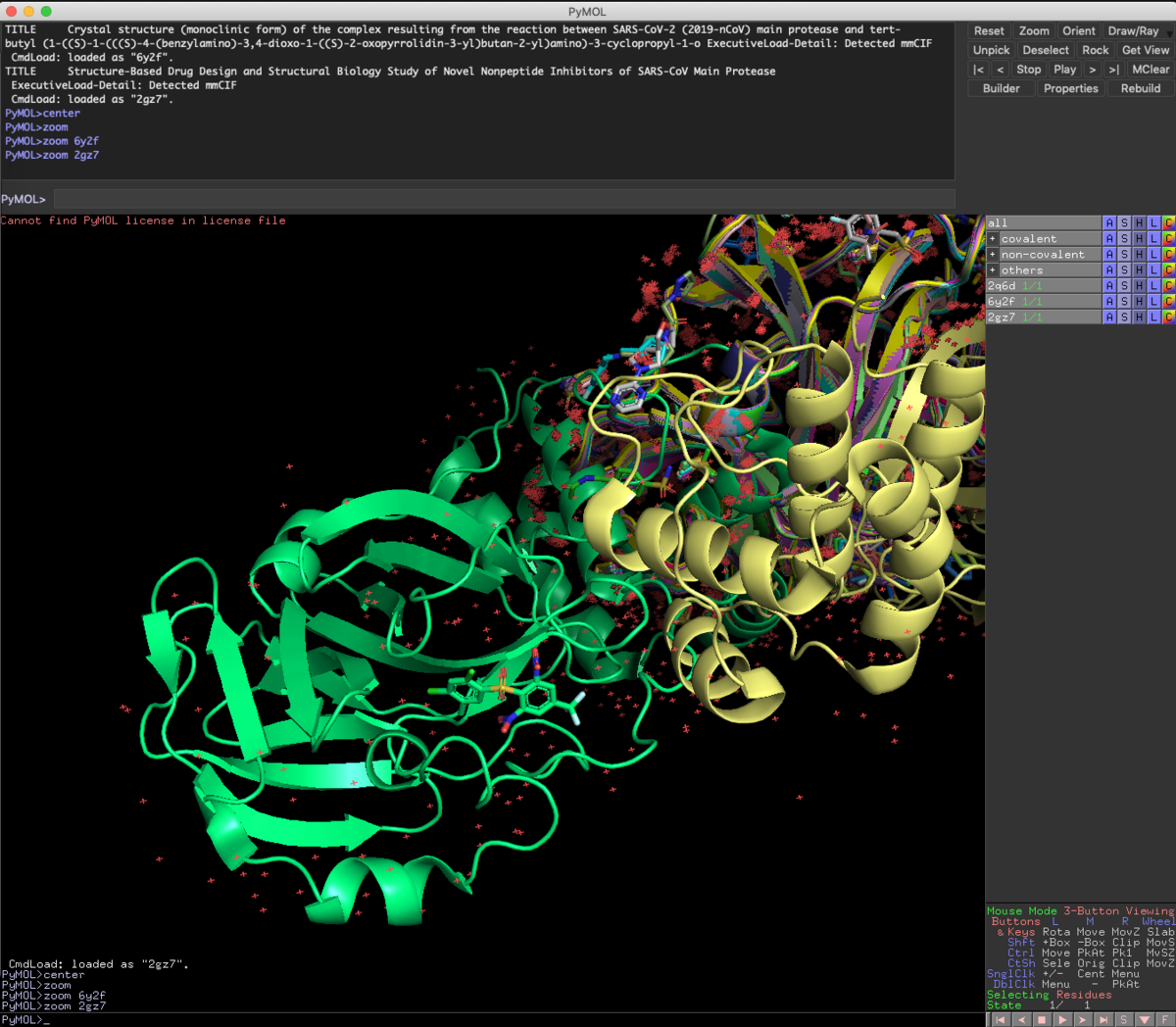
Zoom
オブジェクトを特に指定していないと全体表示されます.
今回Fetchで読み込んだ3つのファイルは,さすがに重ね合わせが必要だとわかりますね.
Alignto 2GZ7
Aligntoコマンドは対象のオブジェクト 2GZ7に残り全てのオブジェクトを重ね合わせます.通常はChain Aが重なると思います.オブジェクト数が多いので今回は結構時間かかります...

グループ化と重ね合わせはここまでなので,Pymolのsession fileとして一度保存しとくことをオススメします.
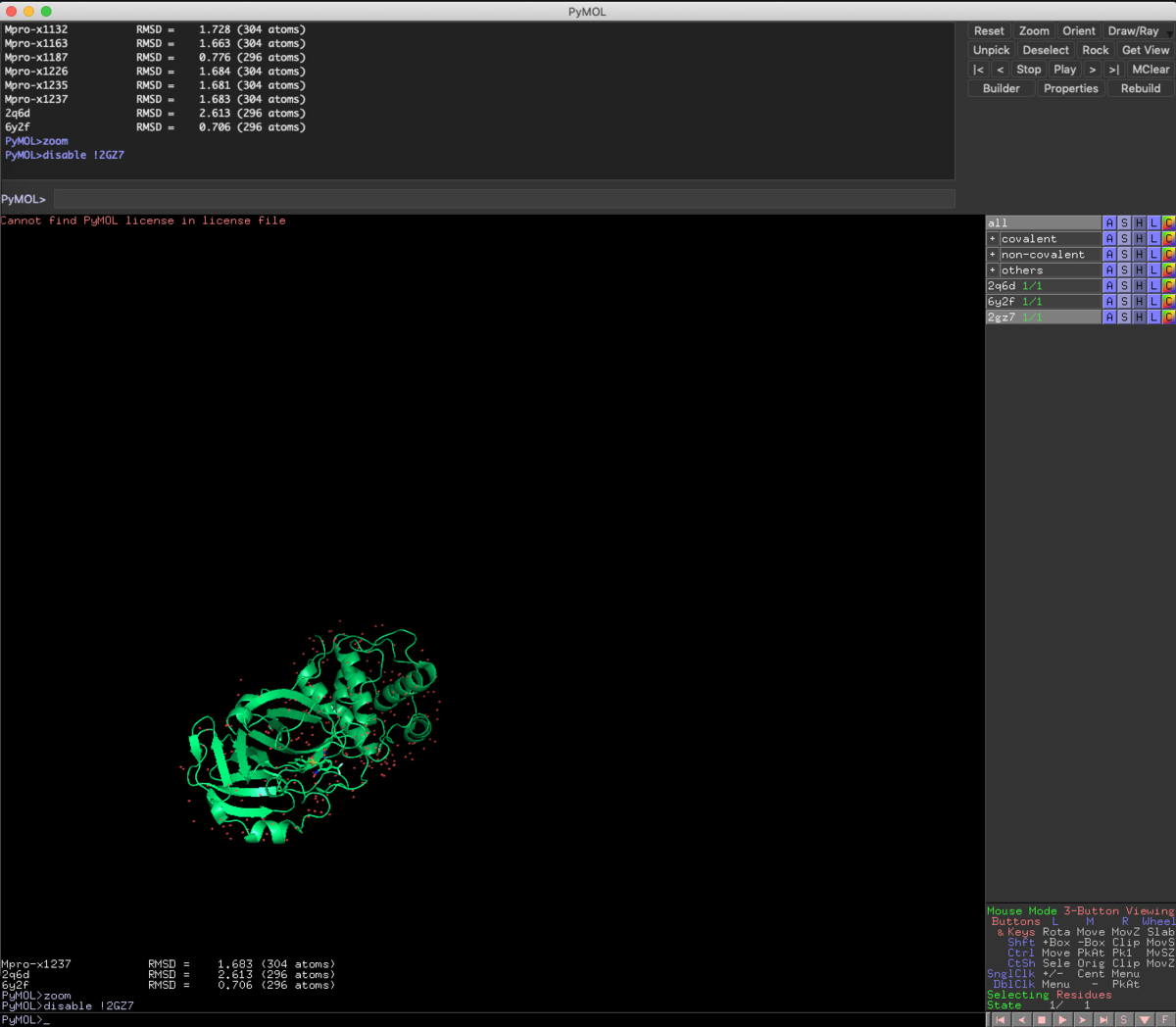
disable !2GZ7
対象の前に!を付与すると,対象が反転します.今回は2GZ7以外を非表示にしました.
zoom byres organic around 5
Ligandはorganicで指定できるので,周辺5Åにzoomします.なんかちゃんとZoomされてないですね...
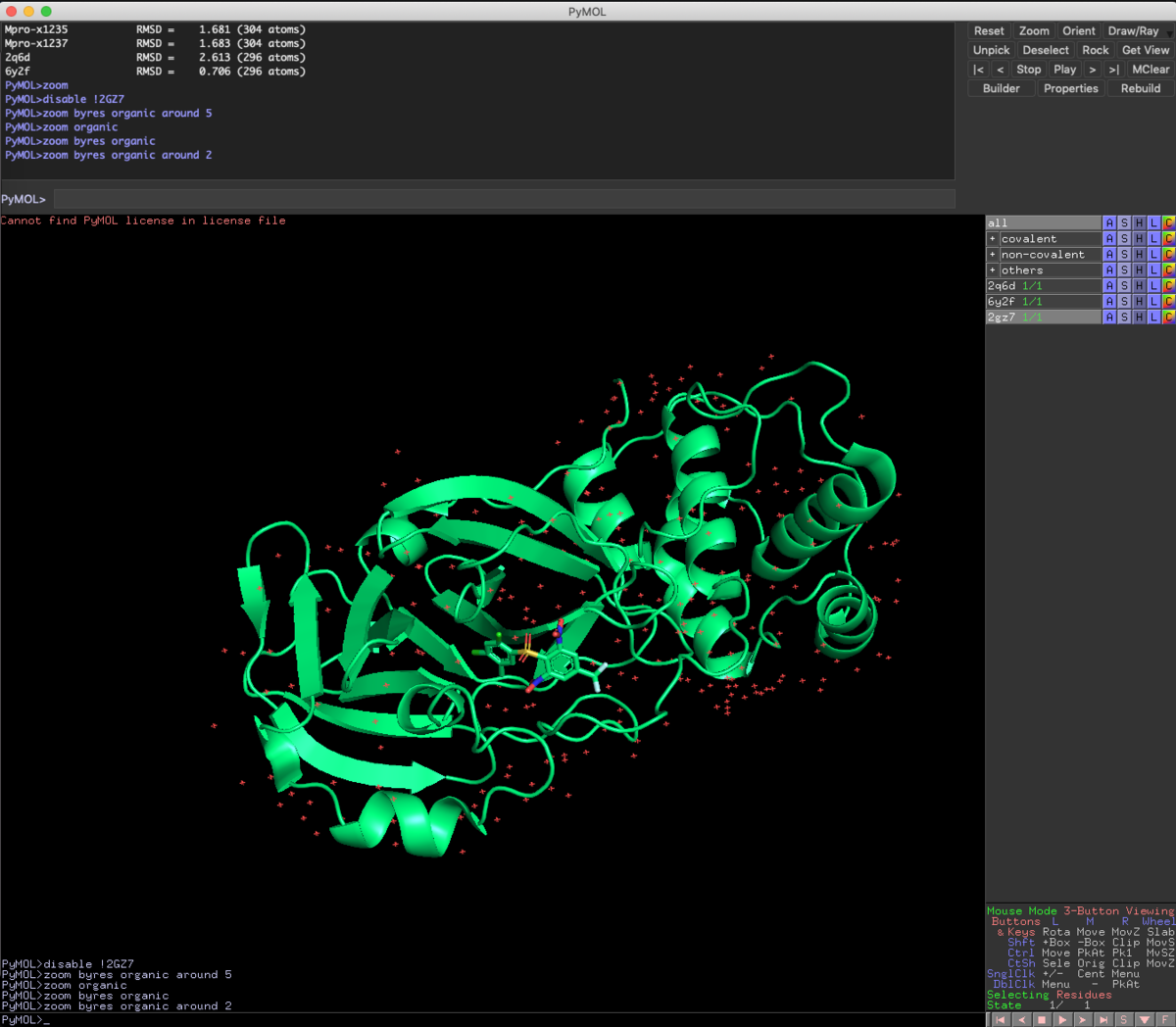
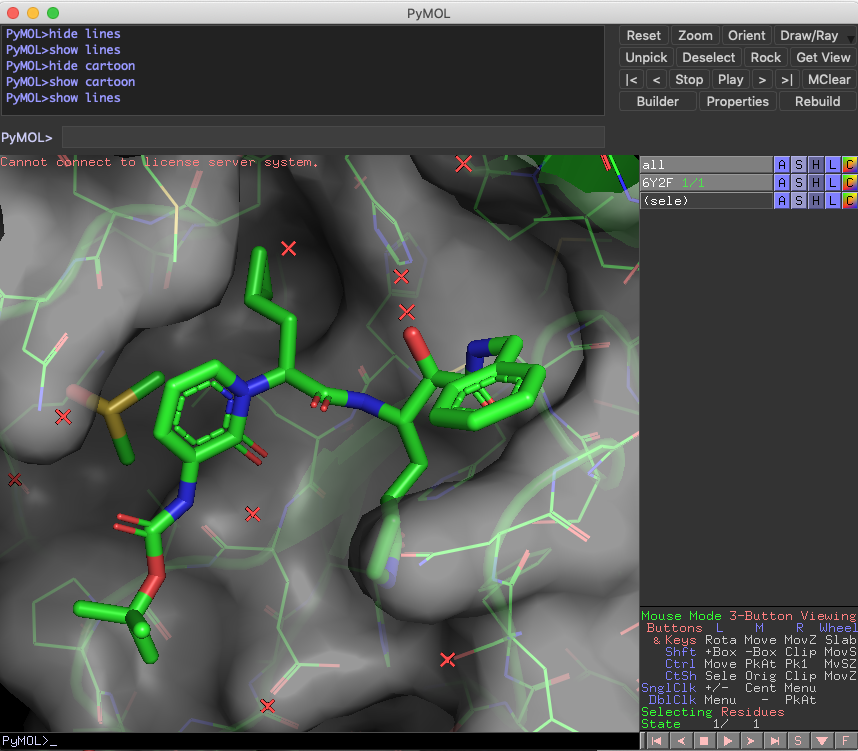
この化合物はRSC PDBに登録されているnon-covalent inhibitorです.
ちょっとLigand周辺の結合様式も見ておきましょう.
preset > ligand site > cartoon
です.
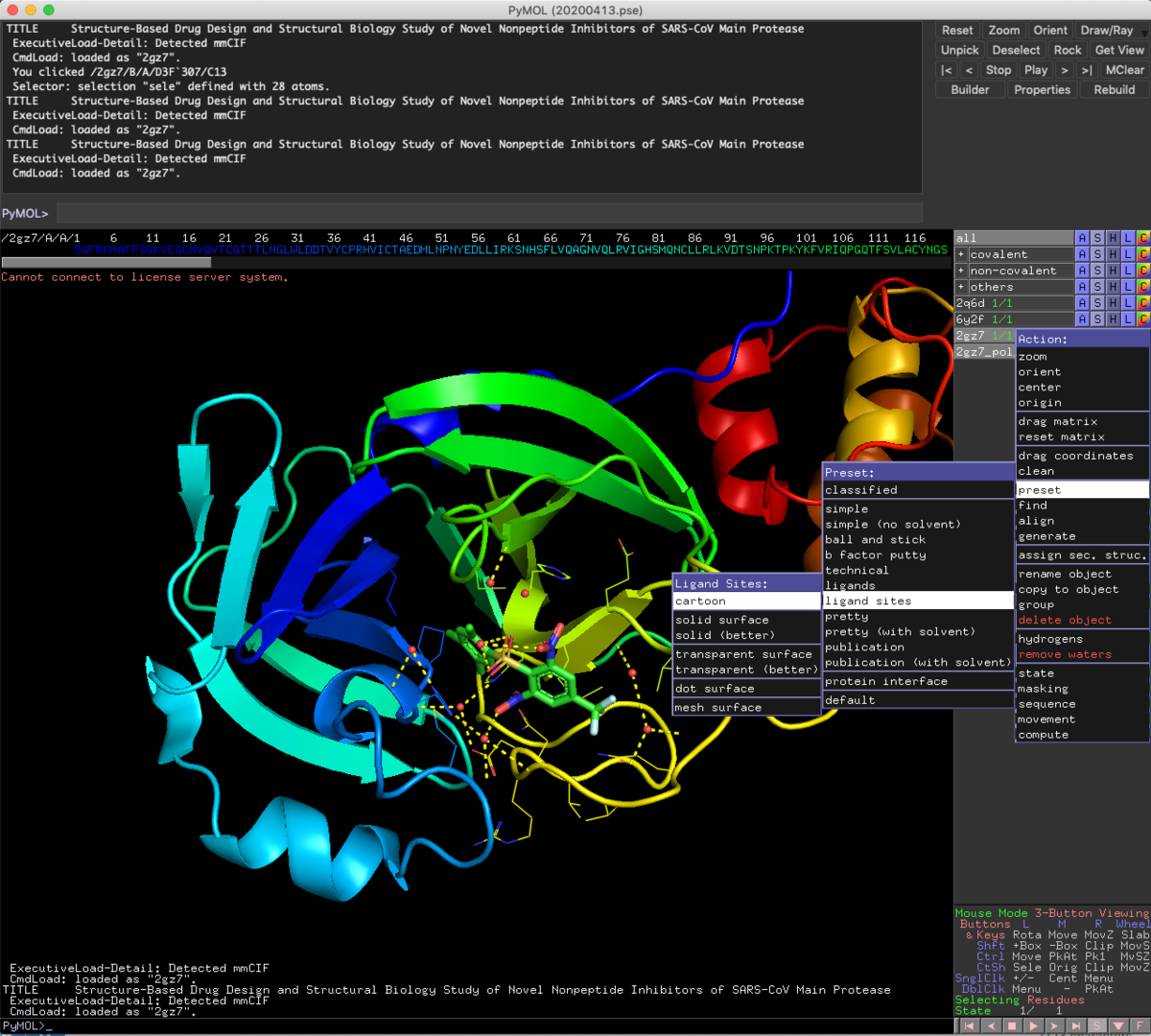
え?
タンパクと相互作用してるの,水を介したニトロ基の一個だけ?
ちょっと他の共結晶とも比較しておきましょう.

disable all
enable 6Y2F
preset > ligand site > cartoon
前回軽く触れましたが,このリガンドは基質ペプチドのミミックなのでバリバリ相互作用してますね.
ここからはちょっと端折って行きます.
disable all
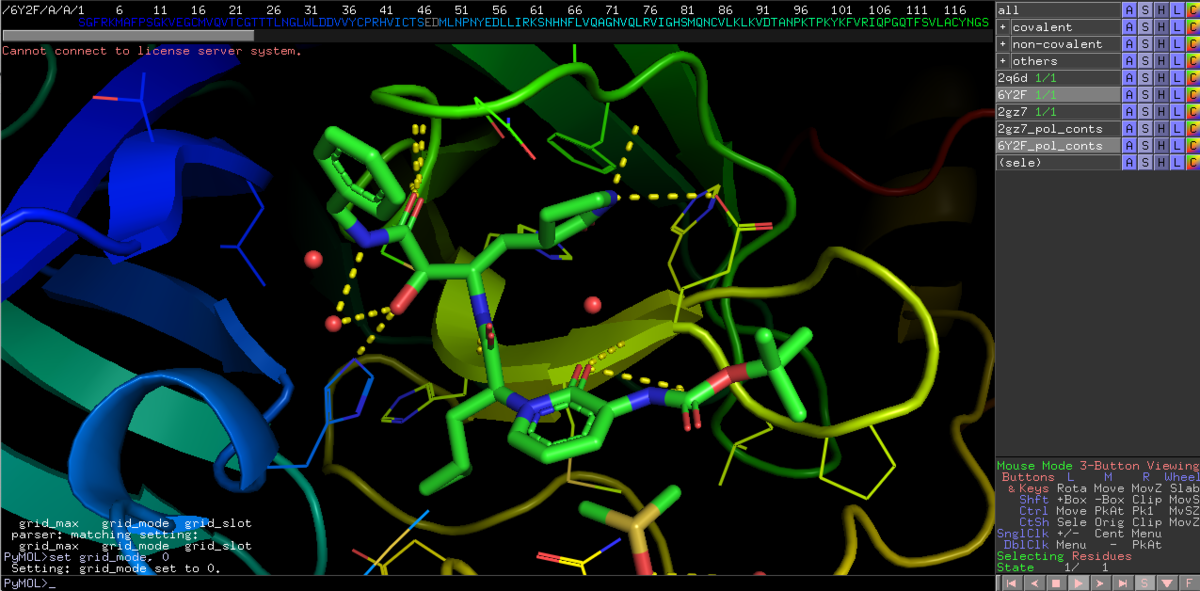
enable 6Y2F 2GZ7
set grid_mode, 1
show surface, byres organic around 5
set surface_color, gray
こんな感じに見えるはずです.
6Y2Fのリガンドである13bの赤枠脂溶性ポケットを三置換のPh基が埋めてますが,雰囲気は結構違います.スルホンとPh基の結合角もなんだか捻れてて気持ち悪いな...
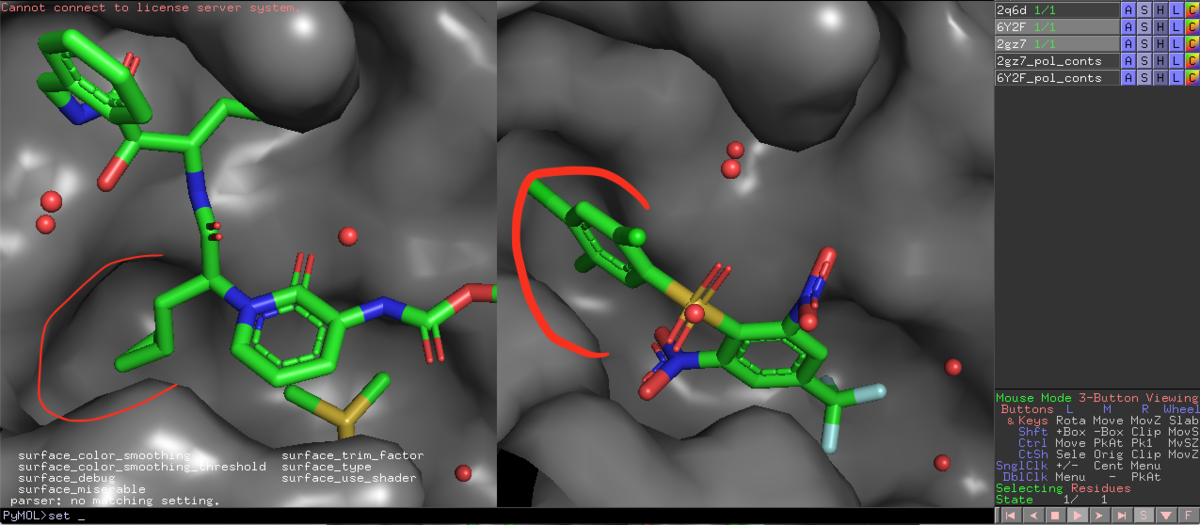
hide surface
脂溶性許容ポケットですが,他に相互作用がなさそうか見ておきましょう.すると...一応ベンゼン環の近くにHis41の側鎖イミダゾールがが平行に配向してますね.左側の6Y2Fと比較しても,フリップしてきてることがわかります.確かこいつは,システインプロテアーゼであるMproの加水分解の活性化を担っていたはずです.
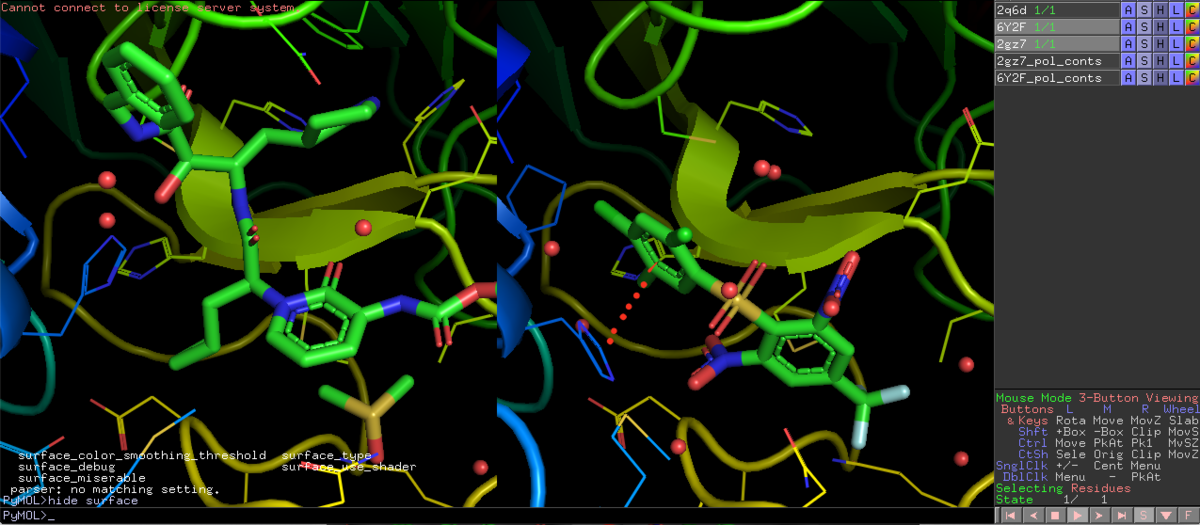
set grid_mode, 0
disable 6Y2F
右下のMouse Modeから3-Button Editingに切り替えて,リガンドと側鎖の距離を見てみましょう.3.8Åなんで,相互作用は有りそうですかね?
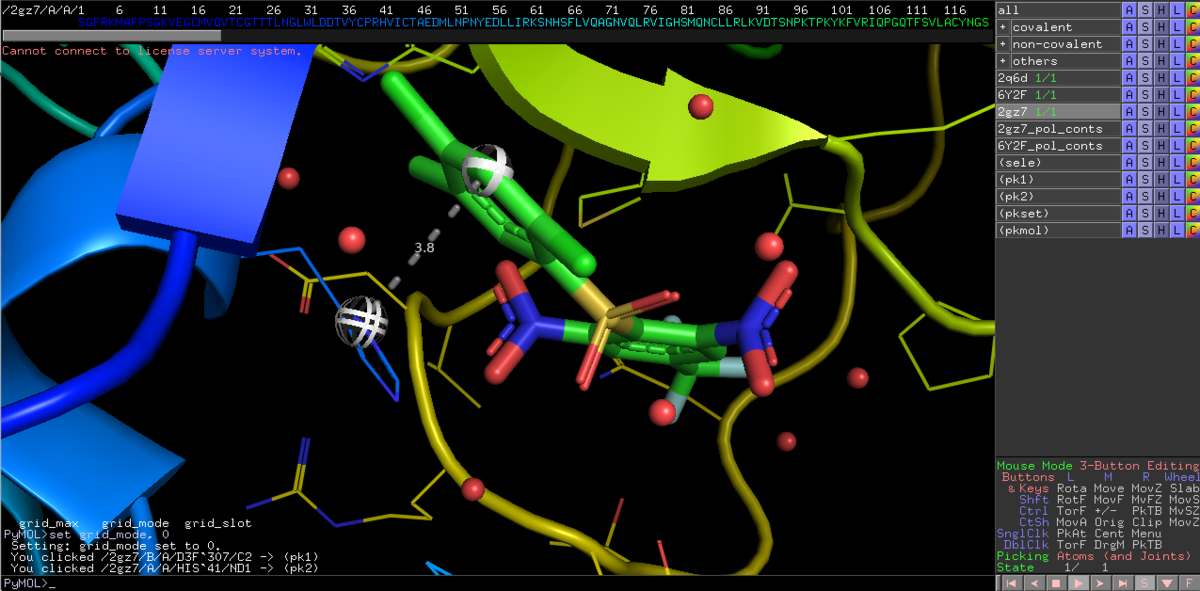
disable 2GZ7
enable 6Y2F
今度は共有結合性ペプチドミミックリガンドと,His41の距離を測ってみましょう.Cys145からの付加反応を受けて,ケトアミドはアルコールとして共結晶が得られています.この立ち上がった水酸基とイミダゾールの距離は2.7Åなので,このHis41が加水分解の触媒として機能しているとみて間違いなさそうです.2GZ7のリガンドは,このHis41を上手く捉えて活性を発現してるってことなんですかね.

ただ,活性値を見ると0.3µMと0.67µMで2GZ7の方が高活性として報告されてるんですよね...ほんまかいな...
共結晶だけでは活性値の推定は中々出来ないので何とも言えませんが,個人的には2GZ7のリガンドの活性は非特異結合か何かが上乗せされてる気がします.
今回のようなプロテアーゼは,基質認識を甘くすることで色んな配列のペプチドに切断出来ることが特徴なんだそうです.つまりここを上手く抑えることが出来れば,virusに必要なタンパクが作られなくなるので効果的と言えますが,一方で色んな基質が結合するなど,選択性の確保も課題になりそうです.上手いこと出来てますね.
enable all
hide cartoon
最後にちょっとだけ,Xchemのフラグメントスクリーニングの結果も見てみましょう.リガンドが集中している黄色で囲った部分がMproが基質を加水分解する部分な訳ですが,othersでgroup化したリガンドが別のところにくっついてます.一つ一つを確認しないと分かりませんが,阻害活性の情報もないですし,あってもアロステリックなので変異が入ったらおしまいです.non-covalentから上手く分子をデザインして,他のシステイプロテアーゼやグルタチオンと選択性を出せれば勝ちってところでしょうか.

今回はこんな感じです.さぁどんな分子をデザインしましょうか?