Dear Pymol Beginners 3 x KNIME 1.3 -リガンドのSDFファイルを書き出してみたり,リガンドの結合様式を比較してみたり-
Grüezi,
Appleで春の学生向けキャンペーンが明日までですね.
新しく出たMacBook AirとiPad Proを順番にコンボで買える日を夢見てましたが,予算の都合上,後者だけになりました笑.オマケのギフトカードで念願のApplePencil2も買うことにします!やっほい!
ひとまず8年選手のMacBook Air2012を使い続けるために,Mojaveをクリーンインストールして...画面が映らない騒動が勃発し...結局あんまり動作は軽くならずに限界を感じつつ...とかやって,ひとまずKNIMEとanaconda, Pymolをインストールし直しました.日本に帰ってからもKNIME遊びは続けられるかなぁ...
と言うわけで今回のWorkFlowでは
- RSC PDBのサイトからPDBとレポートの取得(前回までのマイナーチェンジ)
- レポートからリガンドをSDFとして出力
することが出来ます.
最後に,このWorkFlowの利用例をPymolを交えて紹介してます.
*前回,前々回の重複内容は省きます.
- WorkFlowの全体像
- PDB Connector
- PDB Saver
- Excel Sheet Appender
- RDKit To Molecule
- String Manipulation
- Group Loop Start
- SDF writer
- Variable Loop End
- String Manipulation
- String To URl
- Copy/Move Files
- 実行結果
- 利用例の紹介
- Pymolで観察
- set grid_mode, 1
- show surface, byres organic around 5
- (set surface_color, gray)
WorkFlowの全体像
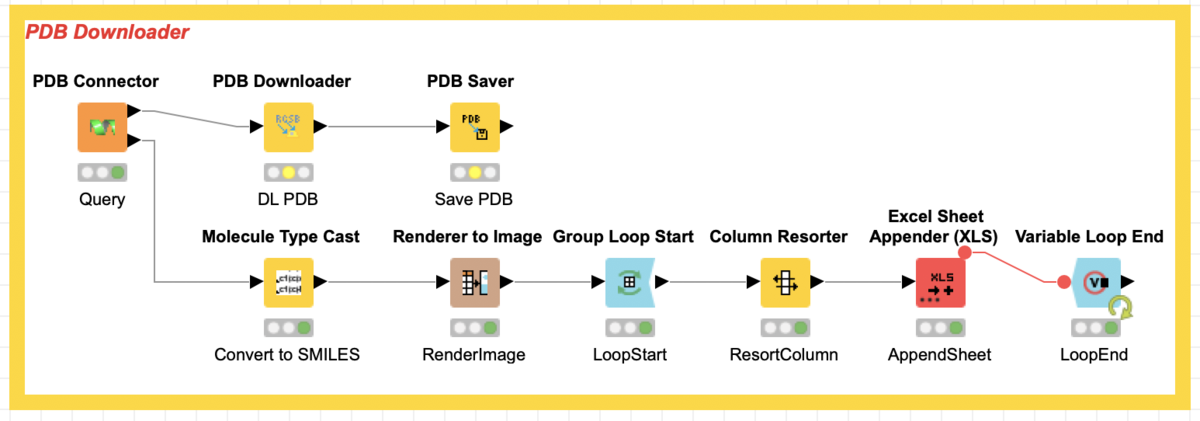
上段と中段は前回とほぼ変わりません.中段先頭に置いていたMolecule Type Castは不要とわかったので削除しました.
下段のWorkFlowでは,PDBのレポートに含まれるSMILESからSDFファイルをPDB IDごとに生成しています.残念ながら今回は利用例を紹介するところまではたどり着けませんでしたが,このWorkFlowを使ってどんなことが出来るかを想像するところを宿題ってことにさせてください.(To be continued...)
PDB Connector
IDs and Keywordsは前回と同じくSARS-CoV-2です.
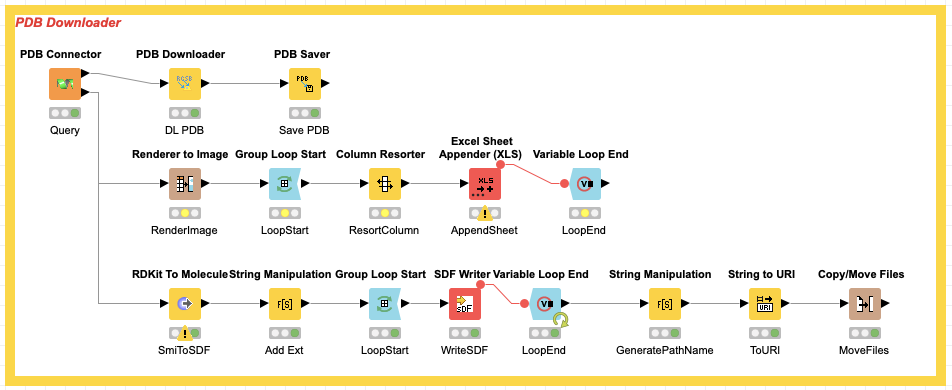
PDB Saver
今回はデスクトップ上に架空ユーザー(xxx)のフォルダを指定しました.
Excel Sheet Appender
今回は上記の架空ディレクトリと同じ場所を指定しています.シート名を変数にして出力するのは前回までと一緒です.

RDKit To Molecule
PDB Connectorから得られるreportのSMILES情報は既にSMILESとして機能してるみたいです.なので,このNodeを使ってSMILESをSDFに変換します.SMILESの方が都合がいいこともあるので,SourceColumnは残してます.Destination FormatはSDFですが,他のファイルも指定できます.

String Manipulation
変換したSDFファイルをリガンドごとに個別に出力したいのですが,そのまま出力するとファイルに拡張子(.sdf)が付いていません.そこで,このNodeで拡張子付きのStringを生成しておきます.ここではPDB IDとしておきます.

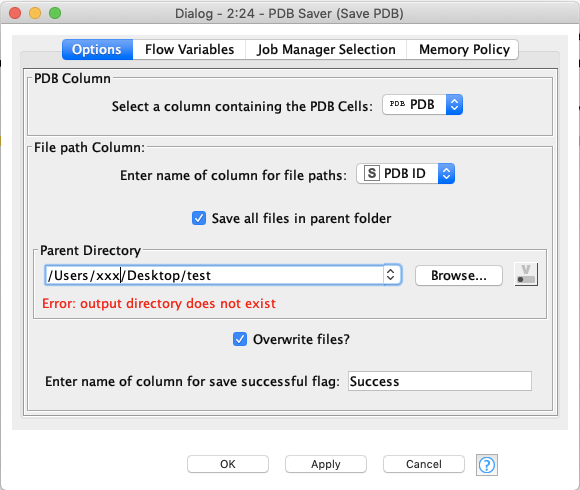
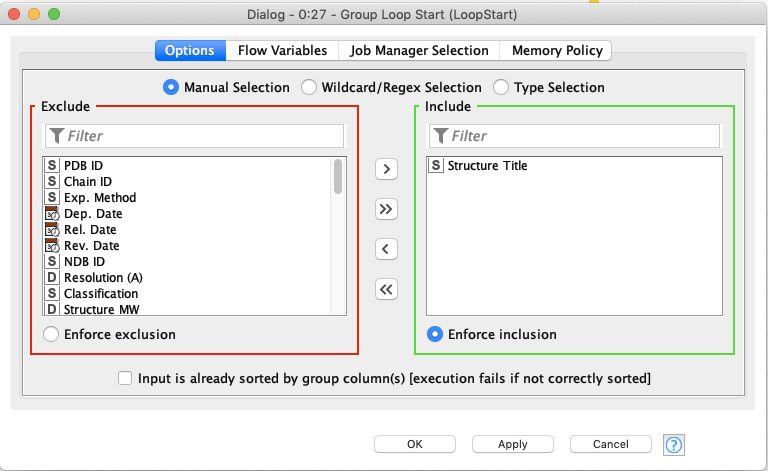
Group Loop Start
上述で生成した拡張子付きのリガンド名ごとにファイルを作りたいので,前項でAppendしたLigandPDBIDというカラムを指定してLoopをスタートします.このカラムについて1行ずつ繰り返し操作を行うってことですね.
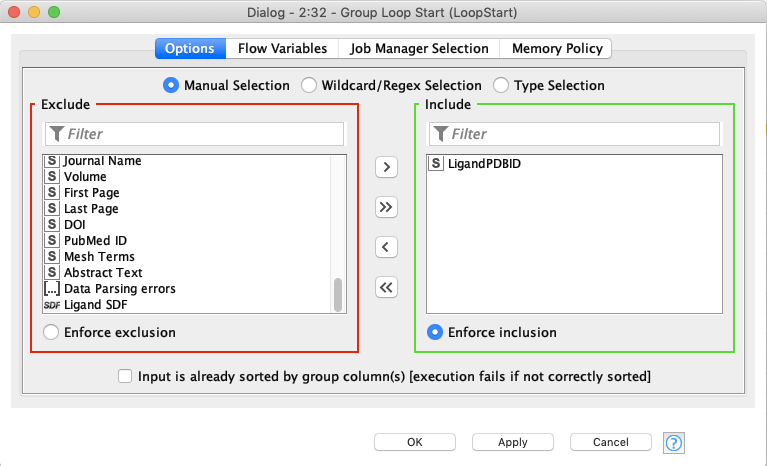
SDF writer
今回は書き出すSDFファイル名が変数になるように設定します.ファイル名の右側にあるvをクリックして,Use VariableからLoopするカラムを指定します.Use Column as molecule titleにチェックしておくと,SDFごとに分子に名前がついて後々便利だったりします.
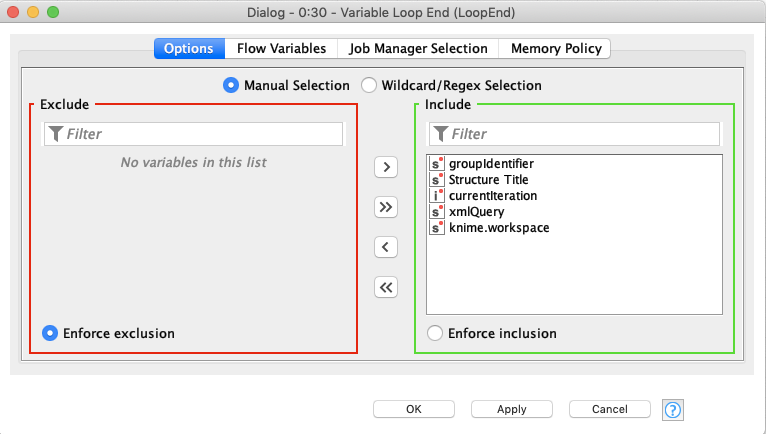
Variable Loop End
特に設定しません.
String Manipulation
個人的にはここが今回一番トリッキーでした.
前々項のSDF Writerはお気付きの通り,出力先を指定していません.すると,私の場合,デフォルトでは/Applications/KNIME 4.1.0.app/Contents/MacOS と言うディレクトリに保存されていました.なので,このSDFファイルをPDBファイルと同じディレクトリに移動させたいと思います.単純に便利だからです.
このNodeでファイルが出力された場所を指定します.ディレクトリ末尾に/を追記した/Applications/KNIME 4.1.0.app/Contents/MacOS/ にLigandPDBIDと言うファイル名のカラムを追記していきます.

String To URl
前項で生成したStringをURlに変換します.

Copy/Move Files
最後にSDFファイルの出力先からPDFのレポートと同じディレクトにファイルを移動させます.
実行結果
以上を実行するとこんな感じになります.

べらぼうな数が吐き出されるので途中は割愛してますが,リガンドのSDF,レポート,PDBが同じフォルダに集まりました.
前回軽く紹介したPDB ID:6Y2Fなんかは,リガンドがDMSOを含めて2つとされています.

この場合,SDFファイルには2つの分子がリガンドとして出力されています.

利用例の紹介
ついでにさっきのレポートの中から,同じターゲットの別のリガンドを探してみたいと思います.main proteaseで検索をかけると,例えばペプチドアナログ以外にこんなリガンドが見つかりました.今回は出来ませんでしたが,IC50値などの活性値が報告されていると,絞り込みの方法はグッと広がると思います.

*追記
ペプチドアナログ以外を見つけたいとか書いておきながら、思いっきりペプチドアナログを見つけてますね…。深夜の疲労のせいということでご愛敬…。
Pymolで観察
このPDB ID:6W63と,6Y2FをPymolで読み込んでみましょう.

今回はそれぞれをfetchで呼び出した後,preset > ligand site > cartoonで表示しています.
次に6W63のオブジェクトから,align > to molecule > 6Y2Fで重ね合わせます.zoom organicでリガンドに照準を合わせるとこんな感じです.

6W63のリガンドは思いっきりペプチドアナログと同じBinding Siteに来てますね!次のコマンドラインでGrid表示してみましょう.
set grid_mode, 1
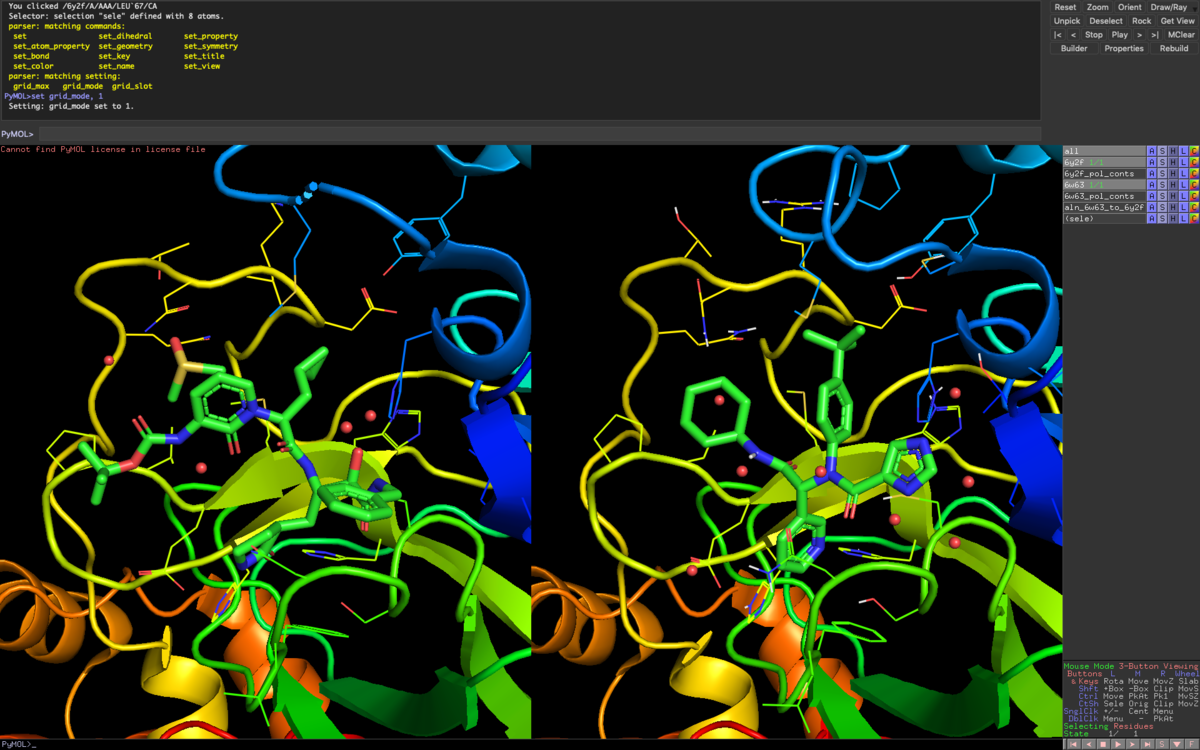
polar contactは一旦非表示にしました.ついでに6W63のnon polar hydrogenも非表示にしてみやすくしてます.次にポケットの比較をしてみます.
show surface, byres organic around 5
(set surface_color, gray)
リガンド周辺部だけSurfaceを表示しました.特に設定をしてない場合は見辛い配色だと個人的に思うので,色を灰色に変えました.

シクロプロピルメチル基が埋めていた(確か)S2ポケットと呼ばれる脂溶性空間にt-Bu基がうまくはまってますね.(下図)

S1ポケットと呼ばれるグルタミンサロゲートが埋めていた部分は3ーピリジルで置換されています.HBDいらないんですね.

件の共有結合部分を観察してみましょう.
6Y2FではC145から思いっきり共有結合しているので,その距離は1.8Åです.

一方,6W63ではC145と共有結合こそ形成していませんが,おそらく該当するであろうアミドとの距離は3.4Åです.プロテアーゼでは通常,コアの反対側にあるヒスチジンからHBDのアシストを受けて加水分解の活性化を受けたりするんですが,今回はそういった残基も特には見当たりませんでした.原子間距離自体は近いので,リガンドの結合にこのアミドは何かしら関与してそうな気はするんですけどね.

他にも細かい相互作用解析はあるんですが,今回はこの辺で切り上げようと思います.
出力したSDFファイルの使い道もWorkFlowが出来たら紹介したいと思います.
Dear Pymol Beginners x KNIME 1.2 -PDBのReportファイルをExcelのシートごとに分けてみたり-
Grüezi,
コロナ騒動がおさまりませんが,私はグラント応募の準備がひと段落したのでKNIME遊びを更新しました.
今回のUpdateはこんな感じです.
- PDB Downloader WorkFlow
- PDB Connector
- Group Loop Start
- Column Resorter
- Excel Sheet Appender (XLS)
- Variable Loop End
前回までにPDB Connetctorで取ってきたPDBと,その付随情報をエクセルで取得するWorkFlowを紹介しました.
今回はLoopを使って,このExcelファイルをもう少し体裁よくします.
Loopを回した結果はこんな感じです.
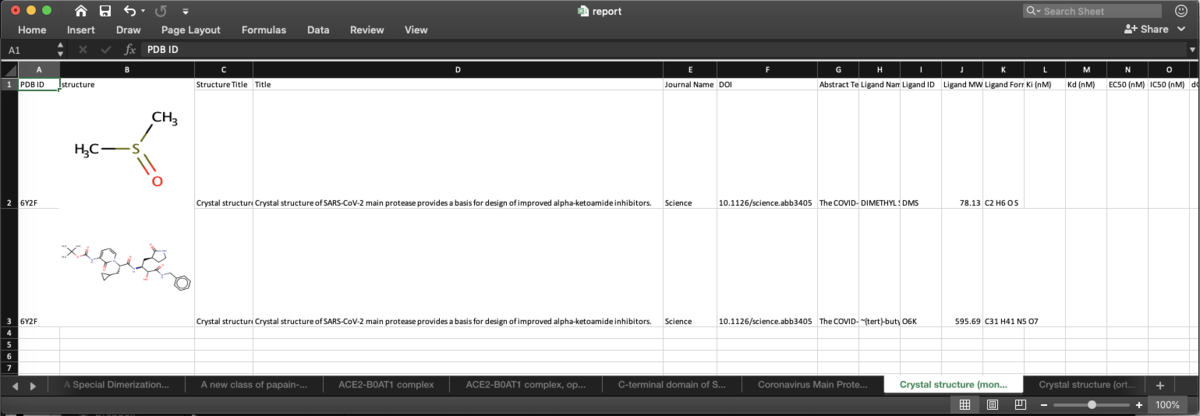
私はケミストなんでリガンドの構造情報が先頭にある方が親近感が湧きます.
阻害剤であればIC50の情報なんかもあると参考になります.
あとはアブストラクトなんかも取得しておくといいかもしれません.
この上から2番目の構造は,Scienceに報告された共有結合性阻害剤で,c&enなんかでも紹介されていました.
ターゲットやキーワードからPDBを検索した時に,リガンドの構造や阻害活性の情報,アブストラクトなんかを俯瞰できると便利かなと思います.今回はやたら検索結果が出てきたので,もう少しうまく絞り込んだ方が良さげですが...
PDB Downloader WorkFlow
まずはWorkFlowをまんま載せるとこんな感じです.
PDB Connector
内容は前回紹介した通りなんで割愛しますが,今回は少し広めのSARS-CoVで検索をかけてみたいと思います.

Group Loop Start
一つ前のRenderer to ImageというNodeでは下のような結果が得られます.一つの文献の中で複数のリガンドが報告されることはよくあることですが,このままExcelに吐き出してしまうと情報量が多くて,個人的には見る気が湧きません.
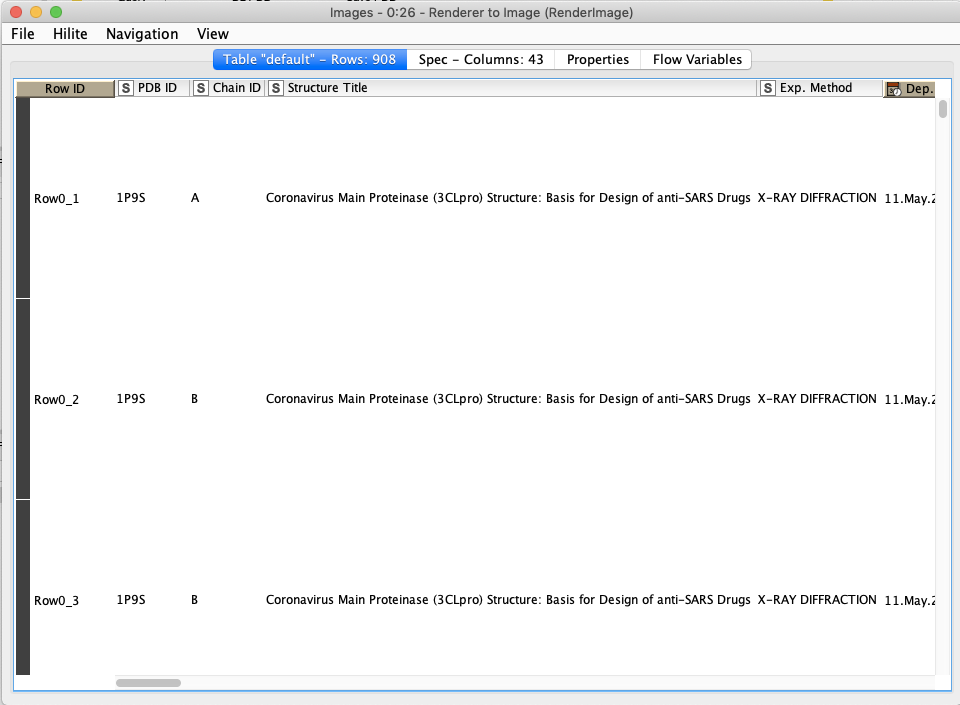
そこで,Group Loop Start Nodeを使って文献ごとにグループ化して,その結果をシートで分けてしまおう,というお話です.
設定からはGroup化したい項目を選択します.複数選択するとより細かくGroupingされます.今回はシートが増えてかえって見づらくなるので,シンプルにStructure Titleだけにしました.
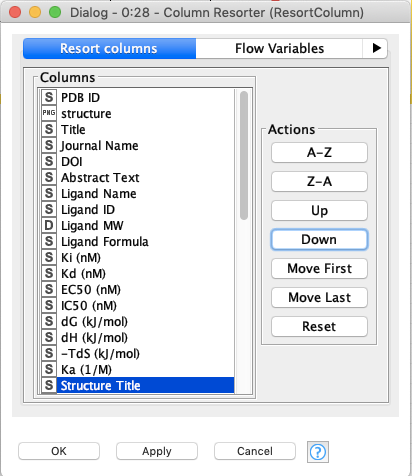
Column Resorter
カラムの並びを好みの順番にします.この辺は好みにあわせて変えるといいと思います.
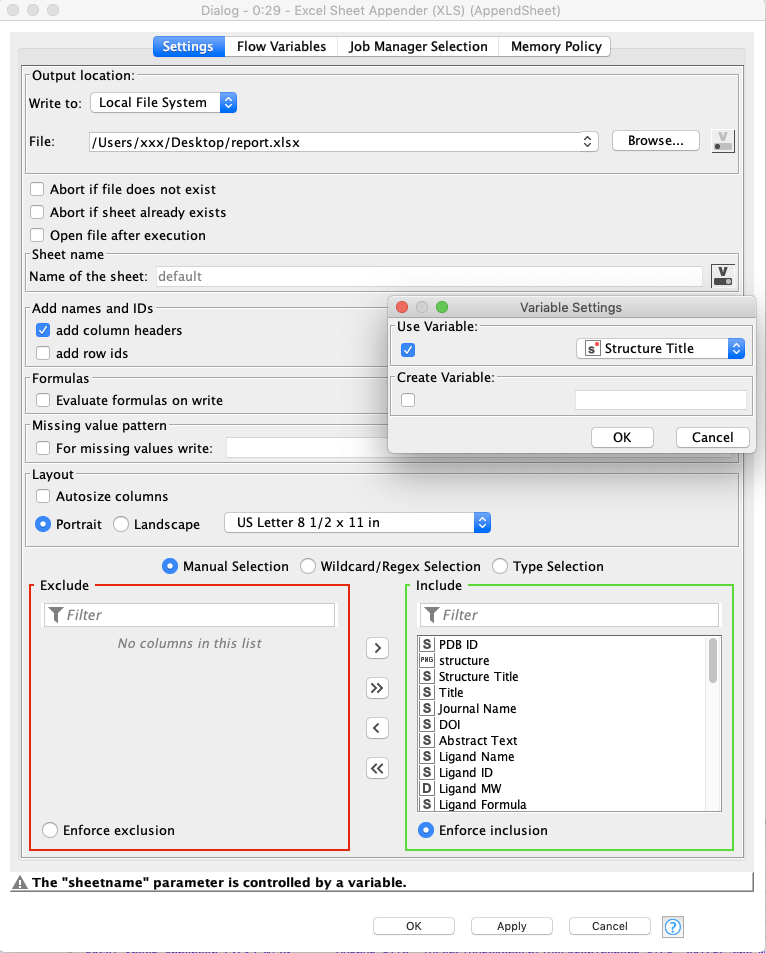
Excel Sheet Appender (XLS)
このNodeでGroupingの結果をエクセルの新規シートとしてAppendします.
Sheet nameの右端にあるvのボタンを押しすことで,前項のGroup Loop Startの結果を変数として扱うことが出来ます.Use Variableにチェックを入れて,どの項目でGroup化するかを選択します.今回はStructure Titleです.
Add names and IDsのadd column headersにもチェックを入れておくと,実行結果のExcelが見やすくなります.
Variable Loop End
最後にここがLoopの終わりだよと宣言するNodeです.特に何も設定してません.
KNIMEを使い始めた頃,「Loop系のnodeは繰り返し処理に使う」というのは知ってはいたんですが,イマイチ使いこなせていませんでした.こういう簡単な処理でLoopを使ってみるのが理解を早めてくれますね.
最後になりますが,今回のWorkFlowはこちらの記事を参考に作成しました.
先人にはいつも大変にお世話になってます.この場を借りて感謝申し上げます.
SDFファイルやPDBファイルも文献ごとにフォルダ分け出来ないかも勉強中です.
KNIMEのVersionあげたら動かなくなったNodeもあって四苦八苦してますが,実験が出来ないので,いい機会だから色々試してみようと思います.
それではWiedersehen.
Dear Pymol Beginner x KNIME -KNIMEを使ってPDBファイルをまとめてDLしてみたり-
Grüezi,
世間ではコロナウィルスが大流行ですが,皆さんいかがお過ごしでしょうか.
さて,この数週間でCOVID-19関連の構造情報が複数公開され,各PDBサイトにも特集が組まれています.
2020/3/14現在でRCSB PDBのサイトには19個の構造情報が公開されています.19個の結晶構造を一変にダウンロードするのは面倒なので,一発で済ます方法を紹介します.*但し,KNIMEユーザーに限る.
*2020/03/18 Molecule Type Cast Nodeは不要だったので削除しました.
本日のコンテンツはこちら.
- KNIMEのWorkFlow
- PDB Connector
- PDB Downloader
- PDB Saver
- Renderer to Image
- Column Resorter
- Excel Writer
KNIMEのWorkFlow
まんま載せるとこんな感じです.

Community Nodes-Vernailsをインストールしておくと,RCSB PDB Toolsというノードが見つかります.それを並べただけのシンプルな構成です.
レポートをLoopで見やすく出来ないかは勉強中です...
PDB Connector
このノードを使うと,RSCB PDBのAdvanced Search(http://www.rcsb.org/pdb/search/advSearch.do)と同じ検索をかけることが出来ます.設定を少し見てみましょう.
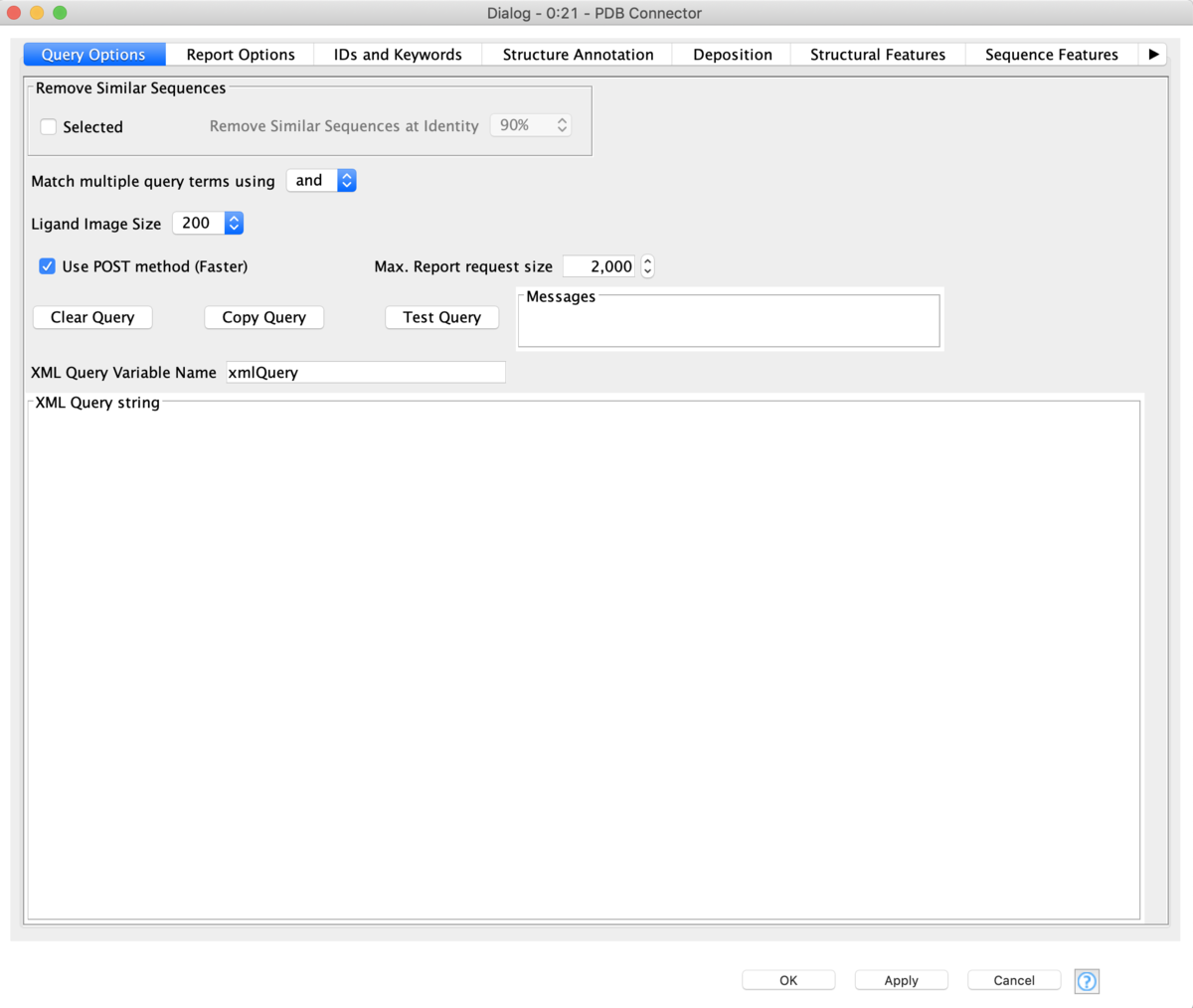
Query Optionsは一番最初に私がハマったポイントです.
Remove Similar SequencesのSelectedからチェックを外してください.そうしないと,同じ報告内の異なるリガンドの共結晶構造ははじかれてしまいます.

Report OptionsでPDB IDからStructure Titleや掲載日など,簡単な情報を取ってきてくれます.結果はノードの下から出力されます.
Select Reportから,上の方にあるCustomizable Tableを選択し,必要な項目をチェックしていきます.とりあえずざっくりとStructure Summary,Ligands,Binding Affinity,Primary Citation辺りをチェックしておきます.後から変更できますし,Row Filterとかでも絞り込めます.基本の設定はここまでです.

IDs and Keywordsから検索したいQueryを作成します.Text Searchをアクティブにするのがシンプルで使いやすいかと思います.今回は話題のcovid-19と入力しておきます.
他にもFilterをかけたい時は他のタブから条件を絞り込んでいけばOKです.解像度や測定方法など,結構細かいことを絞り込めます.
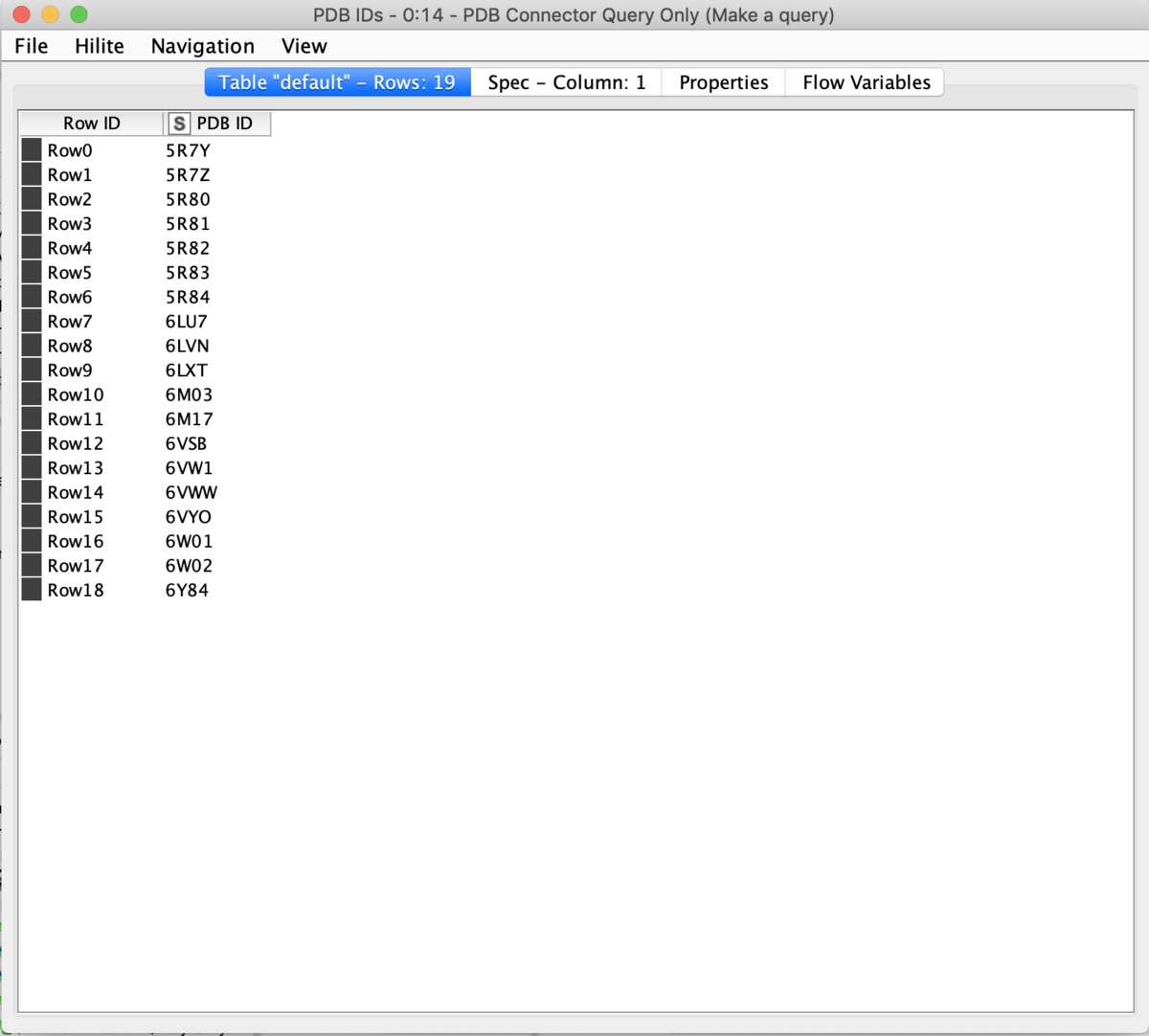
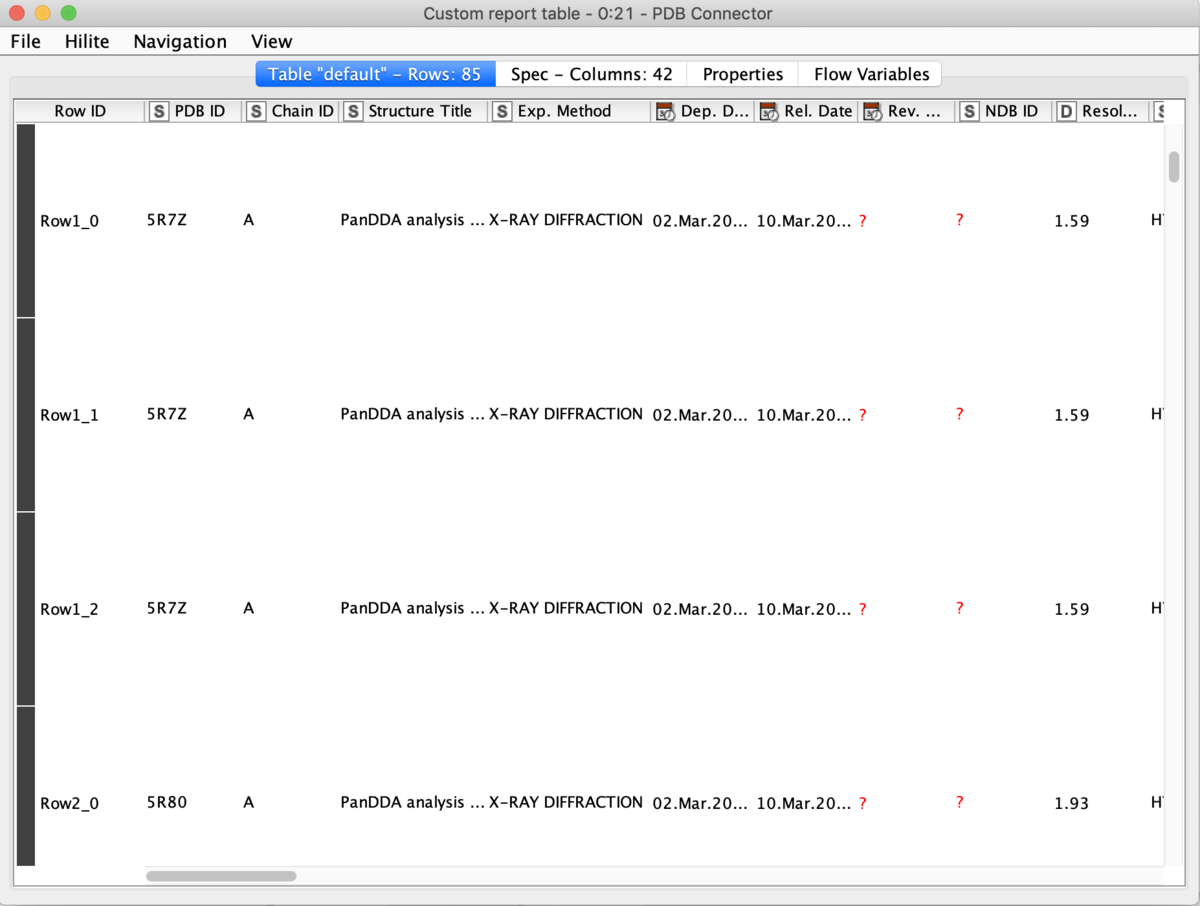
設定を完了して実行すると下記のようにPDB IDとReportが返ってきます.
PDB Downloader
その名の通りでPDBをダウンロードしてきます.この時点で保存はされてません(!)
特に設定不要ですが,ダウンロードしたい項目を増やしたり変更したい時にはいじります.
PDB Saver
こちらもその名の通りで,DownloaderでダウンロードしたPDBをローカルに保存します.保存先のディレクトリに,以下のレポートも保存するといいと思います.
Renderer to Image
SMILESの構造情報は,そのままではExcelで単なる文字列としてしか表示できないので,このNodeでさらにPNGファイルに変換します.

SmilesをMarvinを使ってPng形式で200x200の大きさにして,Structureカラムに出力しますよ,という設定です.
Column Resorter
出力されるReportのカラムを並び替えておきます.
構造情報を前の方に並び替えておく方が,Chemist的にはなんとなくフレンドリーです.
Excel Writer
PDBと同じディレクトリを指定しておくと,色々と作業がしやすくなると思います.
以上,ここまでを一旦流してみると,2020/03/15現在登録されている19個の結晶構造と,そのレポートを一度に取ってこれました.レポートをもう少し見やすくまとめるのにLoopを使えないかなぁ...
Trelloで語学学習のススメ
Grüezi,
スイス🇨🇭に来て7ヶ月.一向に英語力が向上した気がしない,どうも或る化みす途です.
以前にTrelloというタスク管理ツールを紹介しましたが,最近リリースされた機能がどうにも便利なので紹介したいと思います.
過去記事はこちら.
本記事はこんな人にオススメです.
-Trelloって何?使ってみたい.
-語学(に限らないですが)学習に便利な単語帳ツールを探してる.
本記事ではこんなことをします.
では行ってみましょう.
まず少しおさらいですが,TrelloとはBoard, List, Cardから構成されるタスク管理ツールです.
Boardの中にそれぞれListを作って,その中にCard形式でタスクを足します.そのカードを自由に動かして進捗管理したりできます.
今回は普段使いのBoardにLanguageというリストを作って,色んなソースからカードを足していきます.

まずはブラウザからカードを作る
ChromeであればTrelloのExtensionが公開されています.
YouTubeでもよく見ているHapa英会話というサイトのコラムからフレーズを頂戴しましょう.こんな感じになります.

こういうフレーズって会話の最中にはまだまだ自然に出せません...
タイトルを日本語の例文に変えておくと,カードを見る→英文考える→コラムで答え合わせ,の流れができます.
iOSからカードを作る
Trelloのアプリを予めインストールしておく必要があります。ブラウザであれば共有ボタンからさくっとカードを作れます。
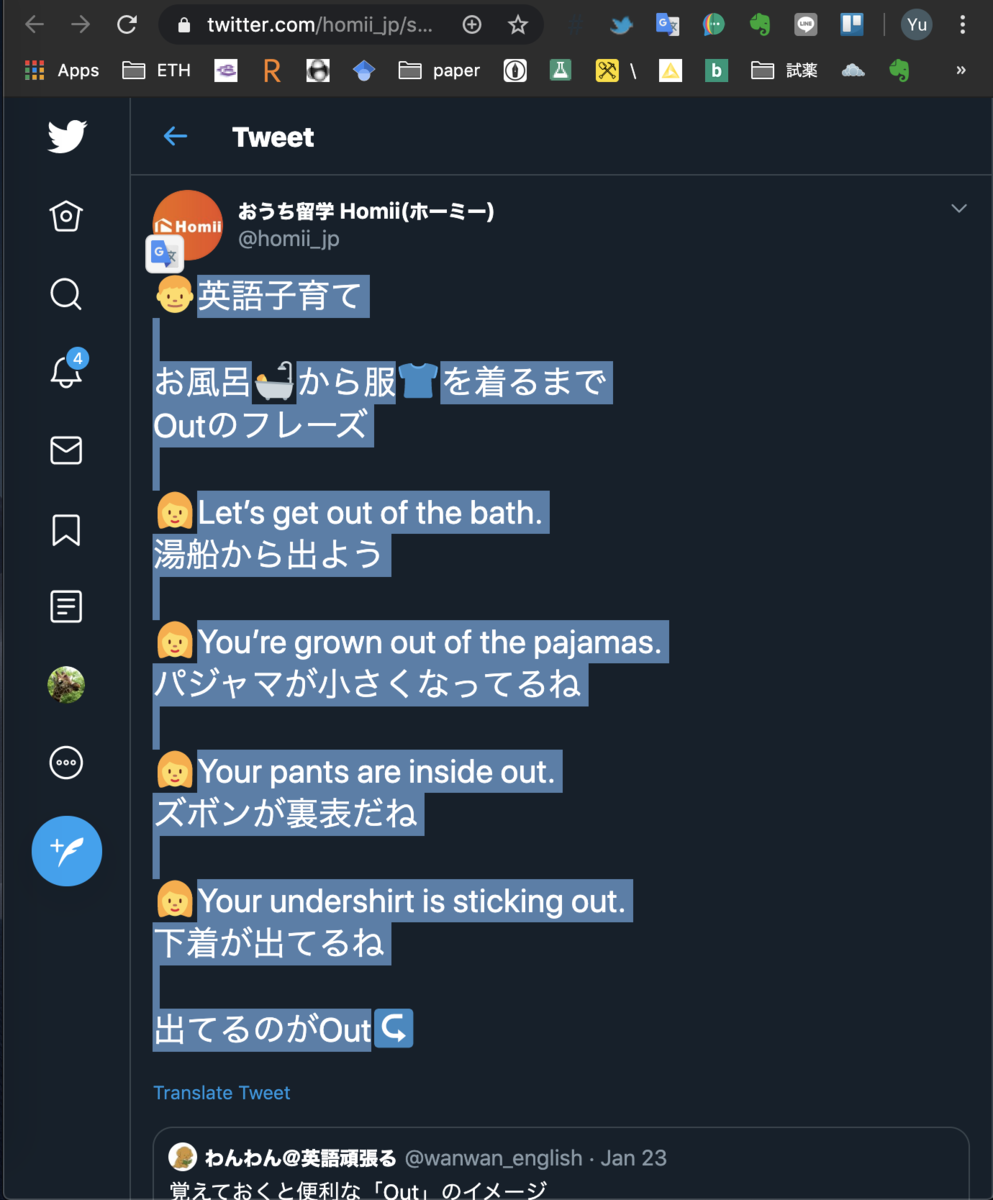
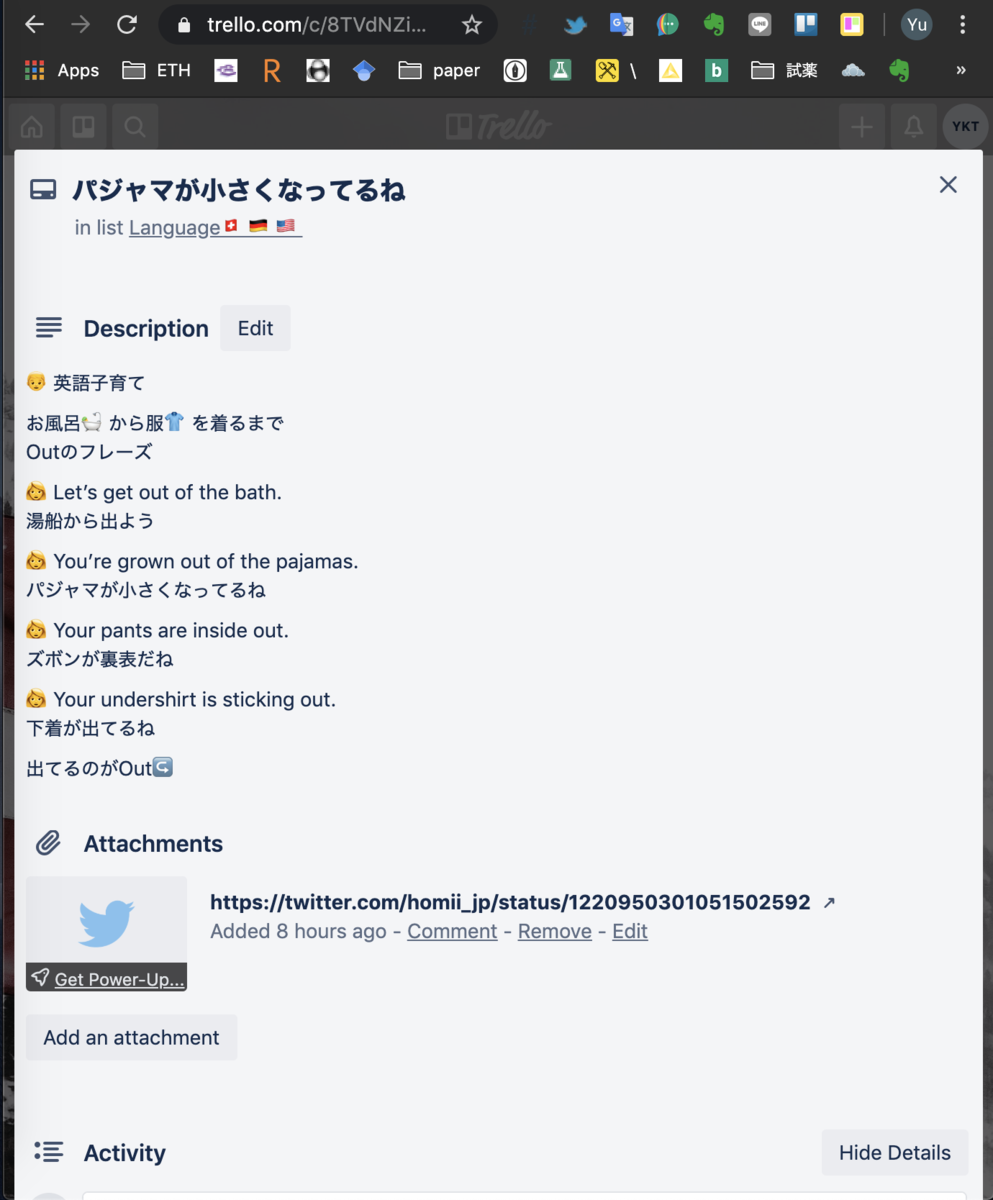
僕は Twitterでフレーズを見つけてカードを作ったりします。例えば@KoalaEnglish180 さんのこちらの tweet画像を長押ししてもカードを作れます。

髪を切ってもらった、みたいな表現も未だに咄嗟に出せません…ということでカードにします。
このやり方だと少し注意が必要です。画像付きカードになるのでスペースを使ってしまいます。
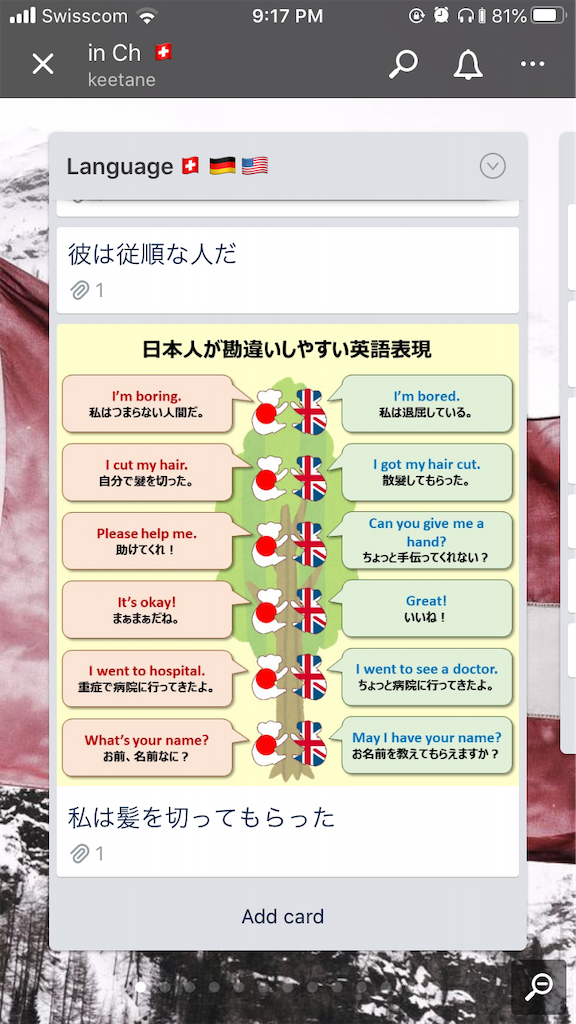
そこで次の操作でイメージを畳みます。
カードを選択→image attachment→右上の…から remove cover imageします。すると、文章だけのカードになります。


これで スッキリとした単語帳になりました.英文を確認したい時はカードを開くだけです.
カードをシャッフルする
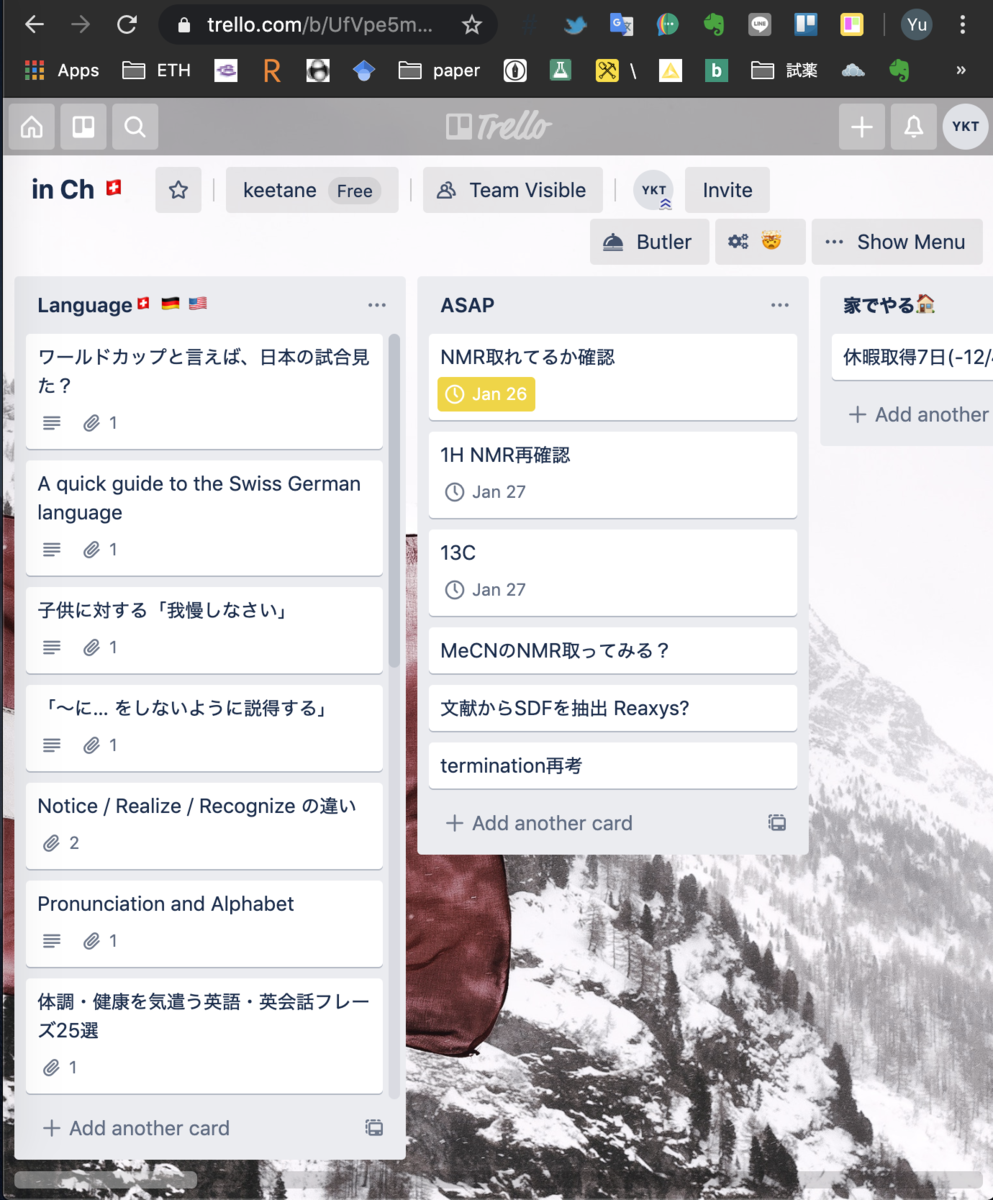
単語をどんどん追加しても,追加した順番でしかカードが出ないといつまでも憶えられませんよね.Evernoteのようなツールだとこれが片手落ちです.Everwordというアプリの連携でシャッフルは解決できるのですが,1)Evernoteの制限でデバイスは2台まで,2)アプリでしかシャッフル出来ない,3)何よりアプリをあんまり開かない,という言い訳で,ノートを使ってもあんまり学習ツールとして機能してませんでした.そこでTrelloに追加された新しい機能"Butler"です.

ButlerはTrello内でAutomated Work Flowを作ることが出来ます.例えば,”カードを動かしたらチームメンバーに通知する”,みたいな具合です.今回はList内のカードをシャッフルするボタンを作ります.
Butlerをクリックした後に表示されるButlerの左側のメニューからBoard Buttonを選択しましょう.

細かい説明は各自読むとして,Creat Buttonしてみましょう.

次にボタンの名前を付けます.ブラウザのブクマもですが,僕は基本アイコンだけで済ませるタイプです.次はAdd actionしていきましょう.

スクロールした一番下に,指定したリストのカードをシャッフルするアクションがあります.リストを指定して" + "を押した後に,Saveしましょう.

こんな感じでButtonが作成出来ました.ついでにチームでシェア出来たりするみたいですね.編集ボタンから他のリストを追加することも出来ます.基本は重要なタスクほど上に来そうなもんですが,シャッフルって何に使うんでしょうね.

こんな感じでボタンが追加されました.
Boardの先頭にリストを置いておくことで,嫌でも目に入る単語帳の完成です.好きなタイミングでボタンを押せば単語をシャッフルしてくれます.
アクションを減らすTips
色んなところからカードを飛ばせて便利なTrelloですが,例えばTwitterからカードを作ると英文の答えが直ぐには出てきません.

リンク先に行って,その他の例文ごとコピペしてしまいましょう.
Descriptionにペーストしておけば,次回以降はもっと簡単に答えを確認できます.ついでにボキャブラリーや用法のパターンも増やしたいところです.
英語の表現力を増やすために,Trelloでこんな運用を始めてみました.今まで使ってたEvernoteよりもフレキシブルなので,結構気に入ってます.
論文読んでる時に気に入った表現なんかもTrelloにListを作っておくと,いざ自分で書く時に便利だと思います.ご参考までに.
それでは.Shonen abig.
Dear Pymol Beginners 2 -リガンドの周辺を観察してみたり-
Grüezi,
前回の記事の手直しも済んでませんが,勢いのある内に次に行こうと思います(済みました23/Jan/2020).
前回はダウンロードしたPDBファイルを見やすく整えるショートカットを紹介したわけですが(←前回言えよ),今回はタンパクやリガンドの周辺環境の可視化に関するショートカットを,実例を踏まえながら紹介したいと思います.
本記事で学びたいことは以下の通りです.
-アミノ酸残基や水分子,リガンドの指定の仕方
-指定した原子団の表示変更の仕方
-指定した原子団から新規オブジェクトを作成する方法
ついでに,
-GFPはなぜ蛍光発色団として機能するのか
です.
Pymol自体はコマンドなんて使えなくてもマウスでポチポチすれば十分便利に使えるので,知らなくても全然大丈夫です.
本日出てくるコマンドは以下の通りです.
- fetch 1EMA
- hide sticks
- show sticks
- hide sticks
- show sticks, organic
- zoom organic
- show spheres, organic
- set sphere_scale, 0.2
- show lines, byres organic around 5
- zoom organic around 10
- set cartoon_transparency, 0.7
- extract sol, solvent
- disable sol
- select 5A, organic around 5
- label 5A and name ca, "%s%s" % (resi, resn)
- create 5A, byres organic around 5
- disable 1EMA
- copy_to 5A, organic
- create 5Aa, byres organic expand 5
- disable all
- enable 5Aa
- zoom organic
- set seq_view, 1
- create CRO, resi 65-67
- create cro, resn CRO
- reinitialize settings
もっと便利に使いこなせるようになりたい時に読みたい
では実践です.
fetch 1EMA
でGFPをダウンロードしましょう.

PymolのDefaultだと思いますが,タンパクはCartoonで,例えばリガンドや結晶化に使った添加剤のような有機物はStickで表示されます.GFPの中心部分に何やらリガンドらしき有機物がありますが,何を隠そう,この有機物と認識されている部分がGFPの蛍光の正体です.詳細はChemStationさんの記事にお願いするとして,GFPの内部という特異な環境でSer65-Tyr66-Gly67の3残基が環化-酸化されることで蛍光を示す発色団を形成します.
hide sticks
発色団が隠れます.
続いて発色団を復元してみましょう.
show sticks
 !!
!!
はい,出来ませんねorz.
このように,Pymolのコマンドでは指定しない限り全オブジェクトに一括で指令してしまいます.
hide sticks
show sticks, organic
zoom organic
発色団のみをStick表示にしてみましょう.cartoonと水分子だけの画面に戻ったはずですね.
下段のコマンドは下記のような英語をイメージすれば分かりやすいかもしれません.
show sticks of the organic
そしてzoom コマンドで発色団に近寄りました.
show spheres, organic

コマンドの意味はもちろん”show spheres of the organic”
このコマンドによって発色団の原子をボール表示にすることが出来ます.このままだとspace-fillingモデルってやつですね.
set sphere_scale, 0.2
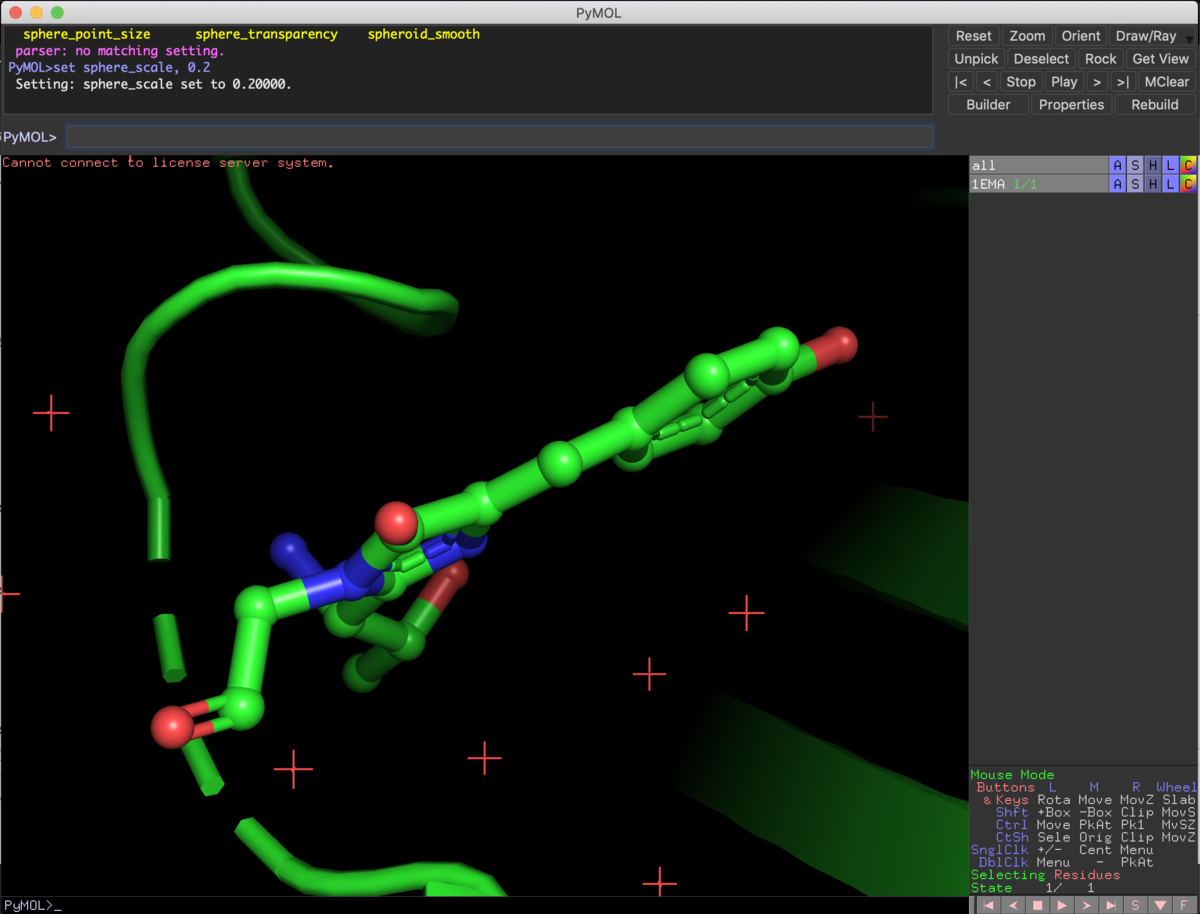
デフォルトのままだとBallが大きすぎるので,いわゆるBall and Stick modelというやつに変更するために,setコマンドでサイズを小さく変更しています.
set the scale (size) of the spheres for 0.2 という英文だとイメージしやすいですかね?覚えるのが面倒だと思うので,set sphまで入力した後にtabキーを押してみましょう.すると,External GUIにコマンドの候補が表示されます.候補が一つしかない場合は自動入力までしてくれます.困った時はtabを押してみるのも方法の一つです.
show lines, byres organic around 5
zoom organic around 10

show lineは先ほどのSticksと同じです.これらのコマンドは" , "の後にどの部分を表示するか指定することが出来ます.英語で書くと何となく意味がわかるでしょうか.
show lines of the residues, which is around (within) 5Å of the organic
みたいな意味です.つまりbyresは周辺残基という意味ですね.コマンドの方が実際の英文法よりも後付け感が強く感じるのは私だけでしょうか.
そして発色団とその周辺を観察したいので,下段のzoomコマンドで拡大しました.対象を大きくすることで縮小表示することもできます.
zoomとcenterは似ていますが,対象が右側サイドバーに表示されているオブジェクトなのか ,GUI内の指定部分なのかで使い方が異なります.(ハンターXハンターのスペル@GI編みたいな説明だな...)
Cartoonが邪魔で少し見づらいですか?
set cartoon_transparency, 0.7

数字を1>で大きくすると透けるので,少し見やすくなると思います.1<の値を入力すると全部透けるだけです.
水分子がチラついて集中出来ませんか?
extract sol, solvent

extractコマンドはオブジェクトを生成するコマンドの一つで,"solvent"を抽出して"sol"というオブジェクトを生成します.
extract the solvent to create an object "sol" みたいな意味になります.
同様に"select", "create", "copy_to"もオブジェクトを生成するコマンドです.
ちなみにremove solventというコマンドを使うと,全オブジェクトから水分子が一斉に削除されます.後で見たかったりするので,extract コマンドでオブジェクトとして保存しておく方が無難だと思います.
disable sol

disableコマンドは指定したオブジェクトを非表示にします.反対に表示したい場合はenableを使います.単発ではあまり便利に感じないかもしれませんが,複数オブジェクトを作成表示した後に,単一オブジェクトに切り替えたい時はdisable ! [target object]が便利です.マウスをたくさんクリックせずに済みます.
select 5A, organic around 5

select コマンドで対象(選択)部分の新規selectionオブジェクトを作成します.
マウスを使って特定の原子を選択していった場合は(sele)というオブジェクトが作成されますが,特定の選択部分についてオブジェクトとして保存できるということです.
これが出来ると何の役に立つんでしょうか?例えば...
label 5A and name ca, "%s%s" % (resi, resn)

labelコマンドを使って,発色団の5Å以内にある残基名を残基番号を表示することが出来ます.残念ながら,このコマンドは正直覚えるのが私は面倒です.そこで,このコマンドはサイドバー右側のL(Label)→residues (one letter)に譲ろうと思います.
create 5A, byres organic around 5

上述のselect コマンドと似ていますが,create コマンドは対象の新規オブジェクトを作成します.実際には下記のようなduplicateの方が直感的にしっくりくるような気もします.
create an duplicant object of the residue, which is around 5A of the organic
disable 1EMA

上述のcreate + aroundコマンドでは,発色団の周辺残基しかされていないはずです.そこで次のようなコマンドで発色団自体を複製します.
copy_to 5A, organic

コマンドが2つだと何だか二度手間に感じますよね.もちろん一発で発色団+周辺5Å
内残基のオブジェクトを作れます.そう,expandならね.
create 5Aa, byres organic expand 5
次に,発色団周辺について,もう少し詳しく見てみましょう.
disable all
enable 5Aa
zoom organic

GUI上を左クリックでドラッグして少し見やすくすると,64PHEから発色団が伸びて,68VALに再び戻っていることがわかると思います.
ついでに実際のSequenceも見ておきましょう.
set seq_view, 1

右下のSをクリックするだけで同じことが出来ますが,一応の紹介です.1でON,0でOFFになります.
Sequence viewerには配列と該当するアミノ酸残基が一文字表記されていますが,GFPの発色団はCROという表記で登録されているみたいですね.さてここで問題です.この発色団に該当する残基の新規オブジェクトを作成するコマンドは何でしょうか?
正解は...
create CRO, resi 65-67
create cro, resn CRO
あくまで一例ですが,こんな感じになります.resiは対象の残基番号を指定できます.一方,resnは残基名を指定できます.例えば,残基名としてHOHを指定すると,水,すなわちsolventを指定するのと同義になります.
reinitialize settings
ついでなんですが,cartoonの透過具合やsphereの大きさなど,やっぱ初期値に戻したい!みたいな時はreinitialize settingsで初期値に戻せます.単語の冒頭だけ覚えてtabで候補を出してくのをオススメします.
今回は以上です.
自分の備忘録には今回の形式がいいかも.
そういえばさっき気づいたんですが,私のpymolのライセンスが切れてますね...
学生さんはこちらからアカデミックライセンスを無償で入手できます.
興味のある方はお試しあれ.
Dear Pymol Beginners 1 -タンパクを切り分けたり重ね合わせたり-
Grüezi,
Hi, today I would like to introduce some pymol commands, which is useful for pymol biginers.
Let's get started!!
今日はPymolでよく使うコマンドの紹介をしたいと思います.初心者の方には結構便利じゃないかなと思います.Pymolのインストールについてはもっと詳しいブログがあるのでanacondaとか色々ググってみてください.
(酔った勢いで投稿しましたが,自分用の備忘録なので編集してます.)
本稿ではこんなことが出来るようになります.
-目的のタンパクをpymolで開く
-タンパクを主鎖ごとに分割してオブジェクトを作成する
-分割した主鎖を重ね合わせる
今回使うコマンドはこんな感じです.
- fetch 1EMA
- fetch 6FS8
- center
- center 1EMA
- center 6FS8
- split_chain
- delete 6FS8 1EMA_A
- enable 1EMA
- align 1EMA, 6FS8_A
- align 6FS8_A, 6FS8_B
- disable 1EMA
- center 6FS8_A
- remove chain B
- set_name 6FS8_A, CEN
では行ってみましょう.
fetch 1EMA
fetch 6FS8
If you use 'fetch 1EMA', at the left-center part of GUI, you can see the crystal structure of GFP protein, which shows the beutiful fluorescene after post-expression in the cell. You can also manage the objects at the right side column.
fetch 1EMA と入力すると,中央のGUIにGFPタンパクの結晶構造が現れます.これは細胞内とかで発現すると綺麗な蛍光を示すやつです.右手のオブジェクトカラムでは,表示する結晶構造を管理することが出来ます.
実際に開くと,下図とは少し違う風に表示されているはずです.今回の記事では,pymolのコマンドを使って自分好みの表示にする方法の一例を紹介します.ぶっちゃけマウスでポチポチやっても同じことが出来ますが,

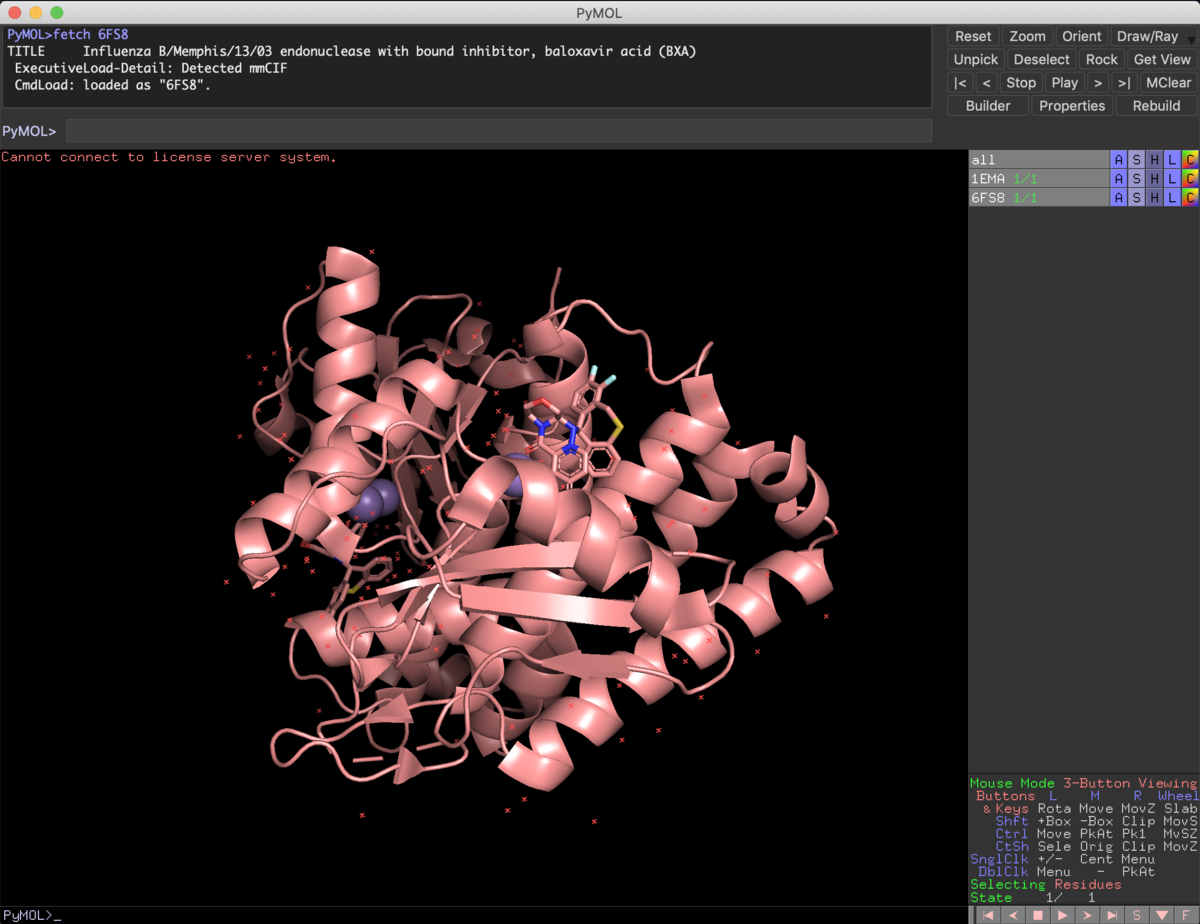
下図が6FS8です.1年前くらいに紹介したXofluzaのターゲットであるCENってやつですね.
center
と入力することで,表示されているタンパクを画面中央に持ってくることが出来ます.
fetchで複数の結晶を表示した場合,ほとんどの化合物は座標が合っていないので,未処理のデータはそれぞれの分子の重心部分が画面中心にくると思います.
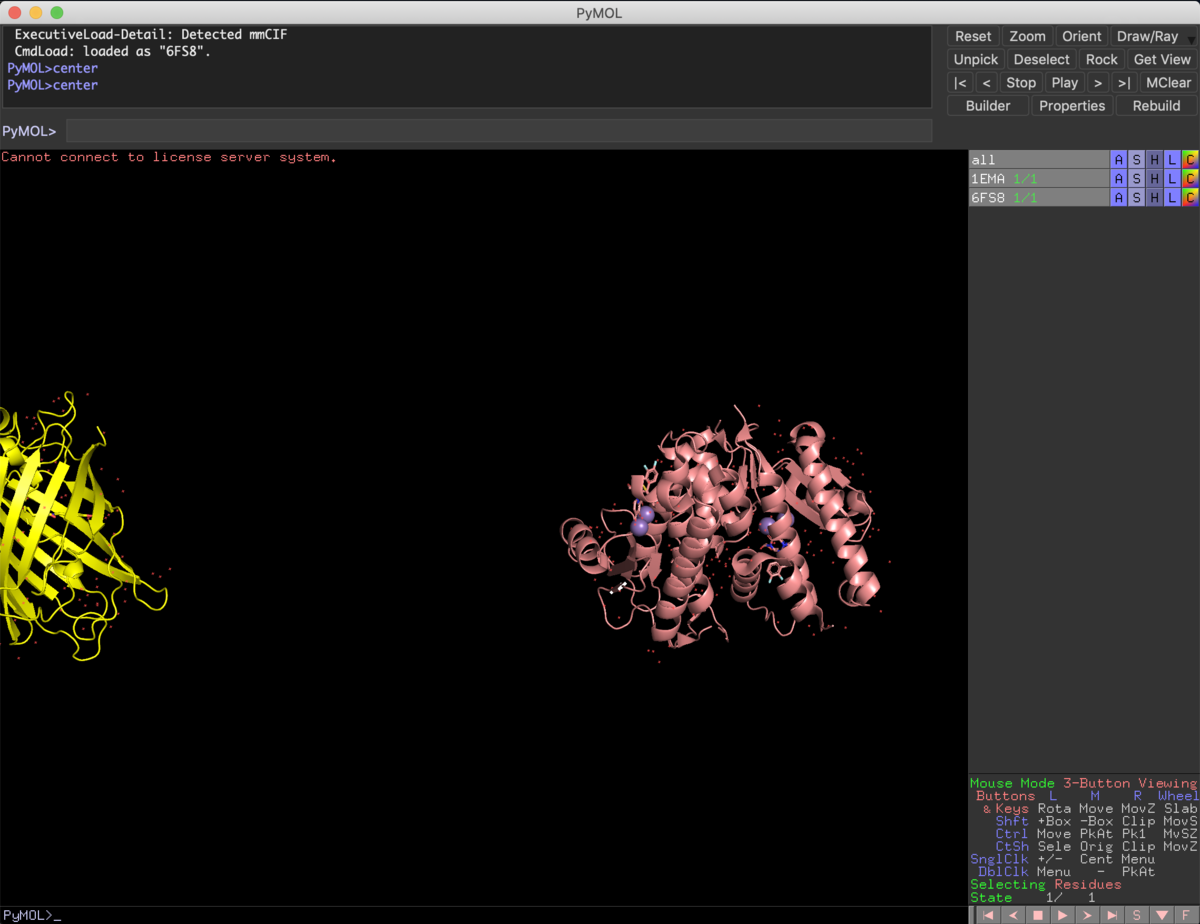
オブジェクトを指定することで,そのオブジェクトをGUIの中央に持ってくることも出来ます.例えば,
center 1EMA
で再びGFPが画面中央に戻ってくるはずです.
center 6FS8
でCENが再び中央に戻ってくると思います.
split_chain
タンパクの結晶というのは,二量体,三量体ましてや四量体で取れることもよくあるそうです.そんな時,主鎖ごとに分割したり,例えばリガンドが異なるなどの別の結晶情報と重ねてみたり,不要なChainは削除したりすることも出来ます.
split_chainと入力すると,表示している全てのオブジェクトの主鎖を別オブジェクトに分割してくれます.全て表示されたままなので,ぱっと見は変わりませんね.
delete 6FS8 1EMA_A
と入力すると,6FS8と1EMA_Aのオブジェクトは削除されます.複数を一度に削除したい時は続けて打ち込むだけでいいってことですね.アクティブだった1EMA_Aを削除したので,1EMAは非表示となってます.

注)delete allと入力すると全オブジェクトが削除されます.全てをやり直したい時だけは便利です.
enable 1EMA
と入力すると,1EMAのオブジェクトがアクティブになります.

ちなみにenableやdisableは対象のオブジェクトを入力しないと全てのオブジェクトに適用されます.全表示or全非表示みたいな感じですね.試してみてください.
align 1EMA, 6FS8_A
次に上記のコマンドでタンパクを重ね合わせてみますが,alignは日本語では揃えるみたいな意味です.英文にするとalign 1EMA to 6FS8_A でしょうか.このコマンドで1EMAが6FS8_Aに重なったと思います.全然関係ないタンパクなんで重なり合って見えませんね.
align 6FS8_A, 6FS8_B
disable 1EMA
center 6FS8_A
次にA鎖とB鎖を重ね合わせた後,1EMAを非表示にして6FS8_Aを画面中央に表示しています.
A鎖とB鎖で微妙に構造が違うので,ちょっとずつズレているのがみて取れますね.ちなみにズレを定量化したのがRMSDというパラメーターです.そのうち勉強したら記事にします(願望).

さて,先ほどはオブジェクトを削除するdeleteを紹介しましたが,オブジェクトの中から特定の構造を[remove]で削除することも出来ます.
remove chain B
で全てのオブジェクトからB鎖を除去することが出来ます.当たり前かもしれませんが,B鎖に付属する有機分子や水なんかも一緒に除去されます.そして除去してしまうと元には戻せません.非表示にするならどんなコマンドだったでしょうか.disable object name ですね.
set_name 6FS8_A, CEN
最後におまけですが,set_renameコマンドでオブジェクト名を変更できます.コマンドの記述形式に慣れてきたらわかると思いますが,コマンドの意味は set the object name of 6FS8_A to CEN みたいな意味です.
さて,長くなってきたので今日はこの辺で終わろうと思います.
次回はリガンドに着目したコマンドの例を紹介したいと思います.
結局DNA encoded libraryってどんなんなのさ?
Gruezi wohl
最早三ヶ月前の話なので大変恐縮ですが,ETH(@Zurich)で毎年のように開催されているDNA encoded library (いわゆるDEL)のシンポジウムに参加してきました.ぶっちゃけ文献やネットのまとめ的な記事しか読んでいなかったので,今回のシンポジウムはケミストの視点から刺激的だったので,重い腰をあげて記事にしたいと思います.
DNA encoded libraryというのは,ざっくり言うとDNAタグ付きの低分子ライブラリのことです.通常のライブラリというのは1化合物につき1バイアルなので,High Throughput Screening (HTS)で何百万化合物をアッセイしようとすると,例えば1µMのsingle point assayだとしても,どえらい時間が必要になります.しかも化合物の純度なんてぶっちゃけ分からないので,活性が出た!と思っても再合成+評価したら活性なくなった...なんてこともあるそうです(ちなみに私自身は直接携わったことがありません...).一方,DELのライブラリは10億個(1billion)スケールの化合物がたった1本のeppendorf tubeの中に作製され,target proteinとの結合評価を行った後に,DNAシーケンサーを用いて化合物の同定を行います(Affinity selection).この時点では化合物がtarget proteinに何かしらの形で結合することしか分からないので,DNAタグなしのリガンドを再合成して活性の評価を行います.そのため,HTSと比較して大きなスケールのライブラリを短期間でスクリーニング出来る手法として注目されています.(@masayayamaguchさんのブログの方がよくまとまってるので,そちらも参考にしてください.http://luckprepopp.com/pharma-biotech/screening-del-hts/)
へーそうなんだ.
と,思って深く調べていなかった私が浅はかでした.
DNAタグって実際どんな構造か想像出来ますか?
下記のキーワードでググるとDEL作製に必須のHeadpieceと呼ばれる部分の構造の画像がヒットします.
dna encoded library headpiece
ちなみにリンク先は文献なので,購入するかライセンスがないと読めません.
From haystack to needle: finding value with DNA encoded library technology at GSK
しかし,化学構造が多少わかる人であれば,なるほどDNA二重螺旋の先にPEGを介してアミンが付いてんのね,と理解してもらえると思います.DELはこのHeadpieceを起点としてオリジナルライブラリを作製できるテクノロジーです.
このアミン部分に対して化学反応を,DNA部分に対してBuilding Blockに対応したDNAタグをLigateすることでライブラリを作製していきます.
そしてもう一つ.DELの特徴的な作製方法の一つが”Split and Pool”です.簡単に言うと,混ぜては分けて,また混ぜて,と言うことです.もう少し具体的に説明してみましょう.
先ほどの文献の図が非常にイメージしやすいので,そのまま拝借したいと思います.まずはHeadpieceの溶液を96-wellのプレートに均等分割し,各wellにそれぞれ個別のシーケンスを伸長します(Ligation).続いて各wellに対して,異なるBuilding Blockを用いて同一の化学反応(例えばSnAr反応)を行います.この段階で,各Buiding Blockに対応したDNAタグが付与された状態です.Split and Poolでは,例えば一つのeppendorf tubeの中に96成分を一旦集約してしまいます(Pool).続いて,これを96-wellに再度分割し(split),同様の工程を行います.つまり,2サイクルが終了した時点で,DNAタグ付きの96x96=9216化合物が1本のTubeの中に構築できることになります.これを3サイクル回すだけで884,736化合物,4サイクル回すとあっという間に84,934,656(約8500万)化合物のDNAタグ付きライブラリが,たった一本のTubeの中に構築出来てしまうわけです.96-well plateを使えば,単純に96^nのライブラリを構築出来ると言うわけですね.
ライブラリの規模は正に指数関数的に増幅出来るわけですが,分子構造を理解できるケミストであれば,その欠点も理解出来るかと思います.以下,ぱっと思いつく点を挙げていきます.
-規模の大きいライブラリほど分子量は増大する傾向にある(基本的に分子の足し算)
-ライブラリの多様性は,用いる化学反応とBuilding Blockに依存する
-DNAを有する構造の特徴上,使用できる化学反応は水溶性溶媒に制限される
-DNAを有する構造の特徴上,DNAと反応するような化学反応は使用できない(DNA compatibleな化学反応である必要がある)
-ライブラリ自体の分析が難しい(純度や,そもそもLigandが合成出来ているか)
-もちろん精製も難しい(現時点でHPLCに依存)
私自身はDELに携わっていないので詳細は言及出来ませんが,シンポジウムで議論した時点では上記の印象を持ちました.PfizerやGSKなどのメガファーマは,独自のライブラリ構築に加えて,上記の課題克服に向けた基礎研究を発表していました.
ここまでで,DNA encoded libraryって結局どんなのなの?と言うのをケミストの目線で列挙してみました.
率直に言って,私はめちゃくちゃ面白いなと思いました.
最近流行ってるPhotoredoxなんかも,DNA compatible reactionとして報告されています(Open-Air Alkylation Reactions in Photoredox-Catalyzed DNA-Encoded Library Synthesis).ご自身のラボの強みと言える反応開発が,創薬研究にダイレクトに活用できるかもしれません.アウトプット先が常に構造ベースの議論になってしまいがちの方には,ライブラリ化することで,例えば創薬や農薬開発に活用できないかを検討してもらえるかと思います.
この記事の作成時点(2019/12/4現在)で,日本ではDELが特には流行ってないのかなと思います.投資が必要な設備ももちろんあるとは思いますが、Wetのアウトプットとして,DNA encoded libraryは如何でしょうか?