Dear Pymol Beginners 1 -タンパクを切り分けたり重ね合わせたり-
Grüezi,
Hi, today I would like to introduce some pymol commands, which is useful for pymol biginers.
Let's get started!!
今日はPymolでよく使うコマンドの紹介をしたいと思います.初心者の方には結構便利じゃないかなと思います.Pymolのインストールについてはもっと詳しいブログがあるのでanacondaとか色々ググってみてください.
(酔った勢いで投稿しましたが,自分用の備忘録なので編集してます.)
本稿ではこんなことが出来るようになります.
-目的のタンパクをpymolで開く
-タンパクを主鎖ごとに分割してオブジェクトを作成する
-分割した主鎖を重ね合わせる
今回使うコマンドはこんな感じです.
- fetch 1EMA
- fetch 6FS8
- center
- center 1EMA
- center 6FS8
- split_chain
- delete 6FS8 1EMA_A
- enable 1EMA
- align 1EMA, 6FS8_A
- align 6FS8_A, 6FS8_B
- disable 1EMA
- center 6FS8_A
- remove chain B
- set_name 6FS8_A, CEN
では行ってみましょう.
fetch 1EMA
fetch 6FS8
If you use 'fetch 1EMA', at the left-center part of GUI, you can see the crystal structure of GFP protein, which shows the beutiful fluorescene after post-expression in the cell. You can also manage the objects at the right side column.
fetch 1EMA と入力すると,中央のGUIにGFPタンパクの結晶構造が現れます.これは細胞内とかで発現すると綺麗な蛍光を示すやつです.右手のオブジェクトカラムでは,表示する結晶構造を管理することが出来ます.
実際に開くと,下図とは少し違う風に表示されているはずです.今回の記事では,pymolのコマンドを使って自分好みの表示にする方法の一例を紹介します.ぶっちゃけマウスでポチポチやっても同じことが出来ますが,

下図が6FS8です.1年前くらいに紹介したXofluzaのターゲットであるCENってやつですね.
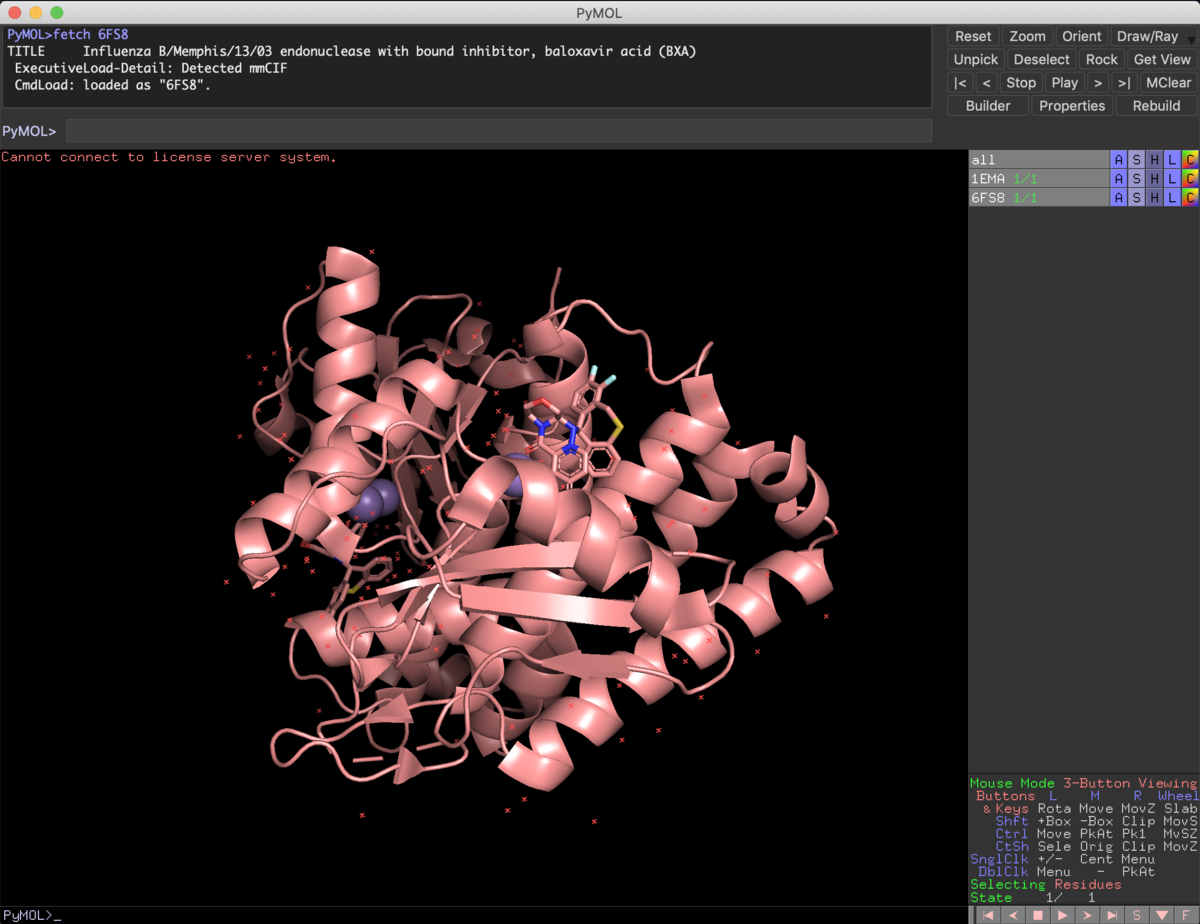
center
と入力することで,表示されているタンパクを画面中央に持ってくることが出来ます.
fetchで複数の結晶を表示した場合,ほとんどの化合物は座標が合っていないので,未処理のデータはそれぞれの分子の重心部分が画面中心にくると思います.
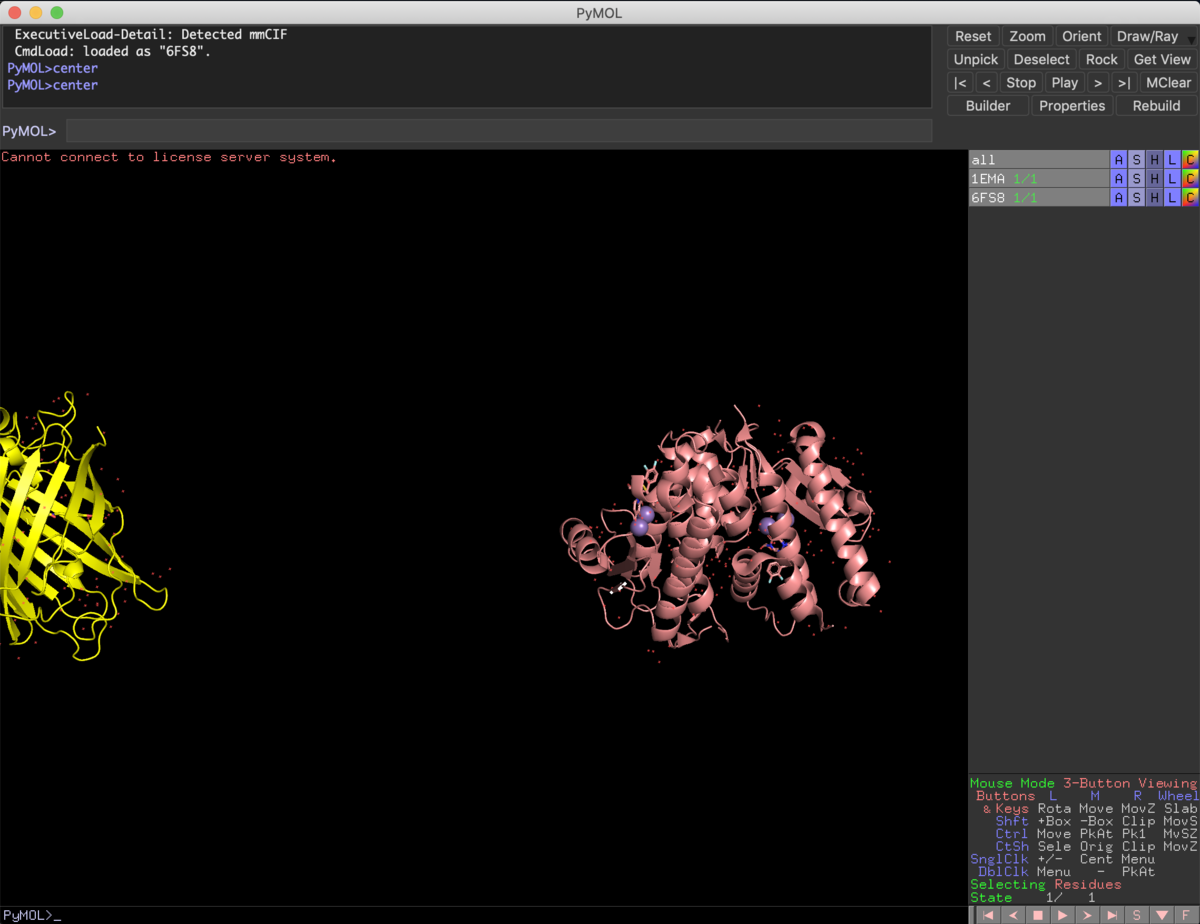
オブジェクトを指定することで,そのオブジェクトをGUIの中央に持ってくることも出来ます.例えば,
center 1EMA
で再びGFPが画面中央に戻ってくるはずです.
center 6FS8
でCENが再び中央に戻ってくると思います.
split_chain
タンパクの結晶というのは,二量体,三量体ましてや四量体で取れることもよくあるそうです.そんな時,主鎖ごとに分割したり,例えばリガンドが異なるなどの別の結晶情報と重ねてみたり,不要なChainは削除したりすることも出来ます.
split_chainと入力すると,表示している全てのオブジェクトの主鎖を別オブジェクトに分割してくれます.全て表示されたままなので,ぱっと見は変わりませんね.
delete 6FS8 1EMA_A
と入力すると,6FS8と1EMA_Aのオブジェクトは削除されます.複数を一度に削除したい時は続けて打ち込むだけでいいってことですね.アクティブだった1EMA_Aを削除したので,1EMAは非表示となってます.

注)delete allと入力すると全オブジェクトが削除されます.全てをやり直したい時だけは便利です.
enable 1EMA
と入力すると,1EMAのオブジェクトがアクティブになります.

ちなみにenableやdisableは対象のオブジェクトを入力しないと全てのオブジェクトに適用されます.全表示or全非表示みたいな感じですね.試してみてください.
align 1EMA, 6FS8_A
次に上記のコマンドでタンパクを重ね合わせてみますが,alignは日本語では揃えるみたいな意味です.英文にするとalign 1EMA to 6FS8_A でしょうか.このコマンドで1EMAが6FS8_Aに重なったと思います.全然関係ないタンパクなんで重なり合って見えませんね.
align 6FS8_A, 6FS8_B
disable 1EMA
center 6FS8_A
次にA鎖とB鎖を重ね合わせた後,1EMAを非表示にして6FS8_Aを画面中央に表示しています.
A鎖とB鎖で微妙に構造が違うので,ちょっとずつズレているのがみて取れますね.ちなみにズレを定量化したのがRMSDというパラメーターです.そのうち勉強したら記事にします(願望).

さて,先ほどはオブジェクトを削除するdeleteを紹介しましたが,オブジェクトの中から特定の構造を[remove]で削除することも出来ます.
remove chain B
で全てのオブジェクトからB鎖を除去することが出来ます.当たり前かもしれませんが,B鎖に付属する有機分子や水なんかも一緒に除去されます.そして除去してしまうと元には戻せません.非表示にするならどんなコマンドだったでしょうか.disable object name ですね.
set_name 6FS8_A, CEN
最後におまけですが,set_renameコマンドでオブジェクト名を変更できます.コマンドの記述形式に慣れてきたらわかると思いますが,コマンドの意味は set the object name of 6FS8_A to CEN みたいな意味です.
さて,長くなってきたので今日はこの辺で終わろうと思います.
次回はリガンドに着目したコマンドの例を紹介したいと思います.