Dear Pymol Beginners 3 x KNIME 1.3 -リガンドのSDFファイルを書き出してみたり,リガンドの結合様式を比較してみたり-
Grüezi,
Appleで春の学生向けキャンペーンが明日までですね.
新しく出たMacBook AirとiPad Proを順番にコンボで買える日を夢見てましたが,予算の都合上,後者だけになりました笑.オマケのギフトカードで念願のApplePencil2も買うことにします!やっほい!
ひとまず8年選手のMacBook Air2012を使い続けるために,Mojaveをクリーンインストールして...画面が映らない騒動が勃発し...結局あんまり動作は軽くならずに限界を感じつつ...とかやって,ひとまずKNIMEとanaconda, Pymolをインストールし直しました.日本に帰ってからもKNIME遊びは続けられるかなぁ...
と言うわけで今回のWorkFlowでは
- RSC PDBのサイトからPDBとレポートの取得(前回までのマイナーチェンジ)
- レポートからリガンドをSDFとして出力
することが出来ます.
最後に,このWorkFlowの利用例をPymolを交えて紹介してます.
*前回,前々回の重複内容は省きます.
- WorkFlowの全体像
- PDB Connector
- PDB Saver
- Excel Sheet Appender
- RDKit To Molecule
- String Manipulation
- Group Loop Start
- SDF writer
- Variable Loop End
- String Manipulation
- String To URl
- Copy/Move Files
- 実行結果
- 利用例の紹介
- Pymolで観察
- set grid_mode, 1
- show surface, byres organic around 5
- (set surface_color, gray)
WorkFlowの全体像
上段と中段は前回とほぼ変わりません.中段先頭に置いていたMolecule Type Castは不要とわかったので削除しました.
下段のWorkFlowでは,PDBのレポートに含まれるSMILESからSDFファイルをPDB IDごとに生成しています.残念ながら今回は利用例を紹介するところまではたどり着けませんでしたが,このWorkFlowを使ってどんなことが出来るかを想像するところを宿題ってことにさせてください.(To be continued...)
PDB Connector
IDs and Keywordsは前回と同じくSARS-CoV-2です.
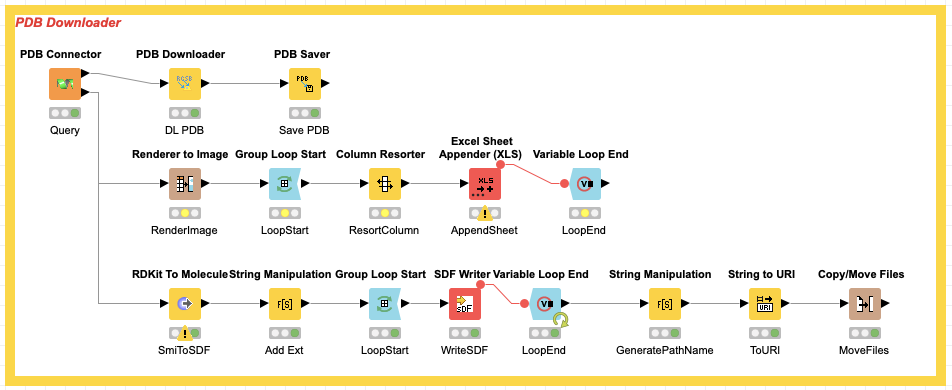
PDB Saver
今回はデスクトップ上に架空ユーザー(xxx)のフォルダを指定しました.
Excel Sheet Appender
今回は上記の架空ディレクトリと同じ場所を指定しています.シート名を変数にして出力するのは前回までと一緒です.

RDKit To Molecule
PDB Connectorから得られるreportのSMILES情報は既にSMILESとして機能してるみたいです.なので,このNodeを使ってSMILESをSDFに変換します.SMILESの方が都合がいいこともあるので,SourceColumnは残してます.Destination FormatはSDFですが,他のファイルも指定できます.

String Manipulation
変換したSDFファイルをリガンドごとに個別に出力したいのですが,そのまま出力するとファイルに拡張子(.sdf)が付いていません.そこで,このNodeで拡張子付きのStringを生成しておきます.ここではPDB IDとしておきます.

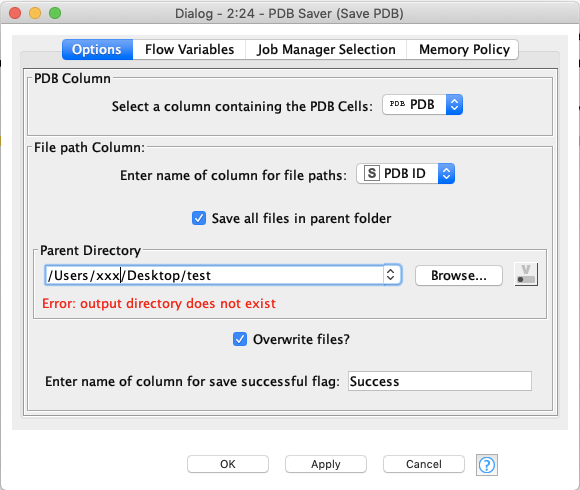
Group Loop Start
上述で生成した拡張子付きのリガンド名ごとにファイルを作りたいので,前項でAppendしたLigandPDBIDというカラムを指定してLoopをスタートします.このカラムについて1行ずつ繰り返し操作を行うってことですね.
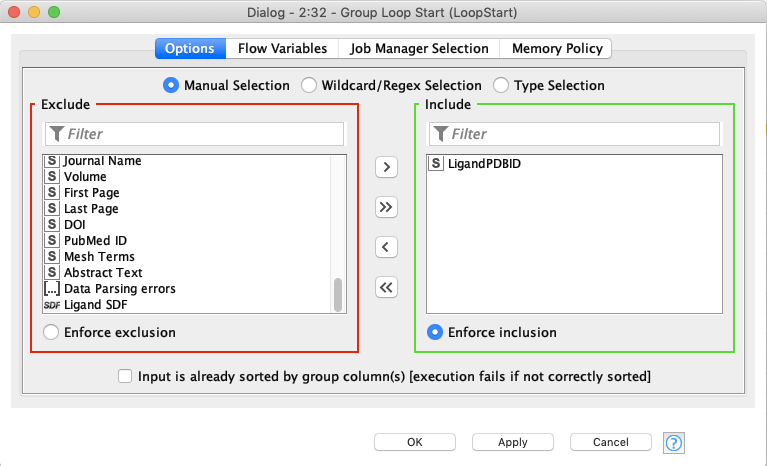
SDF writer
今回は書き出すSDFファイル名が変数になるように設定します.ファイル名の右側にあるvをクリックして,Use VariableからLoopするカラムを指定します.Use Column as molecule titleにチェックしておくと,SDFごとに分子に名前がついて後々便利だったりします.
Variable Loop End
特に設定しません.
String Manipulation
個人的にはここが今回一番トリッキーでした.
前々項のSDF Writerはお気付きの通り,出力先を指定していません.すると,私の場合,デフォルトでは/Applications/KNIME 4.1.0.app/Contents/MacOS と言うディレクトリに保存されていました.なので,このSDFファイルをPDBファイルと同じディレクトリに移動させたいと思います.単純に便利だからです.
このNodeでファイルが出力された場所を指定します.ディレクトリ末尾に/を追記した/Applications/KNIME 4.1.0.app/Contents/MacOS/ にLigandPDBIDと言うファイル名のカラムを追記していきます.

String To URl
前項で生成したStringをURlに変換します.

Copy/Move Files
最後にSDFファイルの出力先からPDFのレポートと同じディレクトにファイルを移動させます.
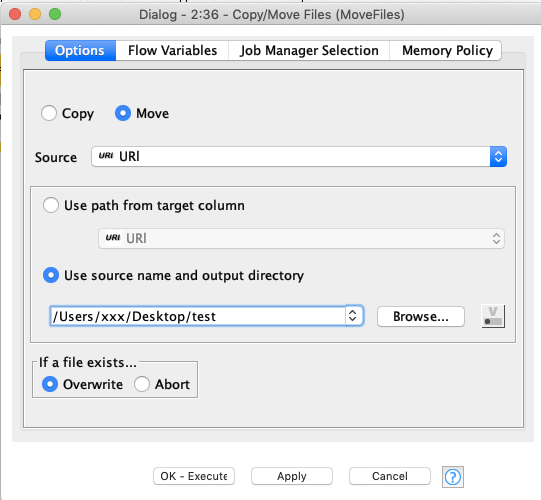
実行結果
以上を実行するとこんな感じになります.

べらぼうな数が吐き出されるので途中は割愛してますが,リガンドのSDF,レポート,PDBが同じフォルダに集まりました.
前回軽く紹介したPDB ID:6Y2Fなんかは,リガンドがDMSOを含めて2つとされています.

この場合,SDFファイルには2つの分子がリガンドとして出力されています.

利用例の紹介
ついでにさっきのレポートの中から,同じターゲットの別のリガンドを探してみたいと思います.main proteaseで検索をかけると,例えばペプチドアナログ以外にこんなリガンドが見つかりました.今回は出来ませんでしたが,IC50値などの活性値が報告されていると,絞り込みの方法はグッと広がると思います.

*追記
ペプチドアナログ以外を見つけたいとか書いておきながら、思いっきりペプチドアナログを見つけてますね…。深夜の疲労のせいということでご愛敬…。
Pymolで観察
このPDB ID:6W63と,6Y2FをPymolで読み込んでみましょう.

今回はそれぞれをfetchで呼び出した後,preset > ligand site > cartoonで表示しています.
次に6W63のオブジェクトから,align > to molecule > 6Y2Fで重ね合わせます.zoom organicでリガンドに照準を合わせるとこんな感じです.

6W63のリガンドは思いっきりペプチドアナログと同じBinding Siteに来てますね!次のコマンドラインでGrid表示してみましょう.
set grid_mode, 1
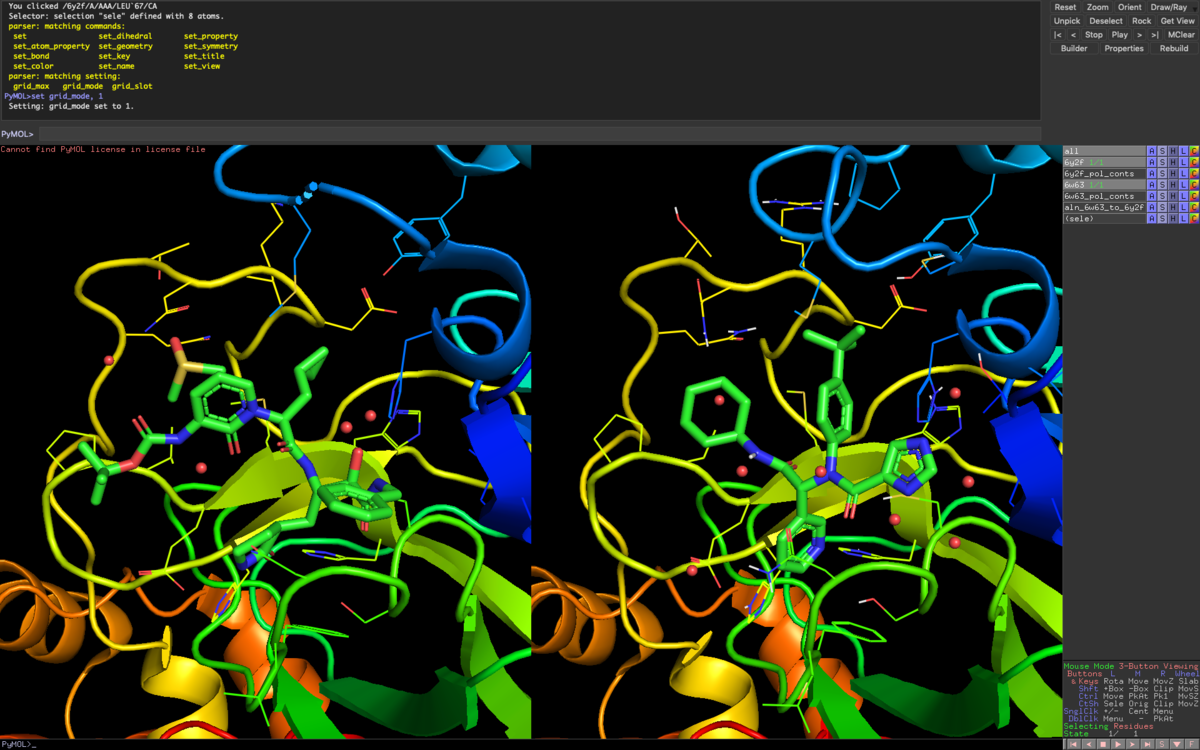
polar contactは一旦非表示にしました.ついでに6W63のnon polar hydrogenも非表示にしてみやすくしてます.次にポケットの比較をしてみます.
show surface, byres organic around 5
(set surface_color, gray)
リガンド周辺部だけSurfaceを表示しました.特に設定をしてない場合は見辛い配色だと個人的に思うので,色を灰色に変えました.

シクロプロピルメチル基が埋めていた(確か)S2ポケットと呼ばれる脂溶性空間にt-Bu基がうまくはまってますね.(下図)

S1ポケットと呼ばれるグルタミンサロゲートが埋めていた部分は3ーピリジルで置換されています.HBDいらないんですね.

件の共有結合部分を観察してみましょう.
6Y2FではC145から思いっきり共有結合しているので,その距離は1.8Åです.

一方,6W63ではC145と共有結合こそ形成していませんが,おそらく該当するであろうアミドとの距離は3.4Åです.プロテアーゼでは通常,コアの反対側にあるヒスチジンからHBDのアシストを受けて加水分解の活性化を受けたりするんですが,今回はそういった残基も特には見当たりませんでした.原子間距離自体は近いので,リガンドの結合にこのアミドは何かしら関与してそうな気はするんですけどね.

他にも細かい相互作用解析はあるんですが,今回はこの辺で切り上げようと思います.
出力したSDFファイルの使い道もWorkFlowが出来たら紹介したいと思います.