Dear Pymol Beginners x KNIME 1.4 -リガンドをクラスタリングして,レポートとPDBファイルのダウンロード先の振り分けに使ってみたり-
Grüezi,
早いもので,帰国まで1ヶ月を切ってしまいました.慣れてしまえばもう少し滞在したいような,でも物価の高さには相変わらず目眩もするし,正直多分虫歯が出来たっぽいしで帰国したいような,そんなどっち付かずの心持ちです.計画してた旅行の残りはコロナウィルスのせいでオールキャンセルなので,やっぱり心残りだなぁ.
さて,最近もっぱらVirtual Screeningなんかをやってるんですが,Libraryが大きくてなかなか終わりません.という訳で,KNIMEを使って合間に出来るPDBの情報収集方法の備忘録です.
今回はざっくりこんな感じのことが出来ます.
- RSC PDB Bankのサイトから取得したPDBの情報からリガンドをClusteringする
- Clusterごとの情報をExcelにExportする
- PDBをClusterごとにフォルダーにダウンロードする
今回紹介するNodeはこちら
- Workflowの全体像
- PDB connector
- Row Filter
- Fingerprints
- Distance Matrix Calculate
- k-Medolds
- Group Loop Start
- Value Counter
- TableRow To Variable
- String Manipulation (Variable)
- Excel Sheet Appender
- Variable Loop End
- Create Directory
- String Manipulation (Variable)
- PDB Saver
- Loop End
- Excel Reportからリガンドの確認
今回も,す(@sumtat_)さんのところのブログを大いに参考に(パクって)しています.いつも勉強になりますありがとうございます.
そんな訳で,今回も行ってみましょう.
Workflowの全体像
Workflowの左側でPDB情報を取得,Filtering,Clusteringを行なっています.分岐した上段では,クラスターごとに情報をグループ化してExcelにシートごとにExportしています.下段では,クラスターごとにDirectoryを作ってPDBファイルをダウンローとしています.
青で囲ったClusteringの部分が今回の肝ですが,思いっきりパクって使わせてもらってます.緑で囲った部分はClusterの命名に便利だなと思ったので採用(つまりパクリ)しましたが,他にいいアイディアがあれば代用していいと思います.つまり何が言いたいかというと,詳細な説明は先達に譲ります(!).

PDB connector
今回はIDs and KeywordsタブのText Searchに"SARS-CoV-2 main protease"と入力しています.Report OptionsはStructure Summary, Ligands, Binding Affinity, Primary Citationをチェックしています.
Row Filter
MW<100のリガンドをFilter Outしています.目的としては溶媒や添加剤,apo体を除くことです.なので,分子量をもう少し大きくしてもいいかもです.

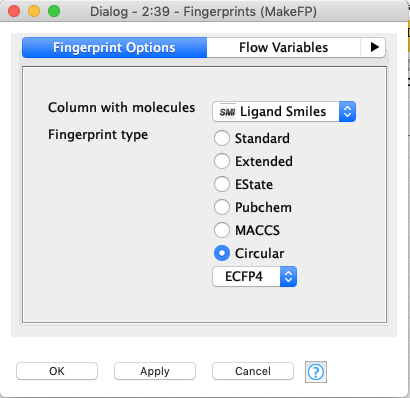
Fingerprints
LigandのいわゆるFingerprintを作ってくれます.設定はパクったので,ぶっちゃけ詳細は説明できません...
Distance Matrix Calculate
同じく詳細は割愛します.
ざっくり言うと,前項で作ったFingerprintの距離を総当たりで計算して吐き出す,みたいなNodeらしいです.かの有名なTanimoto法でお願いします.

k-Medolds
このNodeでClusteringしてくれます.Partition Countで発生させるクラスターの数を選べます.今回はまんまパクって5個にしてます.

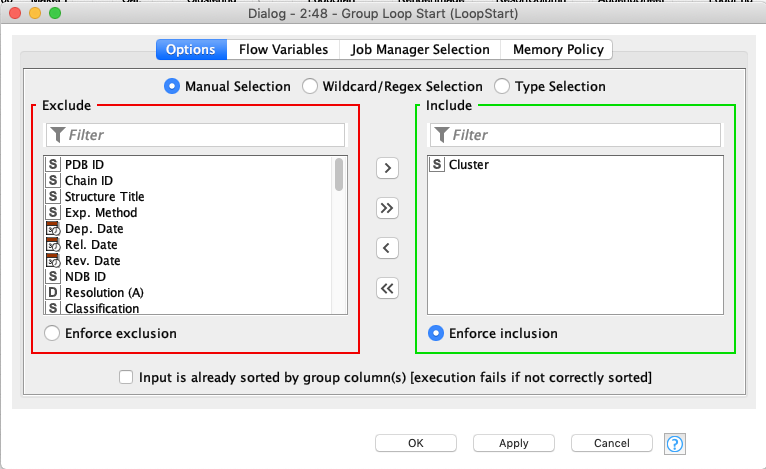
Group Loop Start
発生させたClusterを元にGroup化を繰り返すLoop Nodeです.上段も下段も設定は同じです.
Value Counter
Group化したClusterの中にあるLigandの数を数えます.

TableRow To Variable
Countした数を変数に変換します.特に設定不要です.
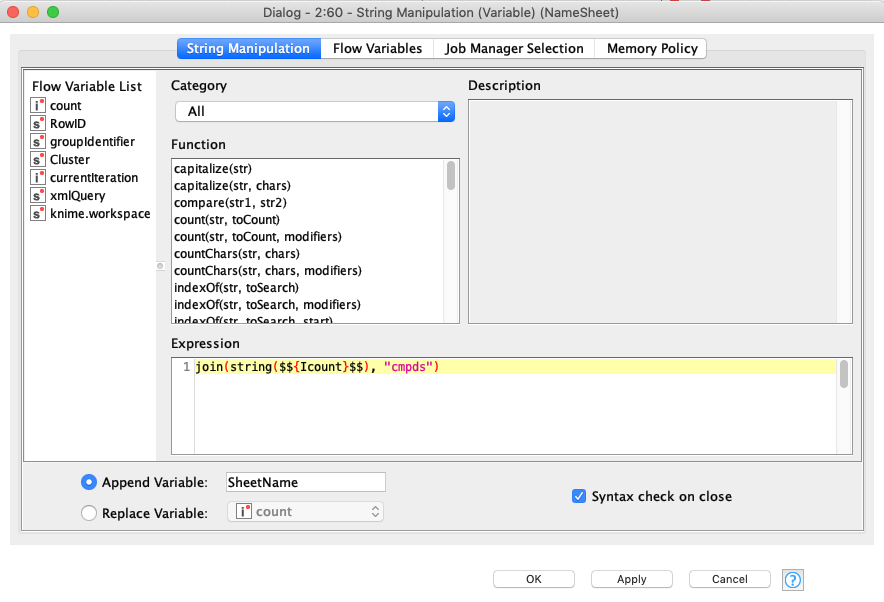
String Manipulation (Variable)
前項で発生させた変数を受けてStringを発生させます.今回は数えた数をそのままExcelのシート名にします.はい,まんまパクってます.
Excel Sheet Appender
ExcelにPDB情報をシートごとにまとめます.File名は自由に設定して,シート名を前項までに作った変数にします.add column headersにチェックしておいた方がいいです.

Variable Loop End
Loopの終わりを指定してあげます.
Create Directory
ClusterごとにPDBファイルを振り分けるDirectory(フォルダ)を作りたいのですが,ちょっとTrickyです.
Locationは前述のExcel Reportと同じ場所を指定しておきます.フォルダ名に変数を指定したいのですが,なぜかここに変数指定ボタンがないので,適当なダミー名(今回はa)を入力した上でFlow Variableのタブから改めて変数を指定します.
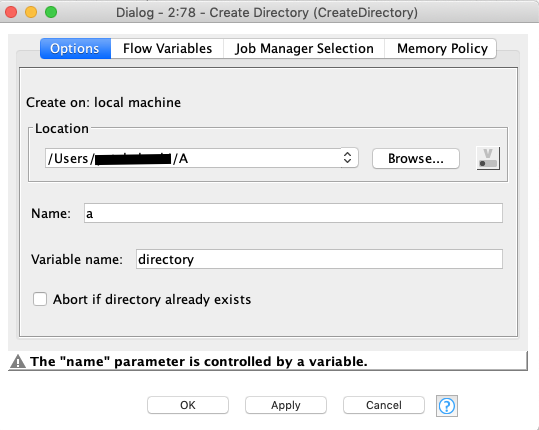
Directory名をTable To Variableで発生させたカラム名に指定します.

実行結果はこんな感じになればOKです.directory名に指定した変数(今回はクラスター内のLigandの数+cmpds)が反映されています.

String Manipulation (Variable)
前項で発生させたdirectoryのValueをPDBの保存先に指定できればよかったのですが,残念ながら"file:"という文字列が先頭に付いてるのでPDB Saverの保存先に使えません.そこで,いつものString Manipulationで保存先を発生させます.Sheetnameを変数に使えばOKです.

PDB Saver
保存先として,Parent Directoryの変数に先ほど発生させたdirectoryを指定します.

保存に成功すると,実行結果の右端にtrueと表示されます.何かしらエラーがあった場合はFailsになります.
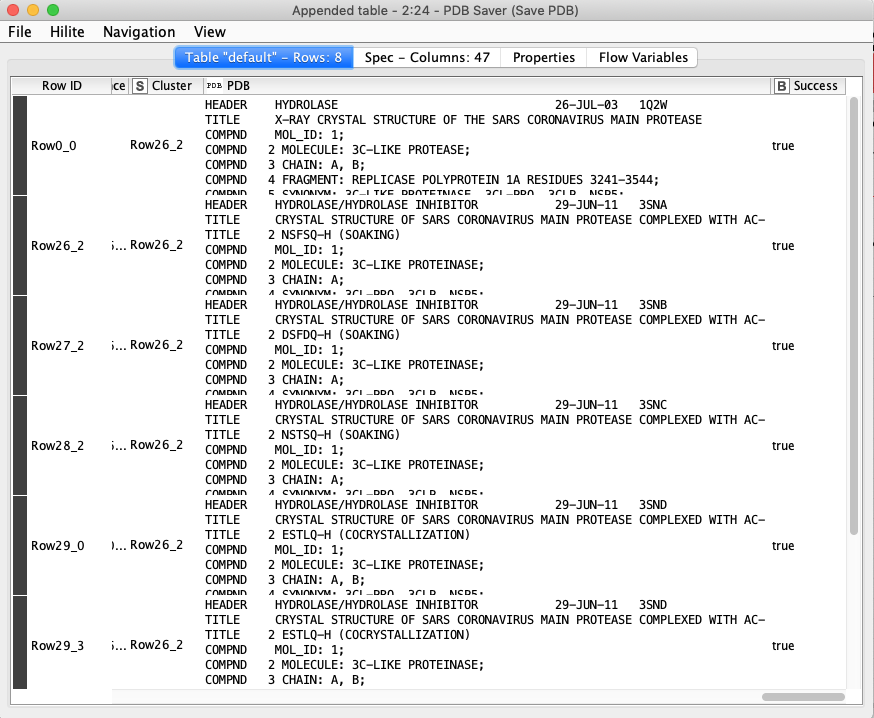
Loop End
Loop処理の終了を宣言します.
実行結果
2020/04/21現在で,実行結果はこんな感じになります.ちゃんと5つのClusterに分かれてPDBファイルが保存されました.あれ?11化合物あるはずなのにファイルは8つ?これは一つのファイルの中に,例えばA鎖とB鎖がある場合,リガンド数が2個とカウントされるためです.実際には一つのPDBファイルの中に格納されています.

PDB Saverの実行結果を見ると納得かと思います.
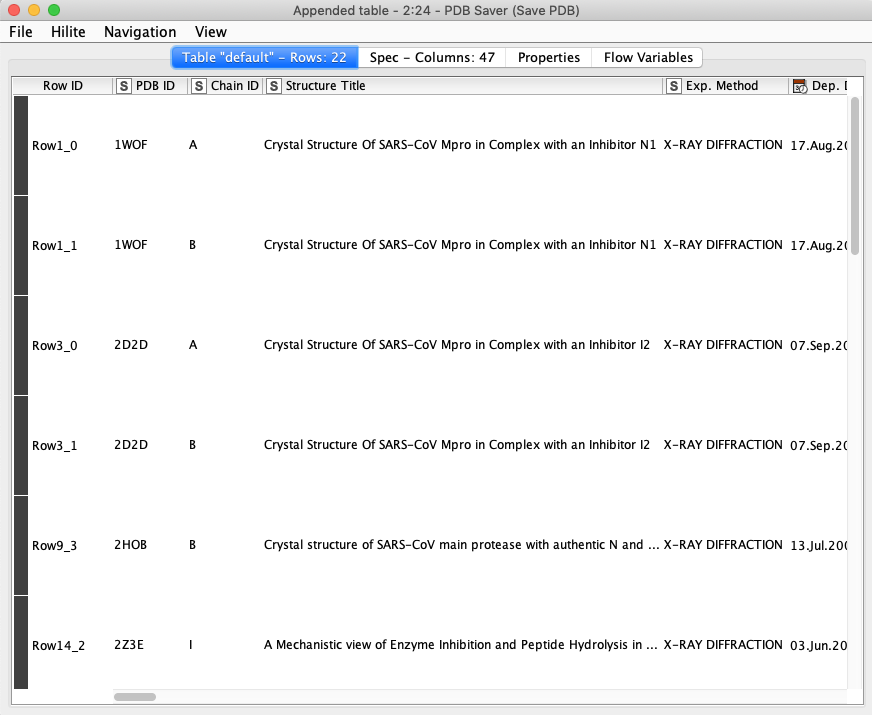
Excel Reportからリガンドの確認
さて,Excelを開いてみましょう.
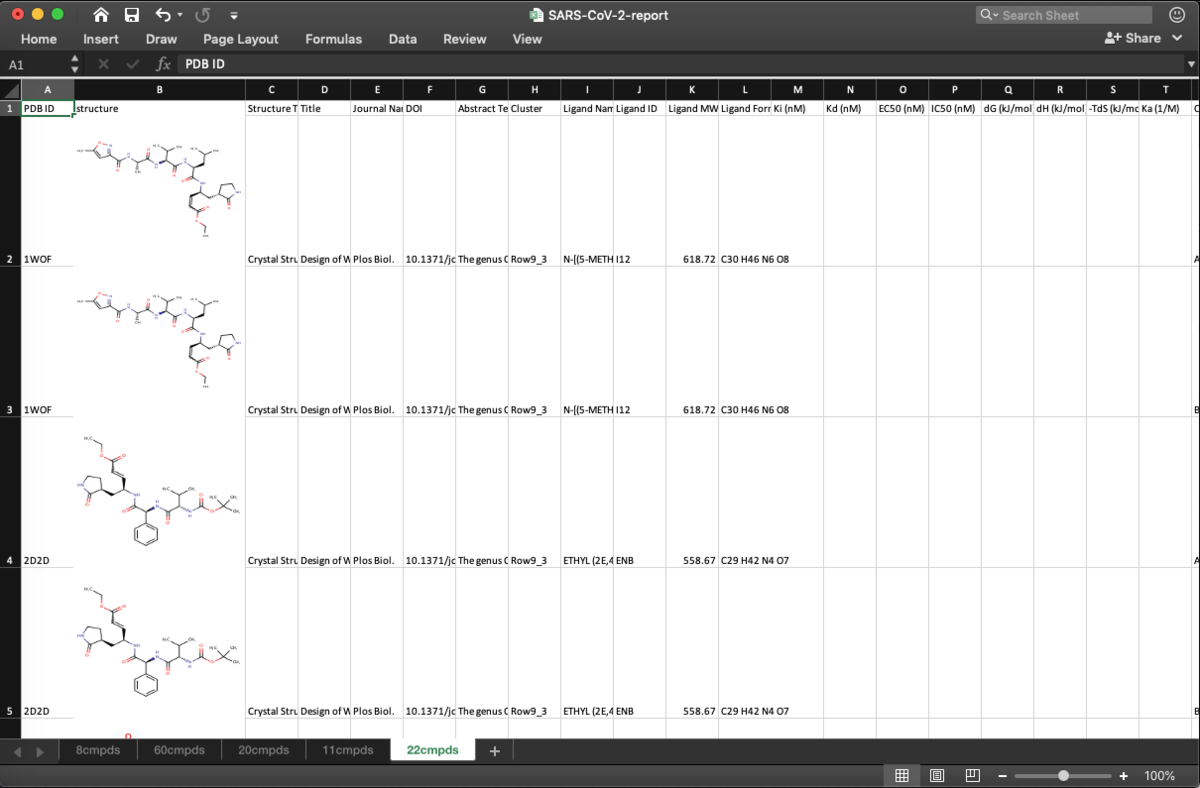
ちゃんと5つのClusterごとにシートが作られてますね.数が一番多い60cmpdsのシートには,先日紹介したX-Chemのフラグメントスクリーニングの結果が含まれています.ExcelのFilter機能を使って絞り込むと,他にもリガンドが見つかりますね.前々回の記事で紹介したリガンドは,こんな感じで見つけてきました.

ついでに22cmpdsのシートも開いてみると,ここにはペプチドミミックのリガンドがまとまってました.中身がさっぱりわかってなくても,Clusteringが結構うまくいってるんだなぁと関心です.
こんな感じでClusterごとに振り分けたPDBファイルを必要な分だけPymolに読み込んでGroup化してやると,プロジェクト初期には有用だったりしないかなと思います.
今回みたいに取得した情報の振り分けを自動化出来ると,特許解析とかに応用出来ないのかなとか妄想中です.
ご参考までに.
追記)後から気が付いたんですが,Creat DirectoryとかしなくてもString Manipulationで保存Directoryを指定してやれば勝手にDirectoryが作成されました.まぁ,Variableの練習をしたってことでご愛嬌.