Dear Pymol Beginners 4 -オブジェクトをグループ化してみたり,リガンドの結合位置や様式を比べてみたり-
Grüezi,
世界中でロックダウンが行われていますが,日本も複数の都市で行われ始めましたね.経済的な影響も心配ですが,何よりもまずは生き残ることだと思います.株の格言でも似たようなのがありましたよね.
さて,SARS-CoV-2の研究だけは活発に行われているわけですが,私も例に漏れず今回をきっかけにターゲットとしてどんなもんなの?と勉強を始めたクチです.折角なので,前回までに使用したKNIMEの実行結果も参考に,解析方法を紹介したいと思います.
今回の覚書はこちら.
- 複数の結晶構造が報告されている同一ターゲットについて,Pymol上でグループ化する.
- それらを纏めて重ね合わせる.
追記
- リガンドの結合部位を俯瞰する
今回使用するコマンドの一覧はこちら
- group covalent, enabled
- disable covalent
- group non-covalent, enabled
- disable non-covalent
- group others, enabled
- enable all
- Fetch 2Q6D 6Y2F 2GZ7
- Zoom
- Alignto 2GZ7
- disable !2GZ7
- zoom byres organic around 5
- disable all enable 6Y2F
- disable all
- enable 6Y2F 2GZ7
- set grid_mode, 1
- show surface, byres organic around 5
- set surface_color, gray
- hide surface
- set grid_mode, 0
- disable 6Y2F
- disable 2GZ7
- enable 6Y2F
- enable all
- hide cartoon
今回はついでに,XChemから公開されているFragment ScreeningのPDBファイルも見てみたいと思います.こちらからダウンロード出来るので,covalent, non-covalent, all othersのカテゴリごとにダウンロードしておくといいと思います.SummaryもExcelファイルにまとまってます.構造情報がSMILESだなんて,infomatician friendlyですね!Chemist FriendlyになるようなKNIME WorkFlowも簡単なので紹介すればよかったな...
ファイルの準備が出来たらPymolを開いておきましょう.そしてまずはCovalentのPDBファイルをがっつりDrag&DropでImportしてください.

さすが!なんの処理もなしにタンパクが重ねわせしてあるので,とっても綺麗に見えますね.
オブジェクトパネルに注目すると,こんな感じです.

さて,これをコマンド一発でグループ化しましょう.
group covalent, enabled
こんな感じでグループ化されました.

ちなみにグループ名の左にある+をクリックするとツリーを開閉出来ますが,group covalentとコマンドを入力しても同様にツリーの開閉が出来ます.
disable covalent
disableコマンドでグループの非表示化も出来ます.
次にnon-covalentのPDBファイルをがっつりimportするとこんな感じです.

group non-covalent, enabled
今度は表示されているオフジェクトをgroupコマンドでnon-covalentとしてグループ化します.
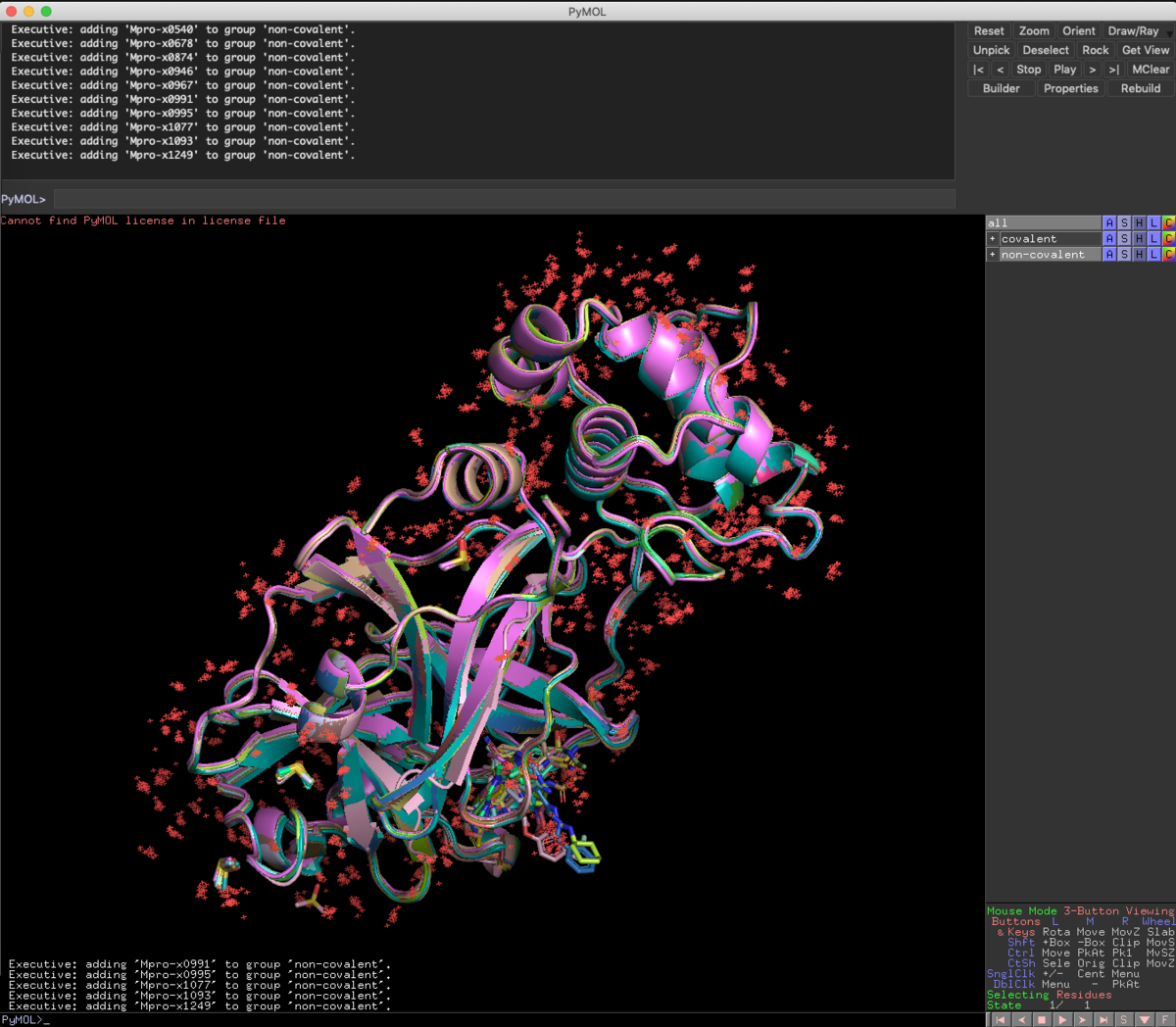
disable non-covalent
先ほどと同様に,一旦全て非表示にします.
続いて,othersのPDBをimportしてきます.

group others, enabled
othersもグループ化しておきます.
enable all
試しに全オブジェクトを表示にすると,全てのPDBファイルが重ね合わせてありますね.ありがたやー.
Fetch 2Q6D 6Y2F 2GZ7
ついでにScienceに報告された共有結合性阻害剤のPDBファイル(6Y2F), 前回紹介したKNIMEの実行結果から見つけた非ペプチドアナログリガンド(2GZ7), 変異プロテアーゼの基質ペプチドとの共結晶構造のPDBファイル(2Q6D)をFetchしてきました.
今回のFetchコマンドのように,複数のファイルやオブジェクトを指定したい時はコンマ(,)は要りません.
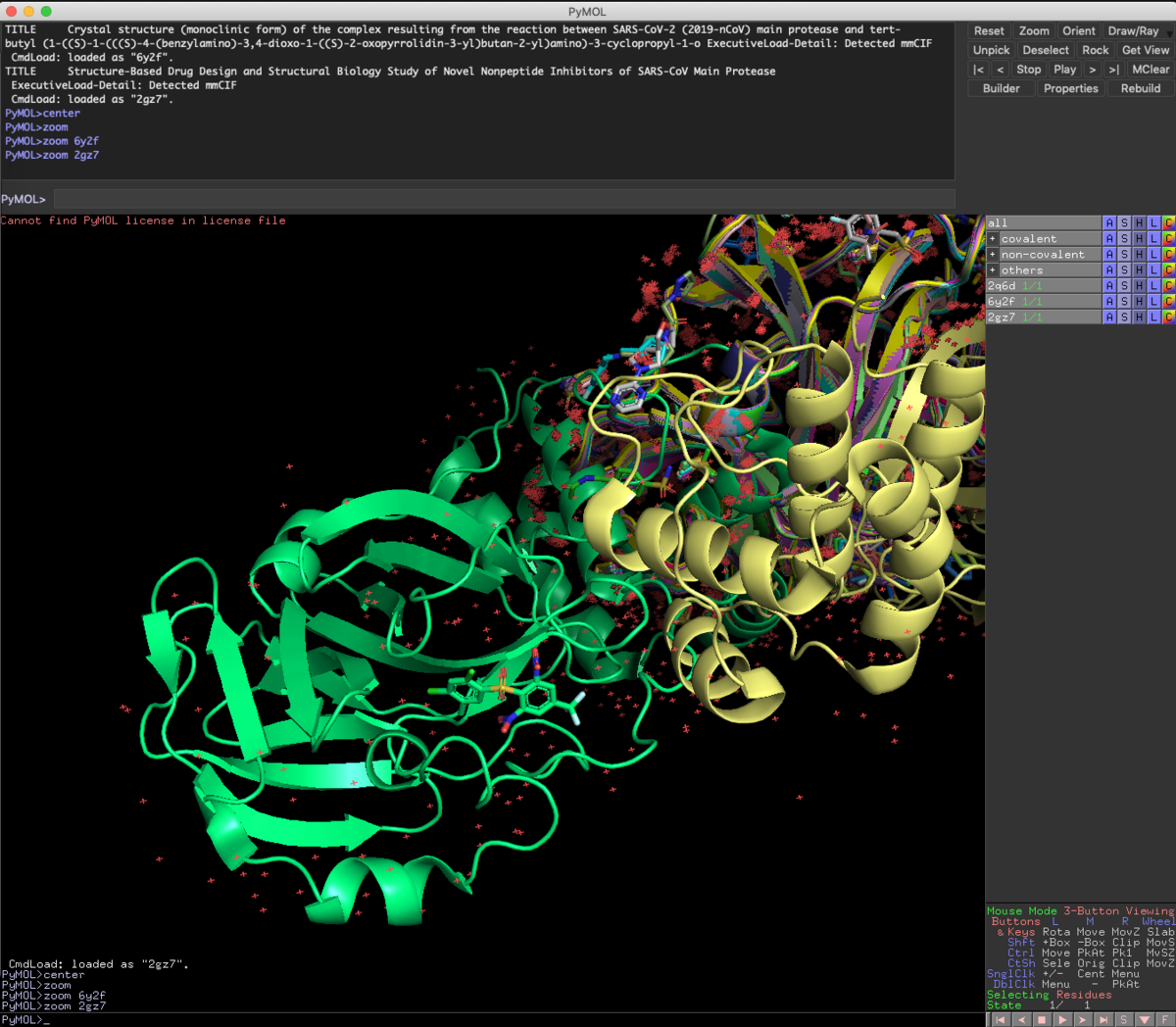
Zoom
オブジェクトを特に指定していないと全体表示されます.
今回Fetchで読み込んだ3つのファイルは,さすがに重ね合わせが必要だとわかりますね.
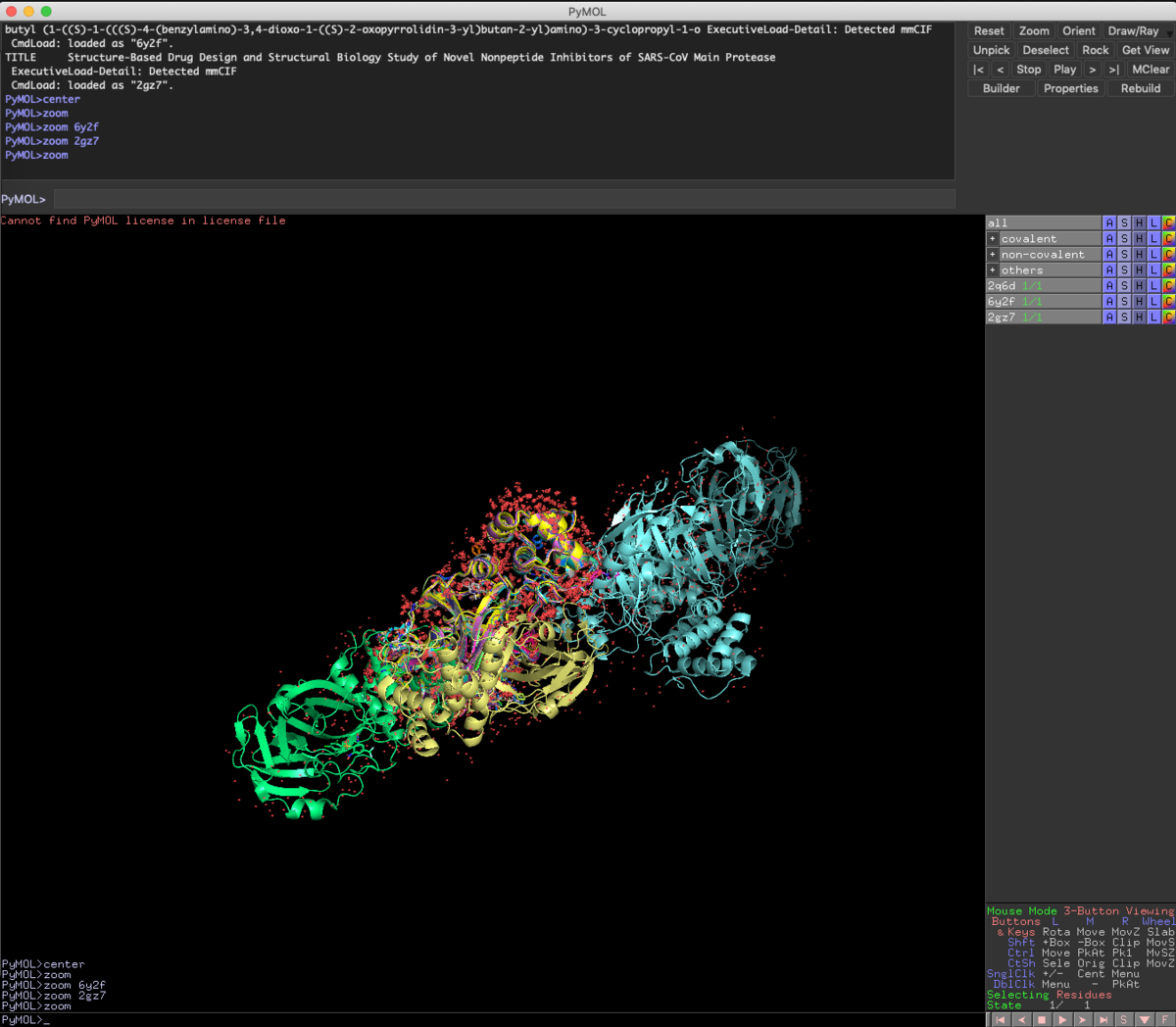
Alignto 2GZ7
Aligntoコマンドは対象のオブジェクト 2GZ7に残り全てのオブジェクトを重ね合わせます.通常はChain Aが重なると思います.オブジェクト数が多いので今回は結構時間かかります...

グループ化と重ね合わせはここまでなので,Pymolのsession fileとして一度保存しとくことをオススメします.
disable !2GZ7
対象の前に!を付与すると,対象が反転します.今回は2GZ7以外を非表示にしました.
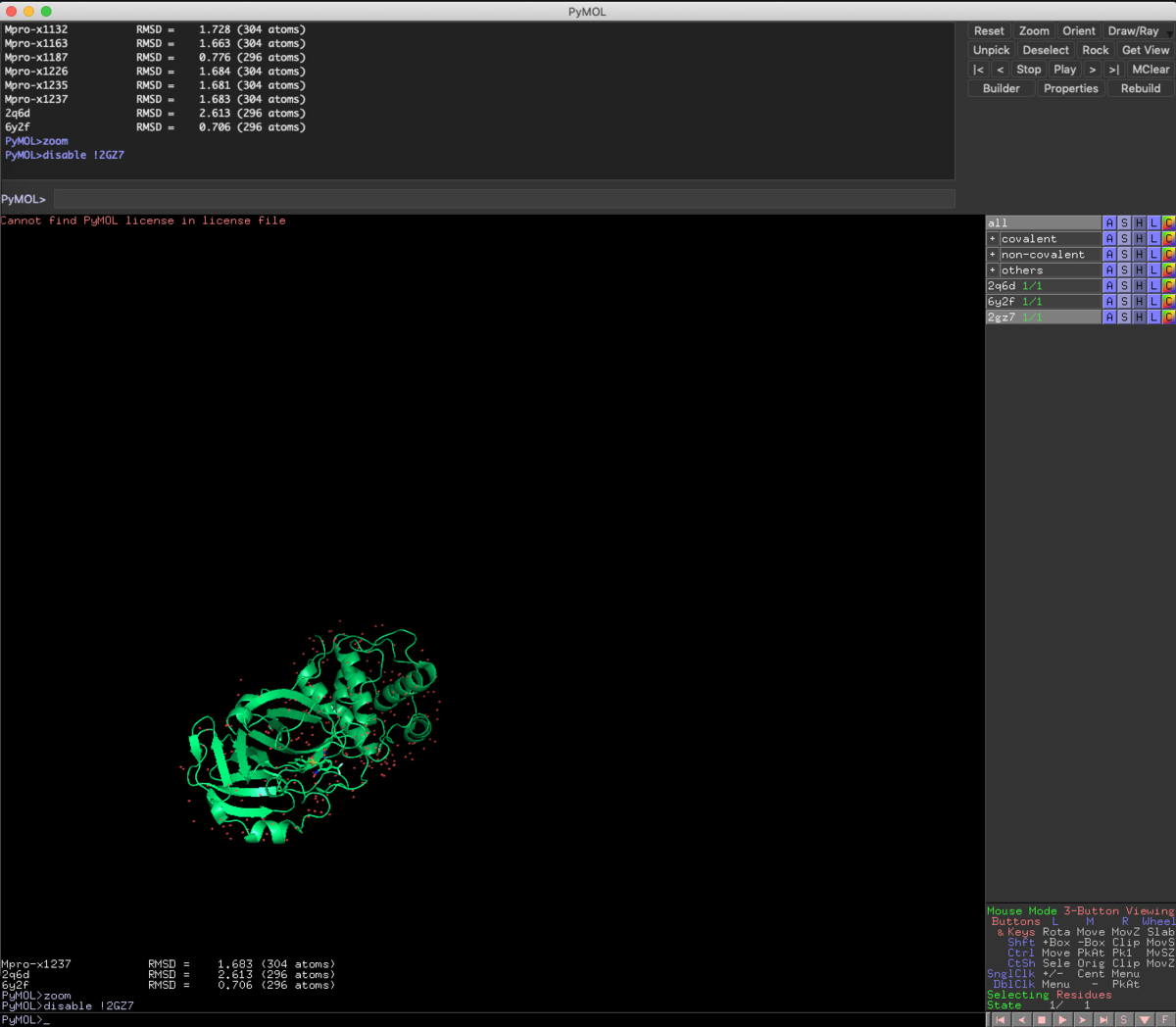
zoom byres organic around 5
Ligandはorganicで指定できるので,周辺5Åにzoomします.なんかちゃんとZoomされてないですね...
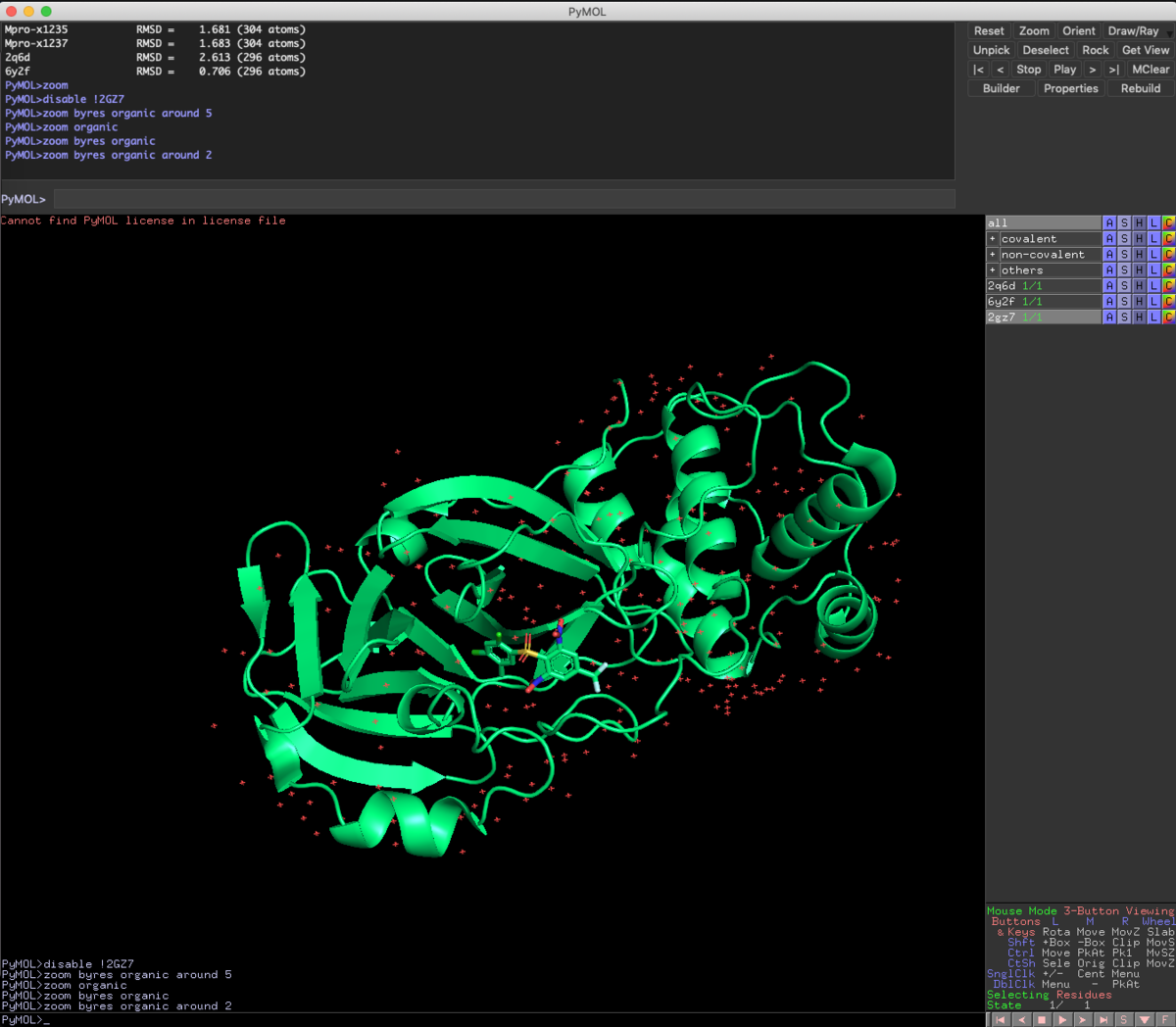
この化合物はRSC PDBに登録されているnon-covalent inhibitorです.
ちょっとLigand周辺の結合様式も見ておきましょう.
preset > ligand site > cartoon
です.
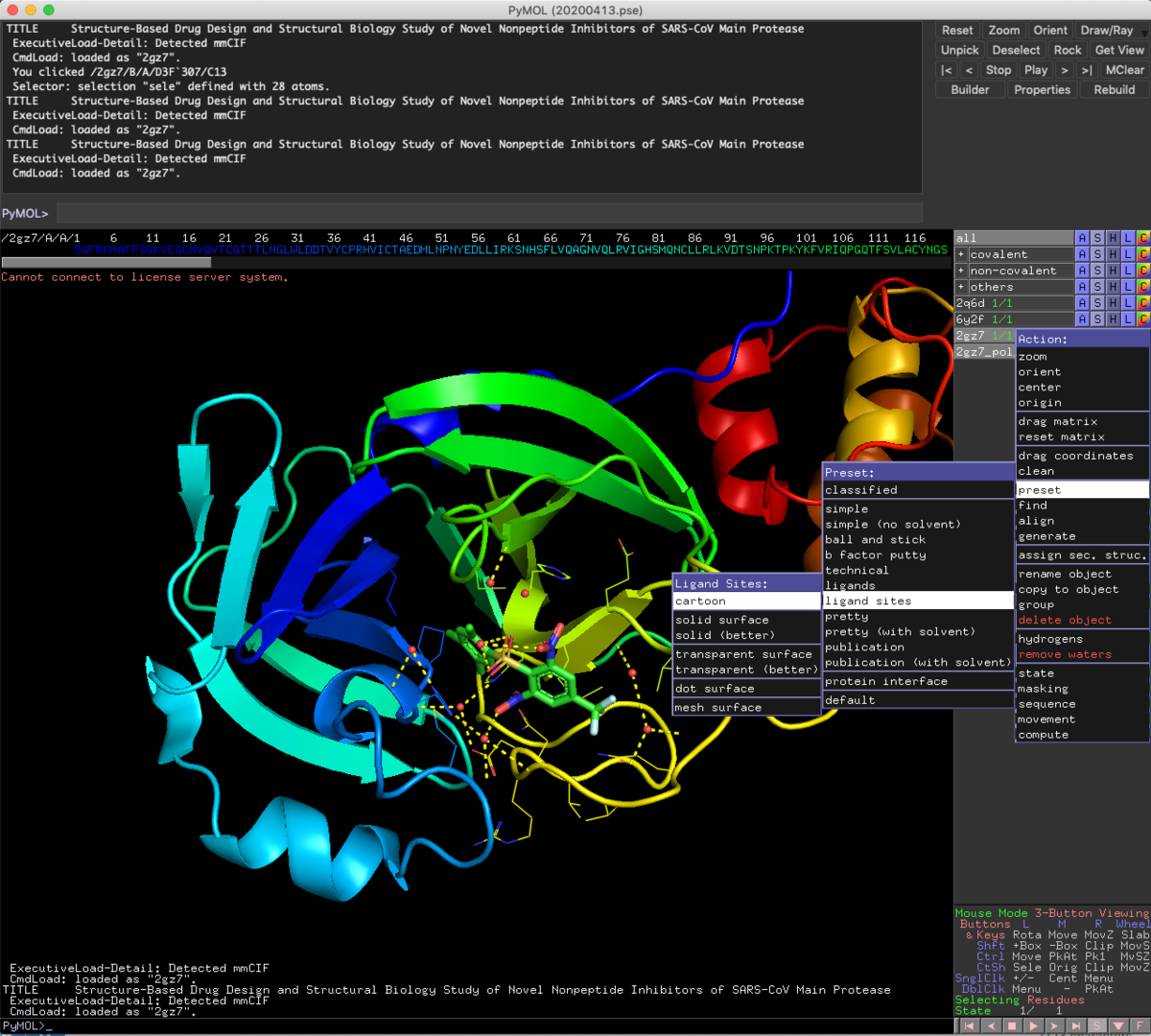
え?
タンパクと相互作用してるの,水を介したニトロ基の一個だけ?
ちょっと他の共結晶とも比較しておきましょう.

disable all
enable 6Y2F
preset > ligand site > cartoon
前回軽く触れましたが,このリガンドは基質ペプチドのミミックなのでバリバリ相互作用してますね.
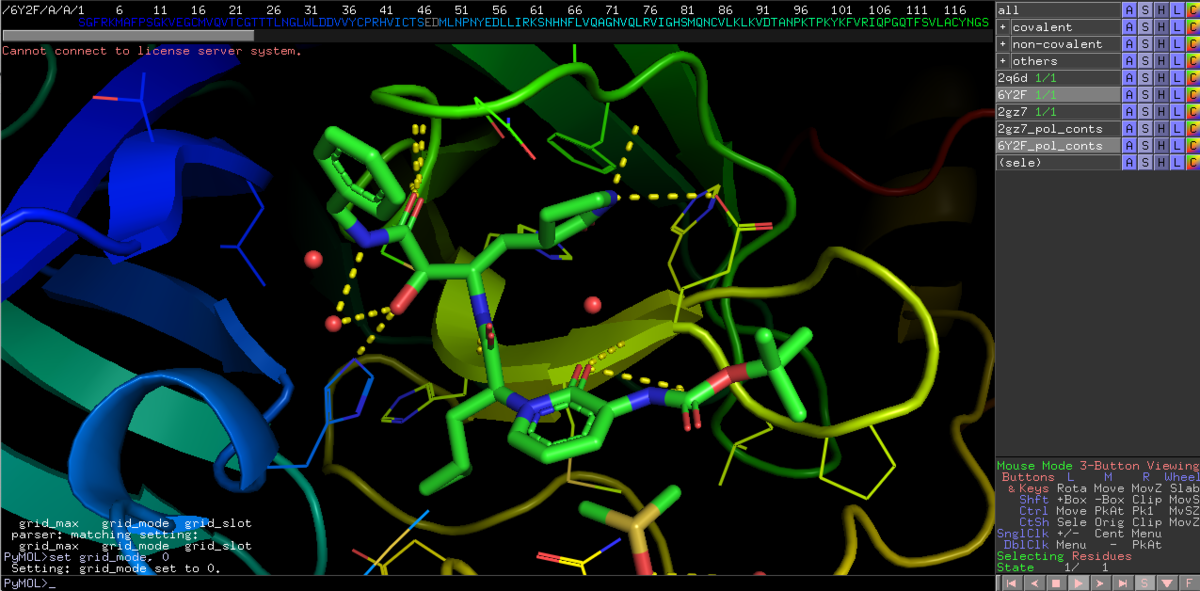
ここからはちょっと端折って行きます.
disable all
enable 6Y2F 2GZ7
set grid_mode, 1
show surface, byres organic around 5
set surface_color, gray
こんな感じに見えるはずです.
6Y2Fのリガンドである13bの赤枠脂溶性ポケットを三置換のPh基が埋めてますが,雰囲気は結構違います.スルホンとPh基の結合角もなんだか捻れてて気持ち悪いな...
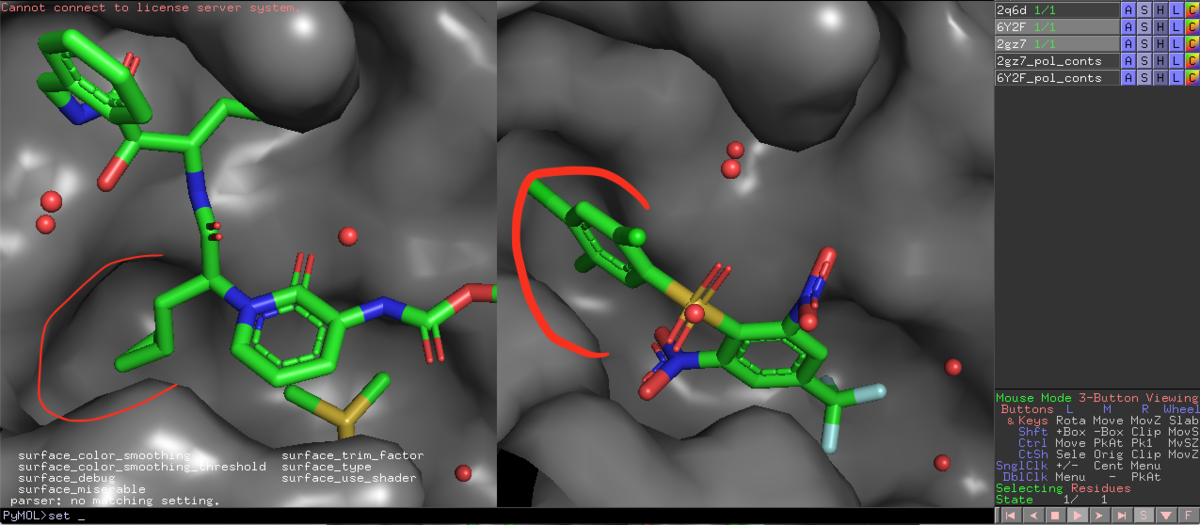
hide surface
脂溶性許容ポケットですが,他に相互作用がなさそうか見ておきましょう.すると...一応ベンゼン環の近くにHis41の側鎖イミダゾールがが平行に配向してますね.左側の6Y2Fと比較しても,フリップしてきてることがわかります.確かこいつは,システインプロテアーゼであるMproの加水分解の活性化を担っていたはずです.
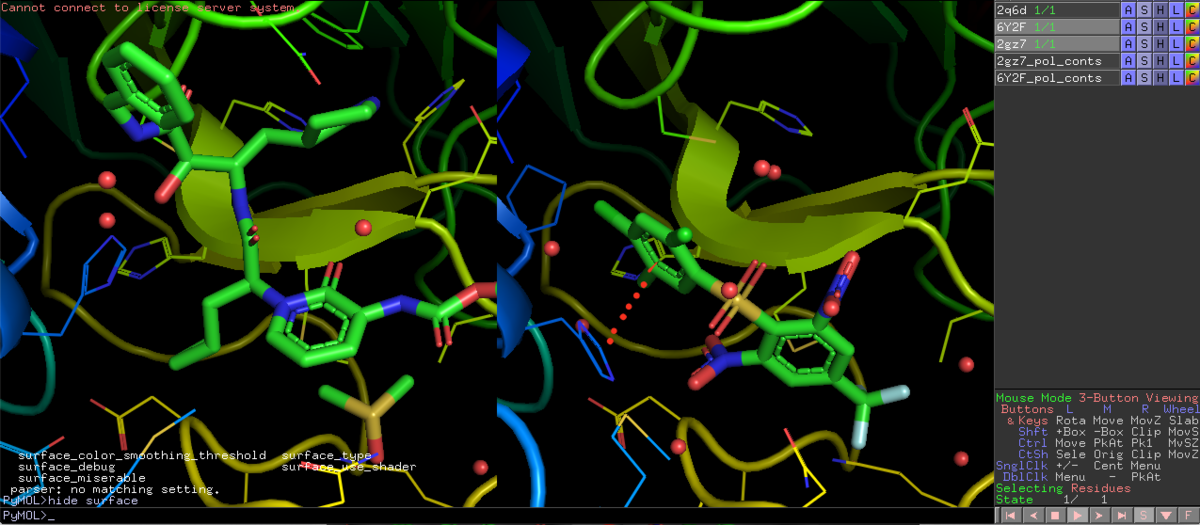
set grid_mode, 0
disable 6Y2F
右下のMouse Modeから3-Button Editingに切り替えて,リガンドと側鎖の距離を見てみましょう.3.8Åなんで,相互作用は有りそうですかね?
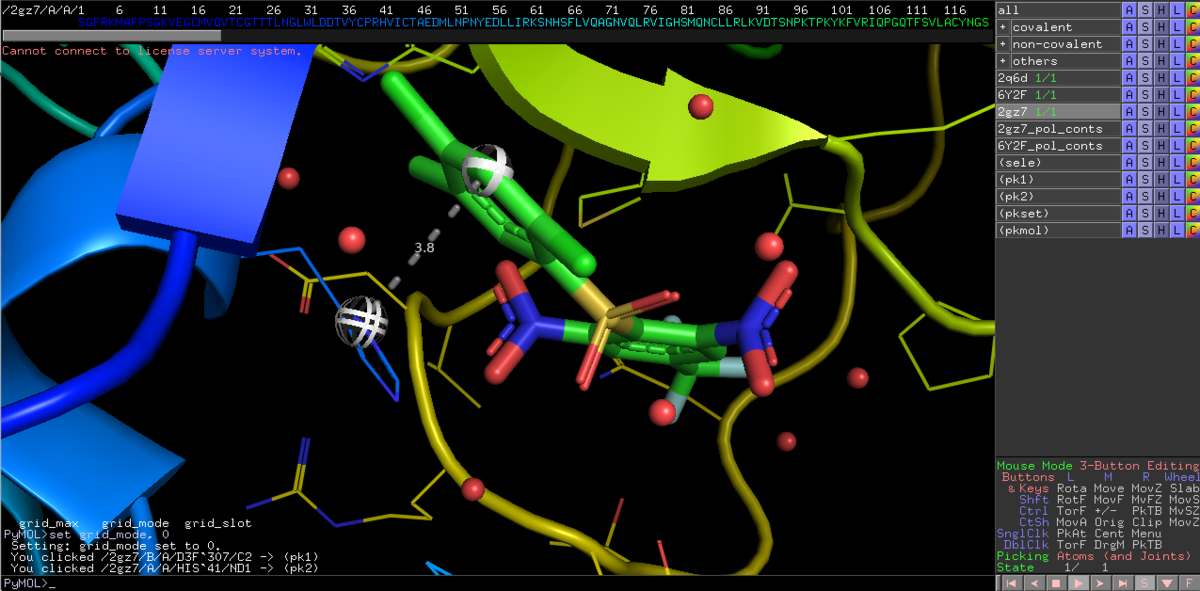
disable 2GZ7
enable 6Y2F
今度は共有結合性ペプチドミミックリガンドと,His41の距離を測ってみましょう.Cys145からの付加反応を受けて,ケトアミドはアルコールとして共結晶が得られています.この立ち上がった水酸基とイミダゾールの距離は2.7Åなので,このHis41が加水分解の触媒として機能しているとみて間違いなさそうです.2GZ7のリガンドは,このHis41を上手く捉えて活性を発現してるってことなんですかね.

ただ,活性値を見ると0.3µMと0.67µMで2GZ7の方が高活性として報告されてるんですよね...ほんまかいな...
共結晶だけでは活性値の推定は中々出来ないので何とも言えませんが,個人的には2GZ7のリガンドの活性は非特異結合か何かが上乗せされてる気がします.
今回のようなプロテアーゼは,基質認識を甘くすることで色んな配列のペプチドに切断出来ることが特徴なんだそうです.つまりここを上手く抑えることが出来れば,virusに必要なタンパクが作られなくなるので効果的と言えますが,一方で色んな基質が結合するなど,選択性の確保も課題になりそうです.上手いこと出来てますね.
enable all
hide cartoon
最後にちょっとだけ,Xchemのフラグメントスクリーニングの結果も見てみましょう.リガンドが集中している黄色で囲った部分がMproが基質を加水分解する部分な訳ですが,othersでgroup化したリガンドが別のところにくっついてます.一つ一つを確認しないと分かりませんが,阻害活性の情報もないですし,あってもアロステリックなので変異が入ったらおしまいです.non-covalentから上手く分子をデザインして,他のシステイプロテアーゼやグルタチオンと選択性を出せれば勝ちってところでしょうか.

今回はこんな感じです.さぁどんな分子をデザインしましょうか?