PDBファイルを検索結果のターゲット分子ごとに割り振ってダウンロードするKNIME WorkFlow
Grüezi,
すっかり投稿するのを忘れてた記事を一つ。
これまで紹介したNodeを使うと、 PDB ConnetcorのReport機能を使ってターゲット分子ごとにPDBを割り振ってダウンロードすることも簡単にできます。
今回はスクショメモしか残してなかったのでかなりざっくりですが、 内容はこんな感じです。
WorkFlow 全体
今回新たに紹介する nodeは多分ありません。上段にはいつも通りリガンドの画像付きExcelにまとめレポートを、下段にはターゲット分子ごとにPDBを振り分けるフローを示してます。

レポートはこんな感じでExportされます。ごめんなさい今回のキーワードは何にしたか忘れました。。。

Table Row To Variant
レポート中のRowを変数に設定します。今回はMacromolecule Nameを選択することで、Excel上ではシートに、PDBファイルはフォルダにそれぞれ割り振りできます。

String Manipulation (Variable)
このNodeを使うことで変数をStringに組み込めます。PDBファイルをダウンロードしたいDirectory名を変数を使って生成します。

Excel Sheet Appender
これもいつも通りSheet nameを変数設定することで、ターゲット分子名ごとにシートに情報を振り分けます。

Export結果
以下、こんな感じです。ソースが違っても分子名が同じであれば同じDirectoryに保管されます。

今回は自分用のWorkFlowの覚書でした。
あ、そういえば日本に帰ってきました。
苦労話はしても疲れるだけなのでしませんが、あーあ帰ってきちゃったなって感じです。
もう少しMacBookAir2012Midで頑張ることになったけど、勉強続くかな…
Dear Pymol Beginners x KNIME 1.4 -リガンドをクラスタリングして,レポートとPDBファイルのダウンロード先の振り分けに使ってみたり-
Grüezi,
早いもので,帰国まで1ヶ月を切ってしまいました.慣れてしまえばもう少し滞在したいような,でも物価の高さには相変わらず目眩もするし,正直多分虫歯が出来たっぽいしで帰国したいような,そんなどっち付かずの心持ちです.計画してた旅行の残りはコロナウィルスのせいでオールキャンセルなので,やっぱり心残りだなぁ.
さて,最近もっぱらVirtual Screeningなんかをやってるんですが,Libraryが大きくてなかなか終わりません.という訳で,KNIMEを使って合間に出来るPDBの情報収集方法の備忘録です.
今回はざっくりこんな感じのことが出来ます.
- RSC PDB Bankのサイトから取得したPDBの情報からリガンドをClusteringする
- Clusterごとの情報をExcelにExportする
- PDBをClusterごとにフォルダーにダウンロードする
今回紹介するNodeはこちら
- Workflowの全体像
- PDB connector
- Row Filter
- Fingerprints
- Distance Matrix Calculate
- k-Medolds
- Group Loop Start
- Value Counter
- TableRow To Variable
- String Manipulation (Variable)
- Excel Sheet Appender
- Variable Loop End
- Create Directory
- String Manipulation (Variable)
- PDB Saver
- Loop End
- Excel Reportからリガンドの確認
今回も,す(@sumtat_)さんのところのブログを大いに参考に(パクって)しています.いつも勉強になりますありがとうございます.
そんな訳で,今回も行ってみましょう.
Workflowの全体像
Workflowの左側でPDB情報を取得,Filtering,Clusteringを行なっています.分岐した上段では,クラスターごとに情報をグループ化してExcelにシートごとにExportしています.下段では,クラスターごとにDirectoryを作ってPDBファイルをダウンローとしています.
青で囲ったClusteringの部分が今回の肝ですが,思いっきりパクって使わせてもらってます.緑で囲った部分はClusterの命名に便利だなと思ったので採用(つまりパクリ)しましたが,他にいいアイディアがあれば代用していいと思います.つまり何が言いたいかというと,詳細な説明は先達に譲ります(!).

PDB connector
今回はIDs and KeywordsタブのText Searchに"SARS-CoV-2 main protease"と入力しています.Report OptionsはStructure Summary, Ligands, Binding Affinity, Primary Citationをチェックしています.
Row Filter
MW<100のリガンドをFilter Outしています.目的としては溶媒や添加剤,apo体を除くことです.なので,分子量をもう少し大きくしてもいいかもです.

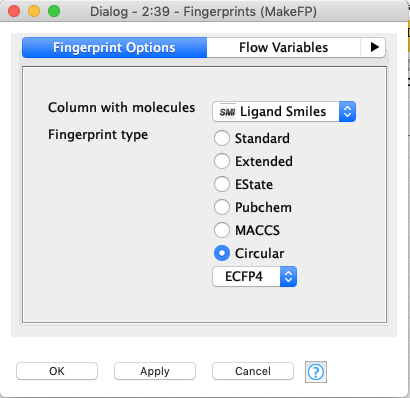
Fingerprints
LigandのいわゆるFingerprintを作ってくれます.設定はパクったので,ぶっちゃけ詳細は説明できません...
Distance Matrix Calculate
同じく詳細は割愛します.
ざっくり言うと,前項で作ったFingerprintの距離を総当たりで計算して吐き出す,みたいなNodeらしいです.かの有名なTanimoto法でお願いします.

k-Medolds
このNodeでClusteringしてくれます.Partition Countで発生させるクラスターの数を選べます.今回はまんまパクって5個にしてます.

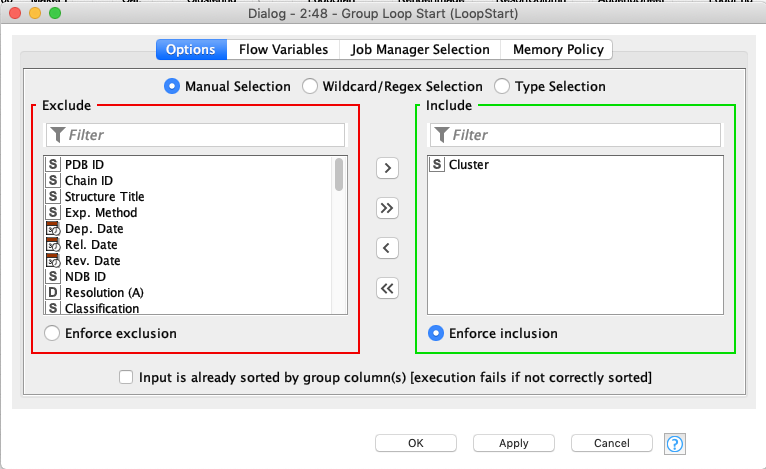
Group Loop Start
発生させたClusterを元にGroup化を繰り返すLoop Nodeです.上段も下段も設定は同じです.
Value Counter
Group化したClusterの中にあるLigandの数を数えます.

TableRow To Variable
Countした数を変数に変換します.特に設定不要です.
String Manipulation (Variable)
前項で発生させた変数を受けてStringを発生させます.今回は数えた数をそのままExcelのシート名にします.はい,まんまパクってます.
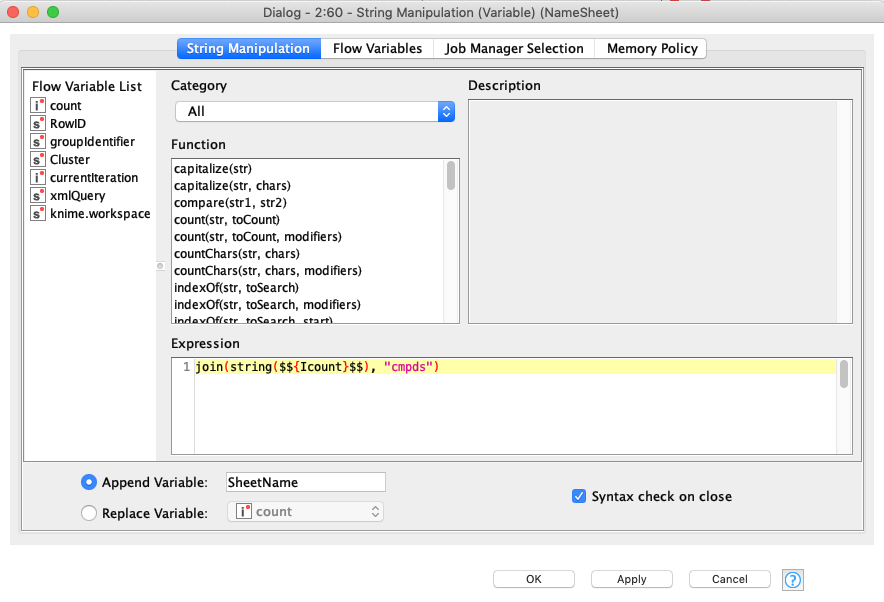
Excel Sheet Appender
ExcelにPDB情報をシートごとにまとめます.File名は自由に設定して,シート名を前項までに作った変数にします.add column headersにチェックしておいた方がいいです.

Variable Loop End
Loopの終わりを指定してあげます.
Create Directory
ClusterごとにPDBファイルを振り分けるDirectory(フォルダ)を作りたいのですが,ちょっとTrickyです.
Locationは前述のExcel Reportと同じ場所を指定しておきます.フォルダ名に変数を指定したいのですが,なぜかここに変数指定ボタンがないので,適当なダミー名(今回はa)を入力した上でFlow Variableのタブから改めて変数を指定します.
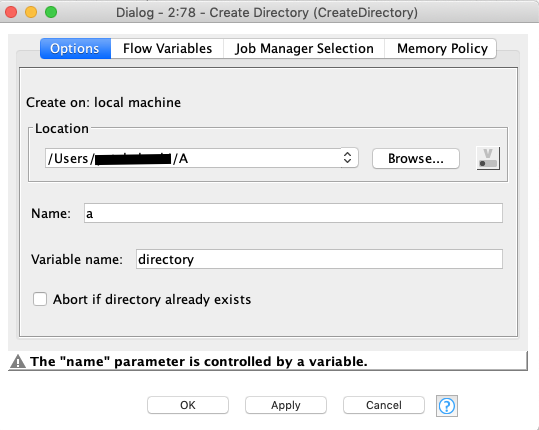
Directory名をTable To Variableで発生させたカラム名に指定します.

実行結果はこんな感じになればOKです.directory名に指定した変数(今回はクラスター内のLigandの数+cmpds)が反映されています.

String Manipulation (Variable)
前項で発生させたdirectoryのValueをPDBの保存先に指定できればよかったのですが,残念ながら"file:"という文字列が先頭に付いてるのでPDB Saverの保存先に使えません.そこで,いつものString Manipulationで保存先を発生させます.Sheetnameを変数に使えばOKです.

PDB Saver
保存先として,Parent Directoryの変数に先ほど発生させたdirectoryを指定します.

保存に成功すると,実行結果の右端にtrueと表示されます.何かしらエラーがあった場合はFailsになります.
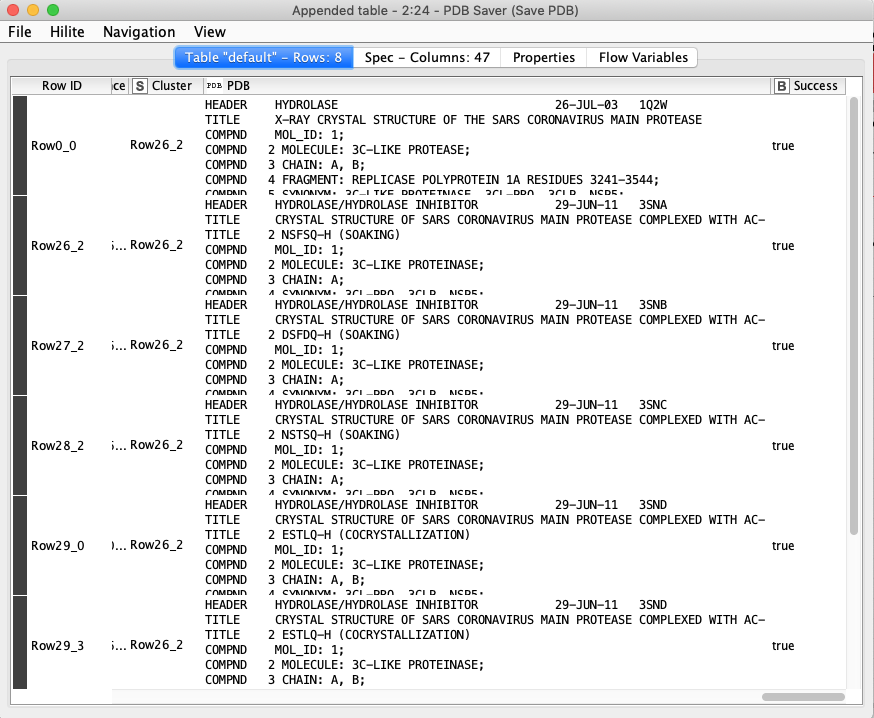
Loop End
Loop処理の終了を宣言します.
実行結果
2020/04/21現在で,実行結果はこんな感じになります.ちゃんと5つのClusterに分かれてPDBファイルが保存されました.あれ?11化合物あるはずなのにファイルは8つ?これは一つのファイルの中に,例えばA鎖とB鎖がある場合,リガンド数が2個とカウントされるためです.実際には一つのPDBファイルの中に格納されています.

PDB Saverの実行結果を見ると納得かと思います.
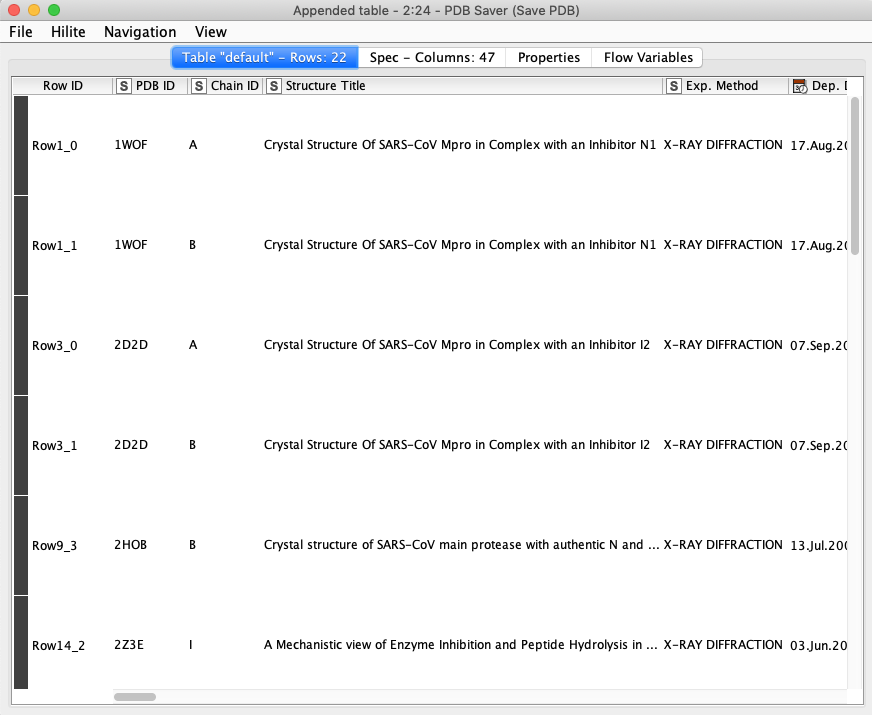
Excel Reportからリガンドの確認
さて,Excelを開いてみましょう.
ちゃんと5つのClusterごとにシートが作られてますね.数が一番多い60cmpdsのシートには,先日紹介したX-Chemのフラグメントスクリーニングの結果が含まれています.ExcelのFilter機能を使って絞り込むと,他にもリガンドが見つかりますね.前々回の記事で紹介したリガンドは,こんな感じで見つけてきました.

ついでに22cmpdsのシートも開いてみると,ここにはペプチドミミックのリガンドがまとまってました.中身がさっぱりわかってなくても,Clusteringが結構うまくいってるんだなぁと関心です.
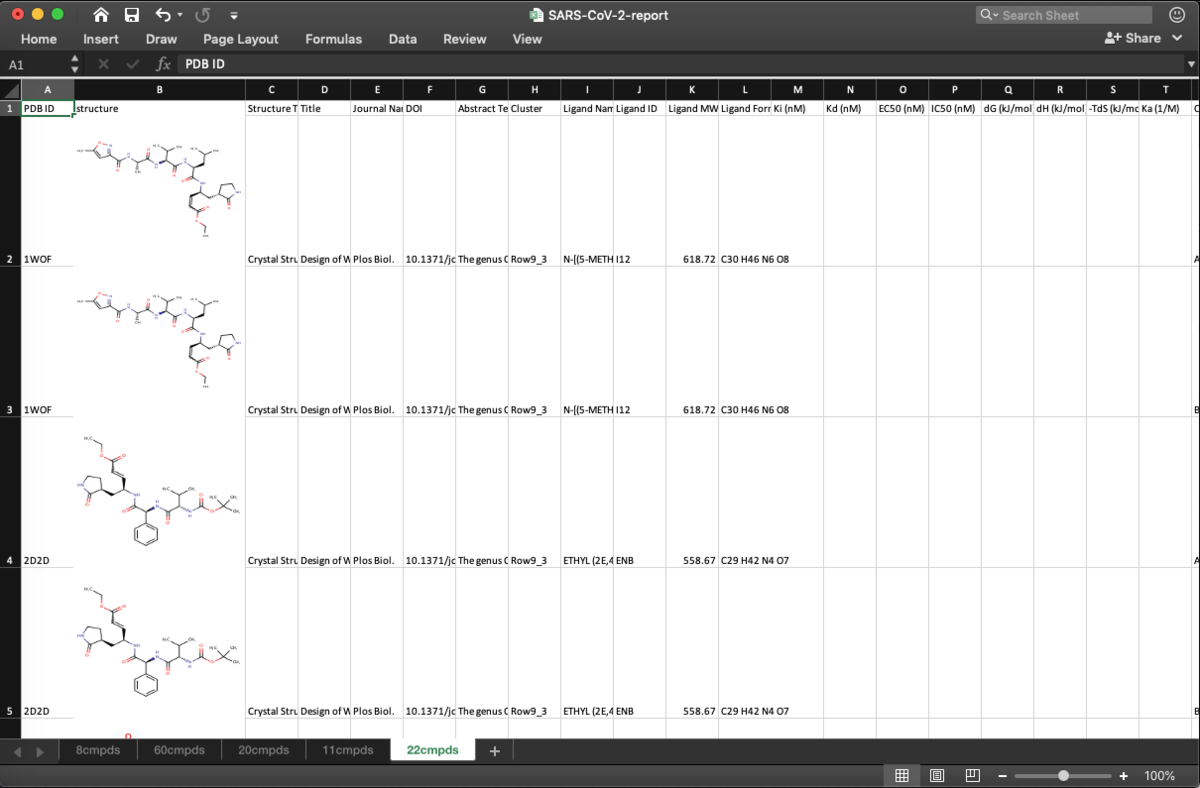
こんな感じでClusterごとに振り分けたPDBファイルを必要な分だけPymolに読み込んでGroup化してやると,プロジェクト初期には有用だったりしないかなと思います.
今回みたいに取得した情報の振り分けを自動化出来ると,特許解析とかに応用出来ないのかなとか妄想中です.
ご参考までに.
追記)後から気が付いたんですが,Creat DirectoryとかしなくてもString Manipulationで保存Directoryを指定してやれば勝手にDirectoryが作成されました.まぁ,Variableの練習をしたってことでご愛嬌.
Dear Pymol Beginner 5 -Pymolのデフォルトを変更してみたり-
Grüezi,
何てことない話なんですが,Pymolのデフォルトセッティングを紹介します.
cd /tmp
set sphere_scale, 0.2
set cartoon_transparency, 0.5
set transparency, 0.6
set surface_color, white
File > Edit pymolrcを開いて上記のTextをコピペ,閉じる時にSaveするだけです.
以下,具体的に比較するとこんな感じです.左がpymolrcなし,右がありです.
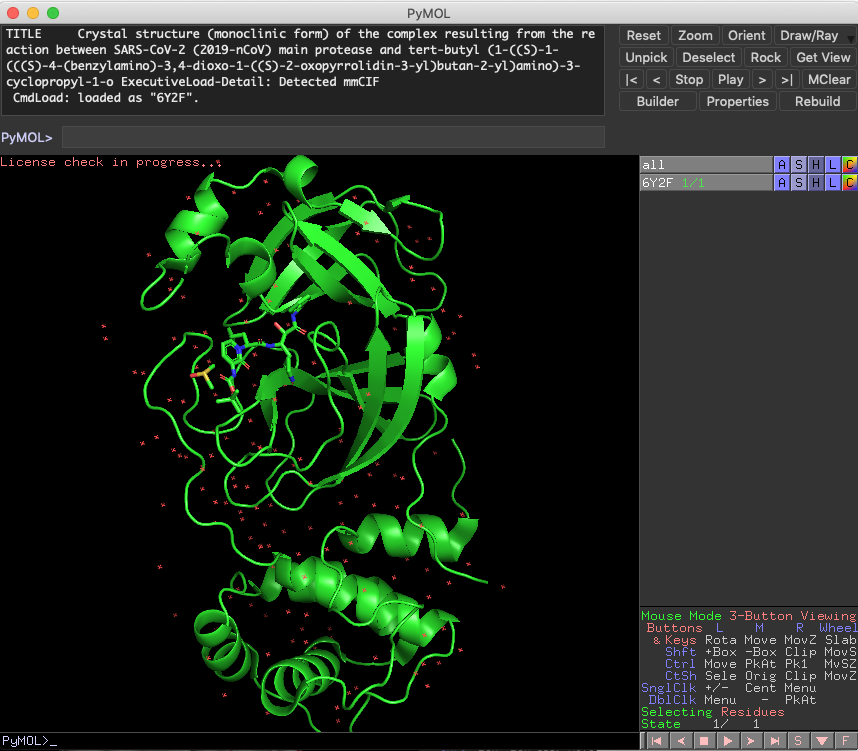
show surfaceすると思いっきり見た目は違います.左は毒々しいですね...
ポケット周辺も具合はだいぶ違います.

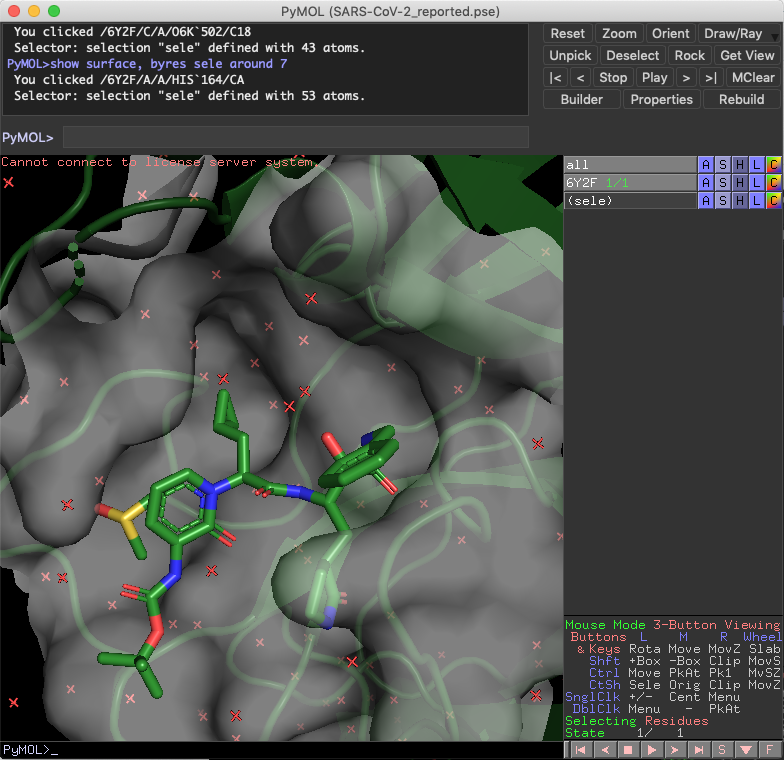
ポケット周辺のアミノ酸残基をshow linesしておくと,相互作用が取れそうな残基が見えるかもしれません.影があった方が隙間が見えて変換の余地を把握出来そうな気がします.
最後にcd /tmp ですが,fetchなどで取ってたきたcif fileの保存先を一時フォルダに変更しています.何もしないとWorking Directoryはユーザー直下になっていて,ふとした時にmessyになってることに気がつきます.
ご参考までに
Dear Pymol Beginners 4 -オブジェクトをグループ化してみたり,リガンドの結合位置や様式を比べてみたり-
Grüezi,
世界中でロックダウンが行われていますが,日本も複数の都市で行われ始めましたね.経済的な影響も心配ですが,何よりもまずは生き残ることだと思います.株の格言でも似たようなのがありましたよね.
さて,SARS-CoV-2の研究だけは活発に行われているわけですが,私も例に漏れず今回をきっかけにターゲットとしてどんなもんなの?と勉強を始めたクチです.折角なので,前回までに使用したKNIMEの実行結果も参考に,解析方法を紹介したいと思います.
今回の覚書はこちら.
- 複数の結晶構造が報告されている同一ターゲットについて,Pymol上でグループ化する.
- それらを纏めて重ね合わせる.
追記
- リガンドの結合部位を俯瞰する
今回使用するコマンドの一覧はこちら
- group covalent, enabled
- disable covalent
- group non-covalent, enabled
- disable non-covalent
- group others, enabled
- enable all
- Fetch 2Q6D 6Y2F 2GZ7
- Zoom
- Alignto 2GZ7
- disable !2GZ7
- zoom byres organic around 5
- disable all enable 6Y2F
- disable all
- enable 6Y2F 2GZ7
- set grid_mode, 1
- show surface, byres organic around 5
- set surface_color, gray
- hide surface
- set grid_mode, 0
- disable 6Y2F
- disable 2GZ7
- enable 6Y2F
- enable all
- hide cartoon
今回はついでに,XChemから公開されているFragment ScreeningのPDBファイルも見てみたいと思います.こちらからダウンロード出来るので,covalent, non-covalent, all othersのカテゴリごとにダウンロードしておくといいと思います.SummaryもExcelファイルにまとまってます.構造情報がSMILESだなんて,infomatician friendlyですね!Chemist FriendlyになるようなKNIME WorkFlowも簡単なので紹介すればよかったな...
ファイルの準備が出来たらPymolを開いておきましょう.そしてまずはCovalentのPDBファイルをがっつりDrag&DropでImportしてください.

さすが!なんの処理もなしにタンパクが重ねわせしてあるので,とっても綺麗に見えますね.
オブジェクトパネルに注目すると,こんな感じです.

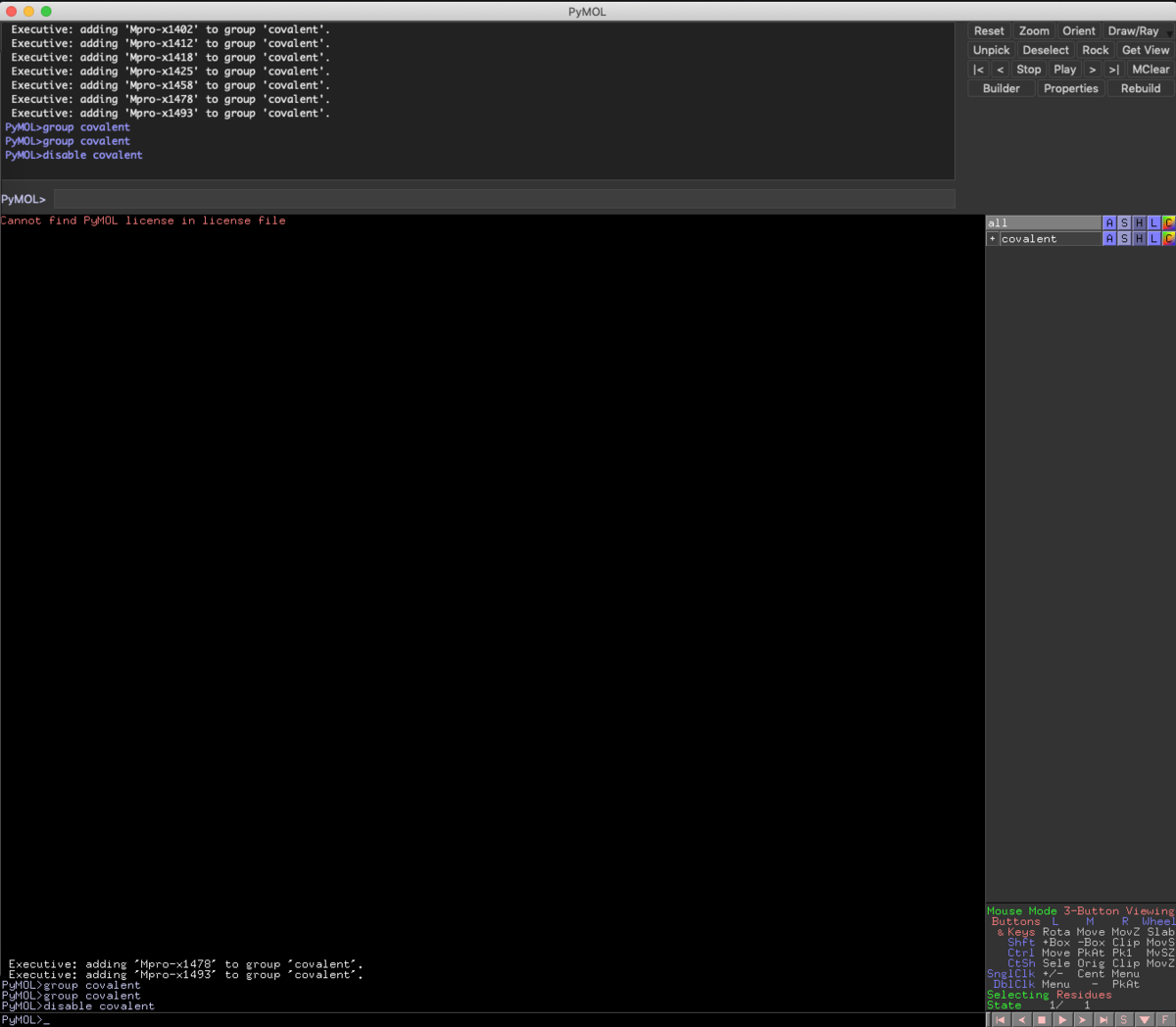
さて,これをコマンド一発でグループ化しましょう.
group covalent, enabled
こんな感じでグループ化されました.

ちなみにグループ名の左にある+をクリックするとツリーを開閉出来ますが,group covalentとコマンドを入力しても同様にツリーの開閉が出来ます.
disable covalent
disableコマンドでグループの非表示化も出来ます.
次にnon-covalentのPDBファイルをがっつりimportするとこんな感じです.

group non-covalent, enabled
今度は表示されているオフジェクトをgroupコマンドでnon-covalentとしてグループ化します.
disable non-covalent
先ほどと同様に,一旦全て非表示にします.
続いて,othersのPDBをimportしてきます.

group others, enabled
othersもグループ化しておきます.
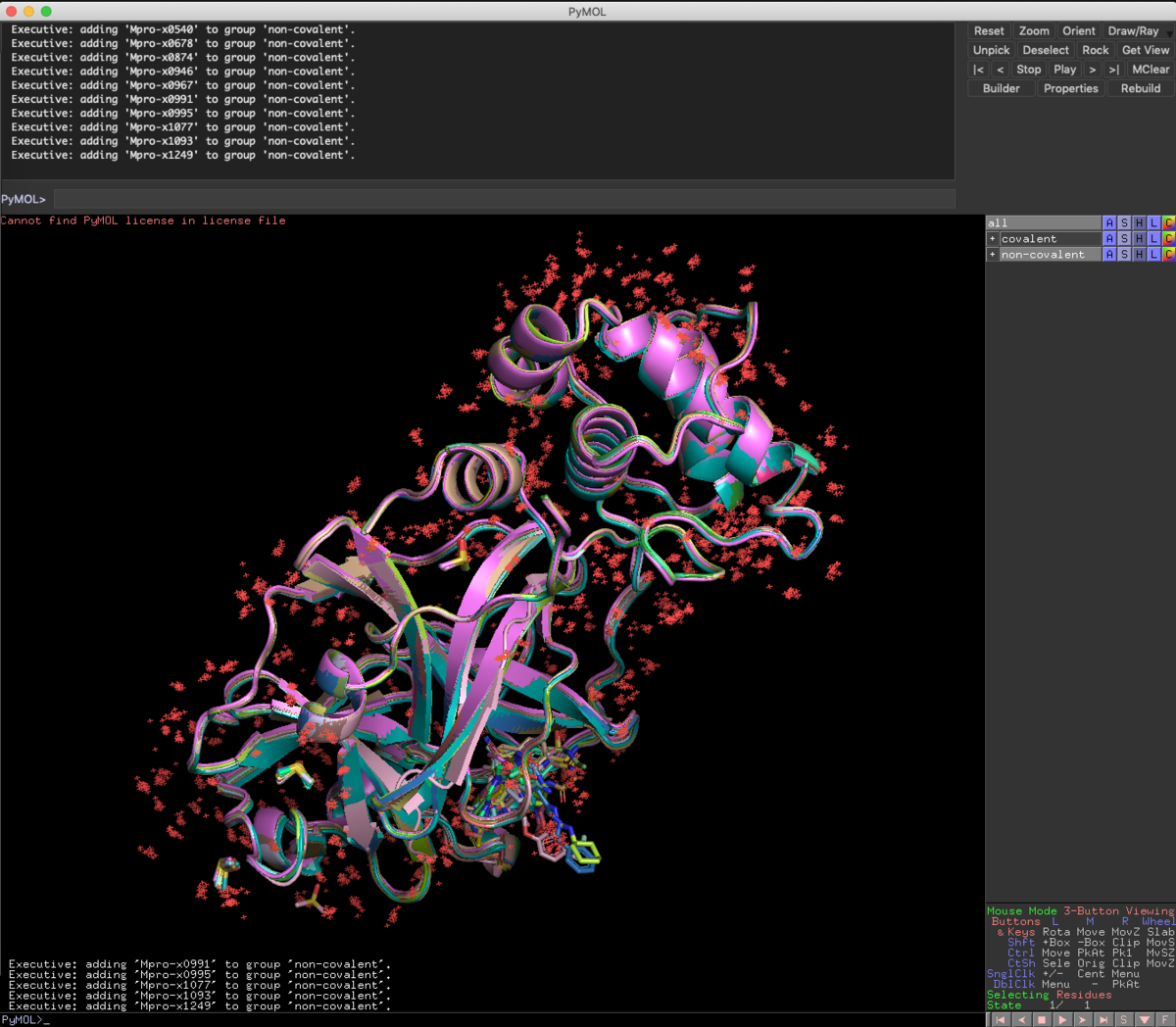
enable all
試しに全オブジェクトを表示にすると,全てのPDBファイルが重ね合わせてありますね.ありがたやー.
Fetch 2Q6D 6Y2F 2GZ7
ついでにScienceに報告された共有結合性阻害剤のPDBファイル(6Y2F), 前回紹介したKNIMEの実行結果から見つけた非ペプチドアナログリガンド(2GZ7), 変異プロテアーゼの基質ペプチドとの共結晶構造のPDBファイル(2Q6D)をFetchしてきました.
今回のFetchコマンドのように,複数のファイルやオブジェクトを指定したい時はコンマ(,)は要りません.
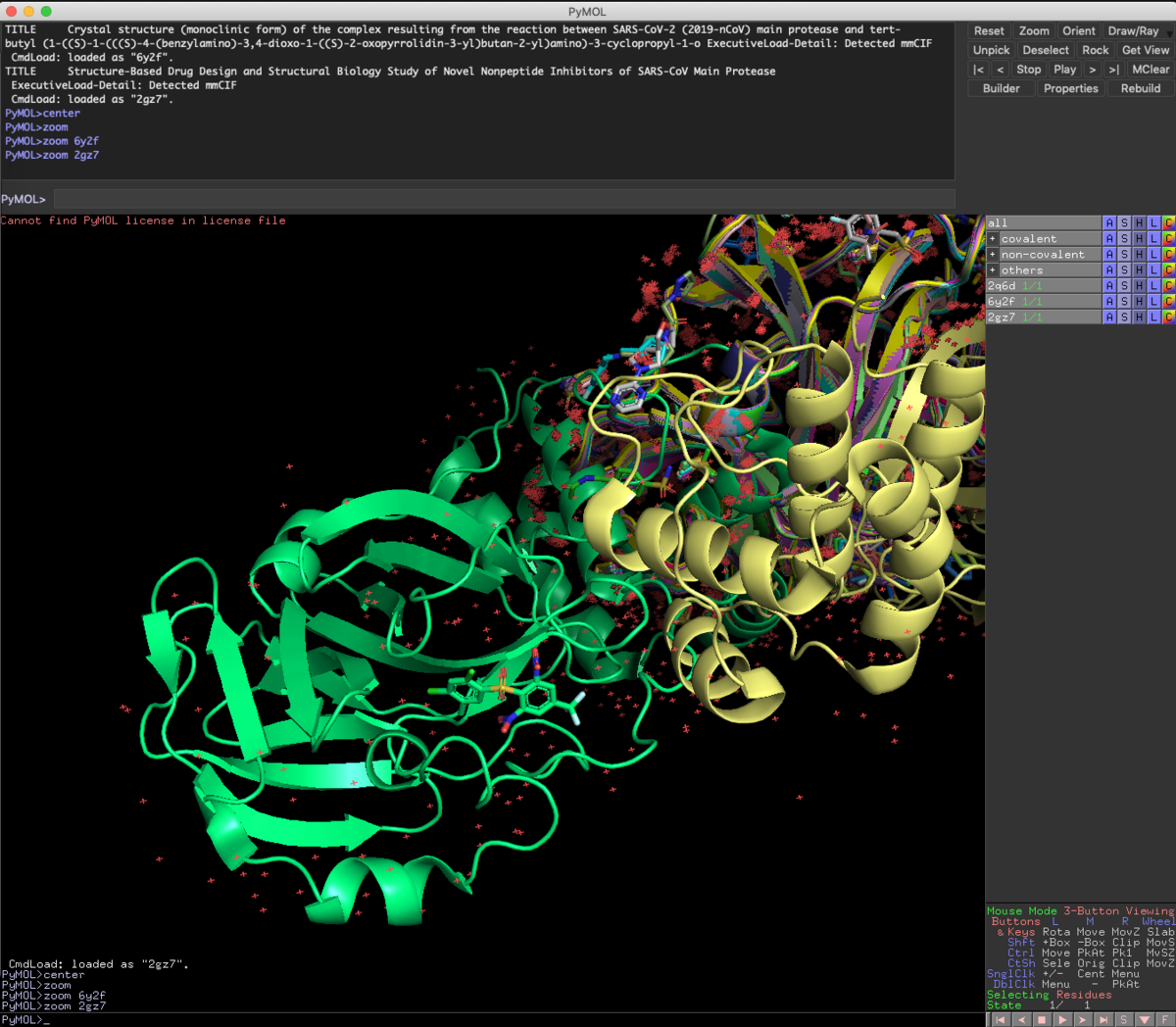
Zoom
オブジェクトを特に指定していないと全体表示されます.
今回Fetchで読み込んだ3つのファイルは,さすがに重ね合わせが必要だとわかりますね.
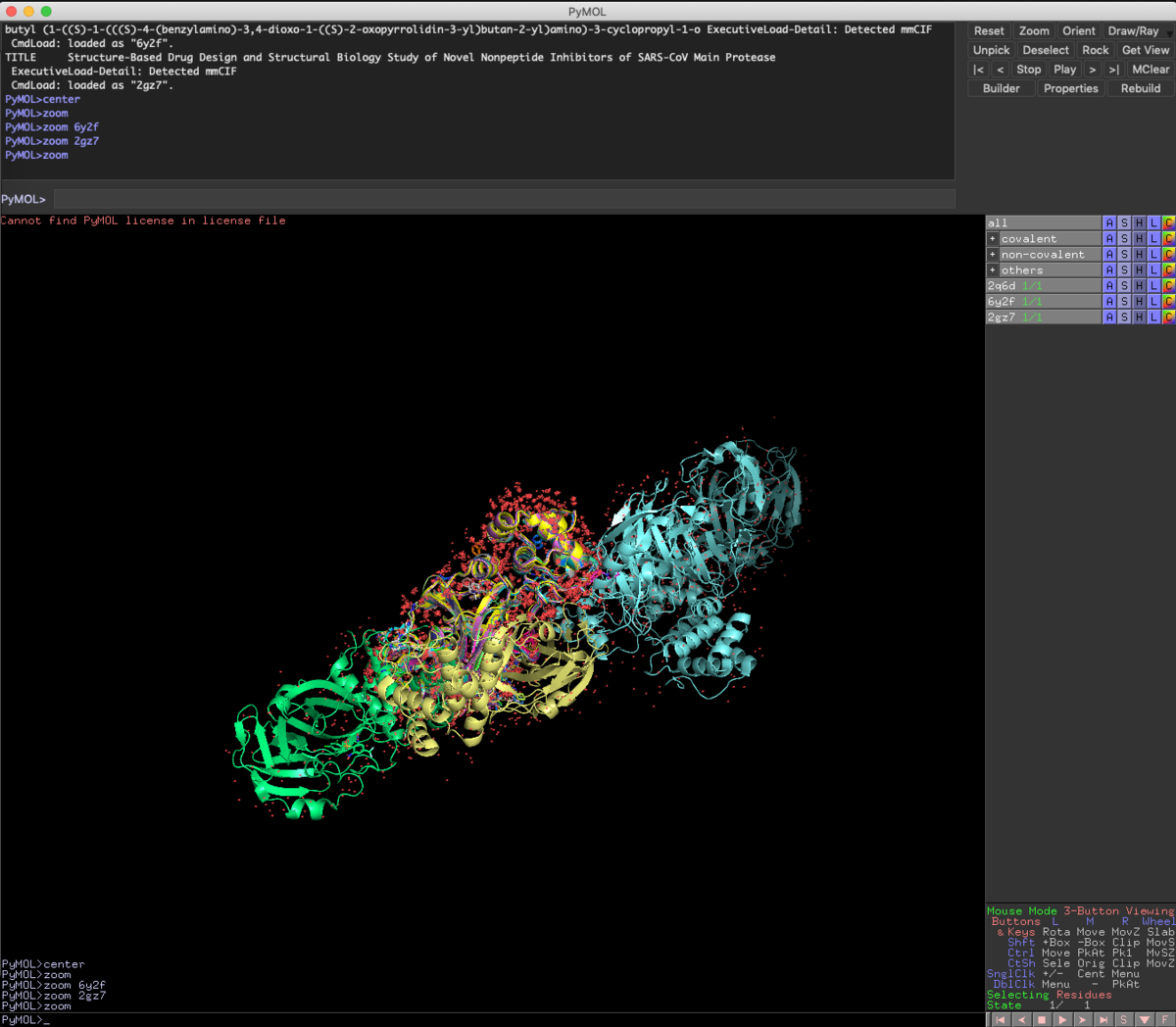
Alignto 2GZ7
Aligntoコマンドは対象のオブジェクト 2GZ7に残り全てのオブジェクトを重ね合わせます.通常はChain Aが重なると思います.オブジェクト数が多いので今回は結構時間かかります...

グループ化と重ね合わせはここまでなので,Pymolのsession fileとして一度保存しとくことをオススメします.
disable !2GZ7
対象の前に!を付与すると,対象が反転します.今回は2GZ7以外を非表示にしました.
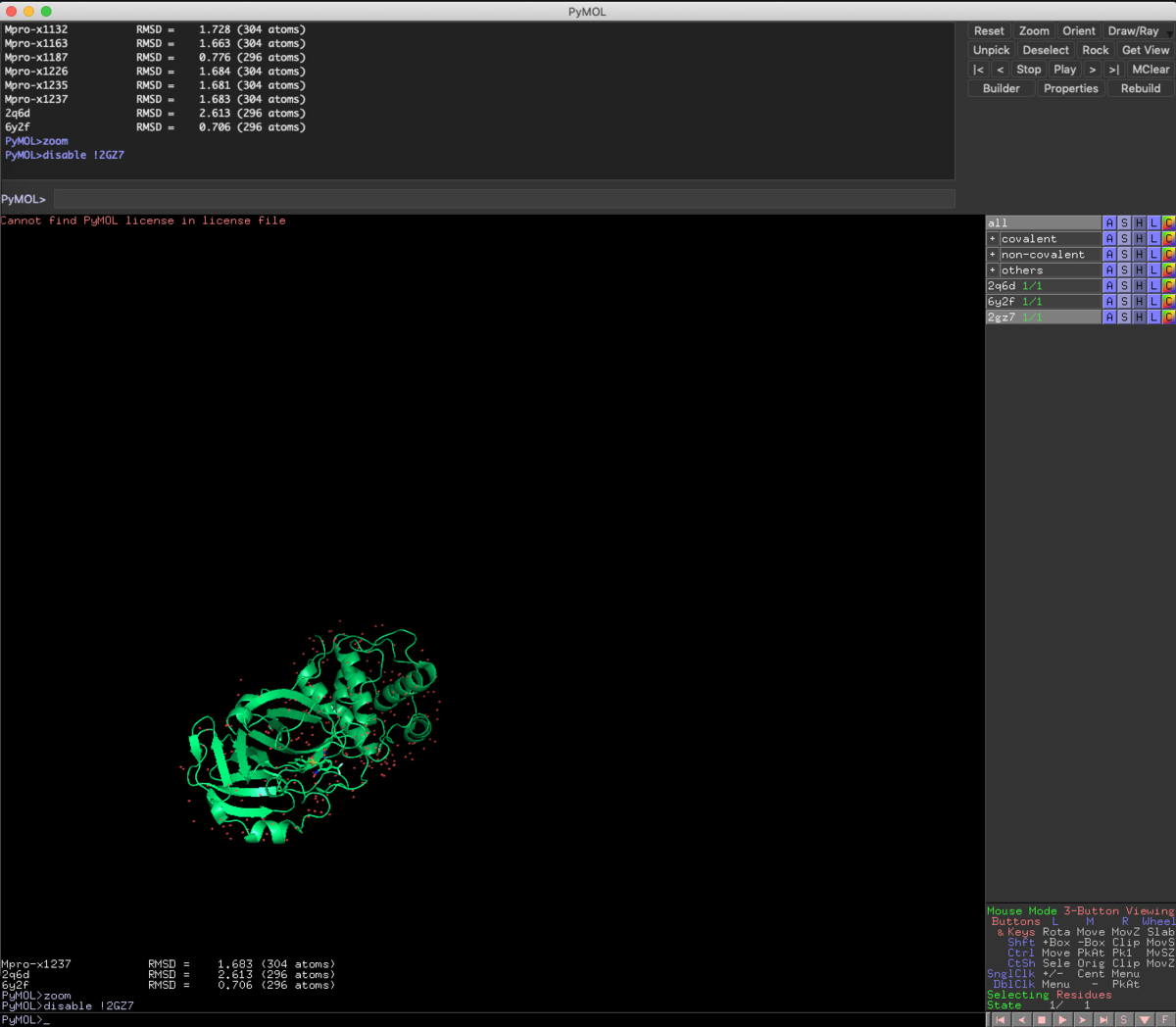
zoom byres organic around 5
Ligandはorganicで指定できるので,周辺5Åにzoomします.なんかちゃんとZoomされてないですね...
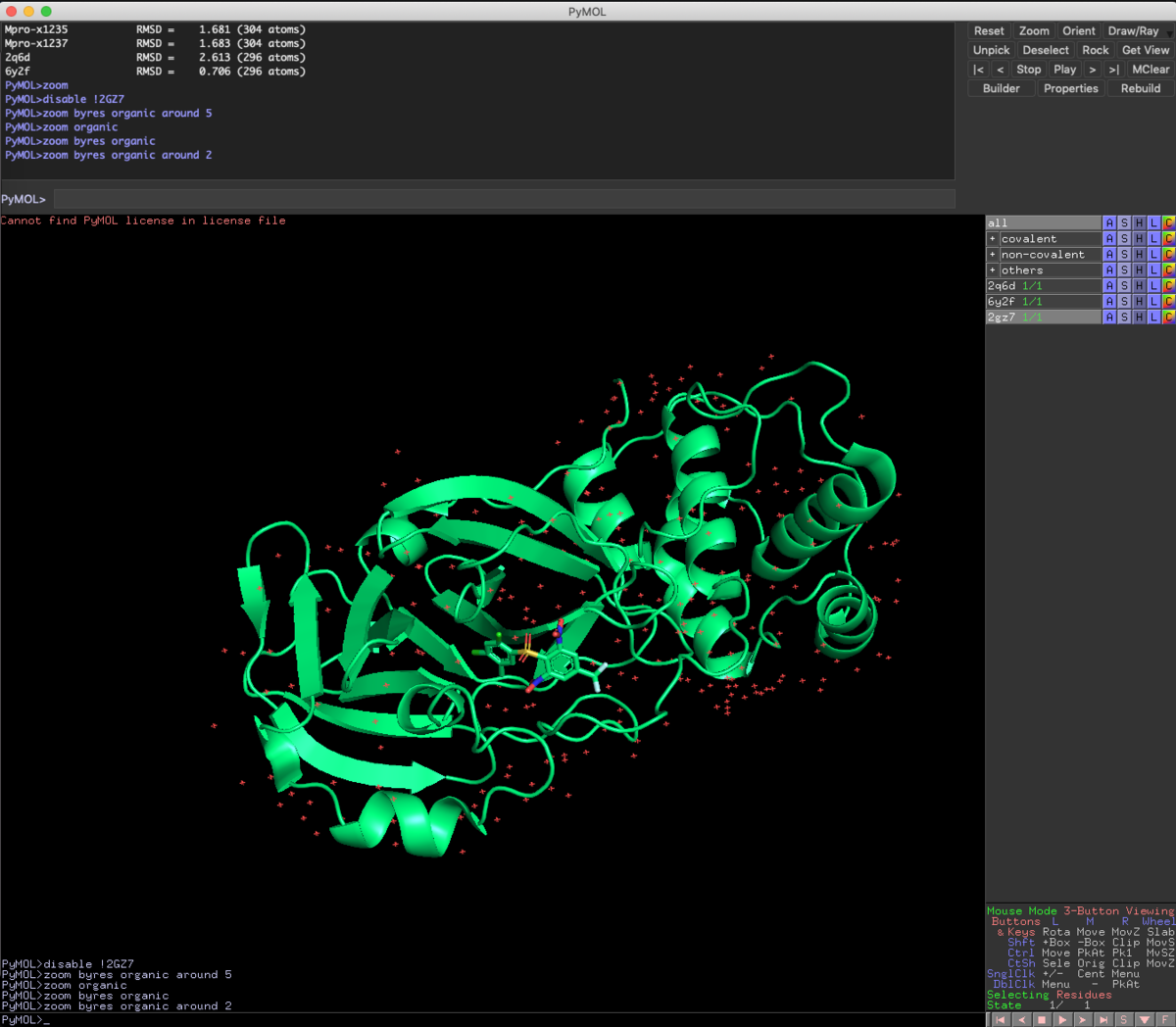
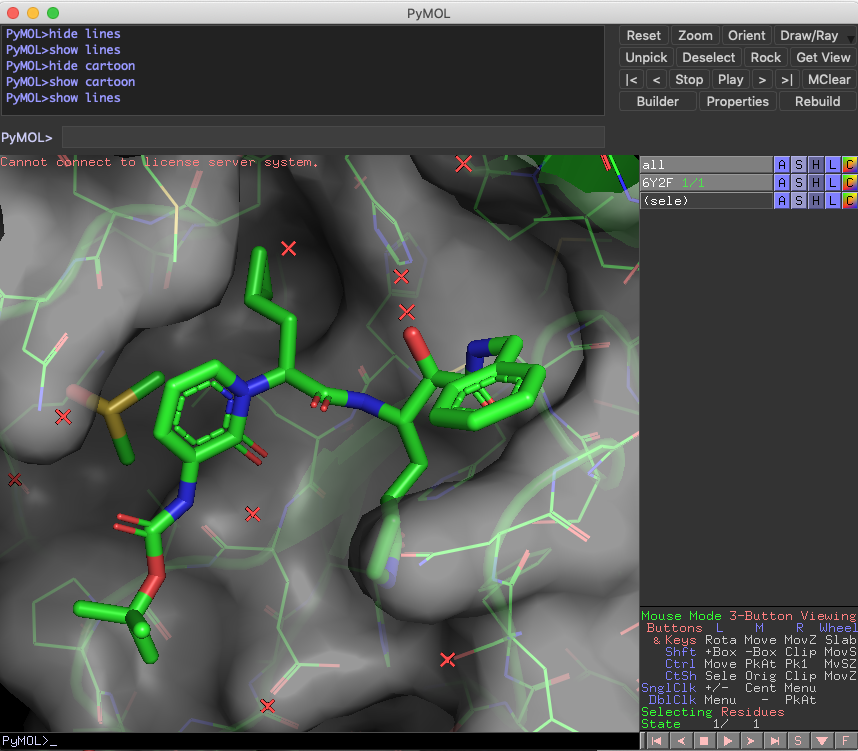
この化合物はRSC PDBに登録されているnon-covalent inhibitorです.
ちょっとLigand周辺の結合様式も見ておきましょう.
preset > ligand site > cartoon
です.
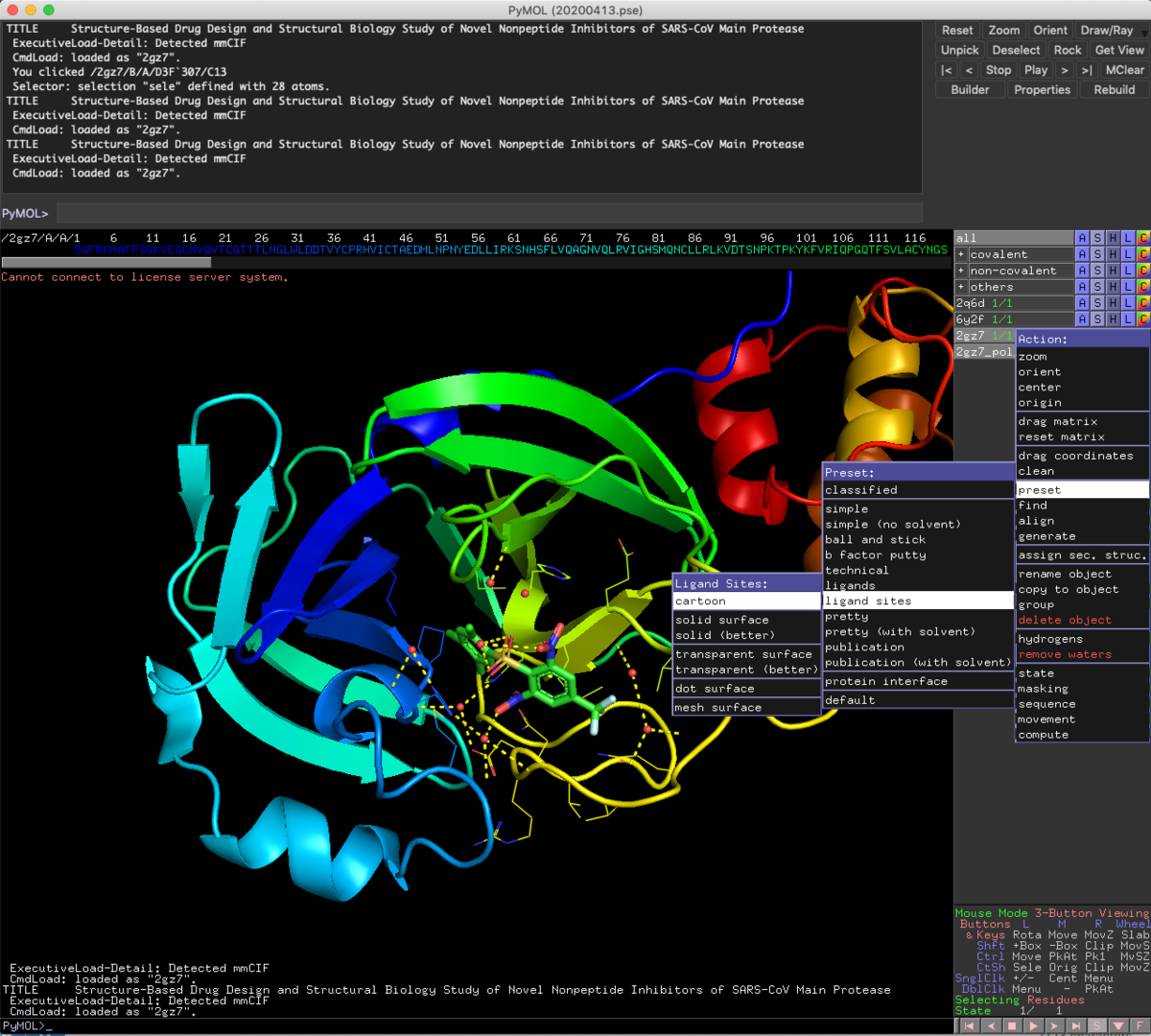
え?
タンパクと相互作用してるの,水を介したニトロ基の一個だけ?
ちょっと他の共結晶とも比較しておきましょう.

disable all
enable 6Y2F
preset > ligand site > cartoon
前回軽く触れましたが,このリガンドは基質ペプチドのミミックなのでバリバリ相互作用してますね.
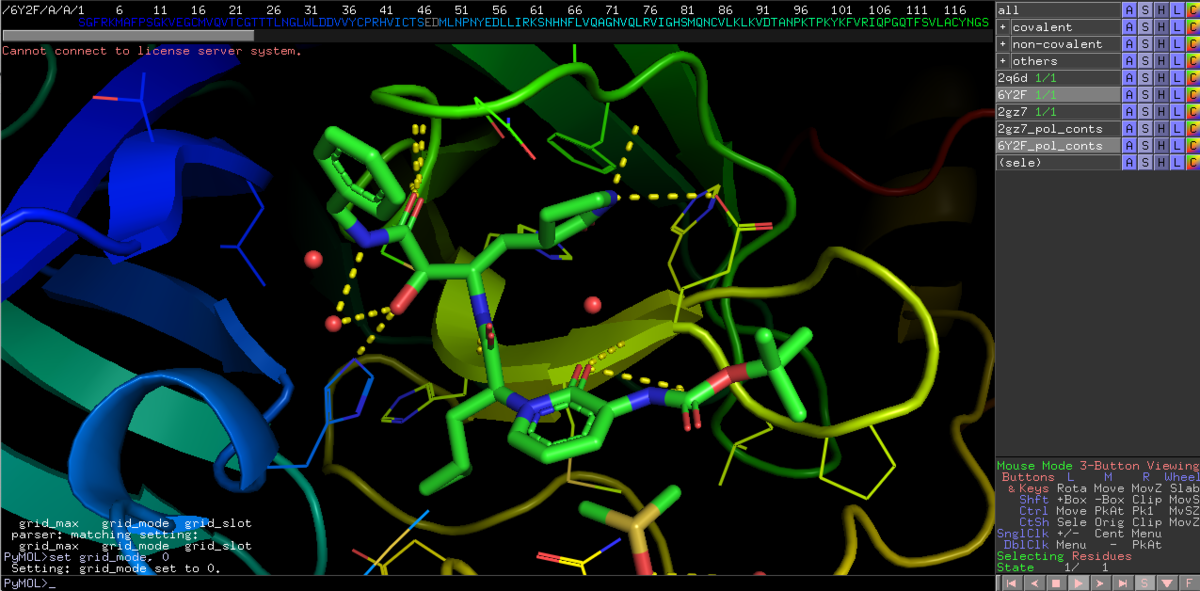
ここからはちょっと端折って行きます.
disable all
enable 6Y2F 2GZ7
set grid_mode, 1
show surface, byres organic around 5
set surface_color, gray
こんな感じに見えるはずです.
6Y2Fのリガンドである13bの赤枠脂溶性ポケットを三置換のPh基が埋めてますが,雰囲気は結構違います.スルホンとPh基の結合角もなんだか捻れてて気持ち悪いな...
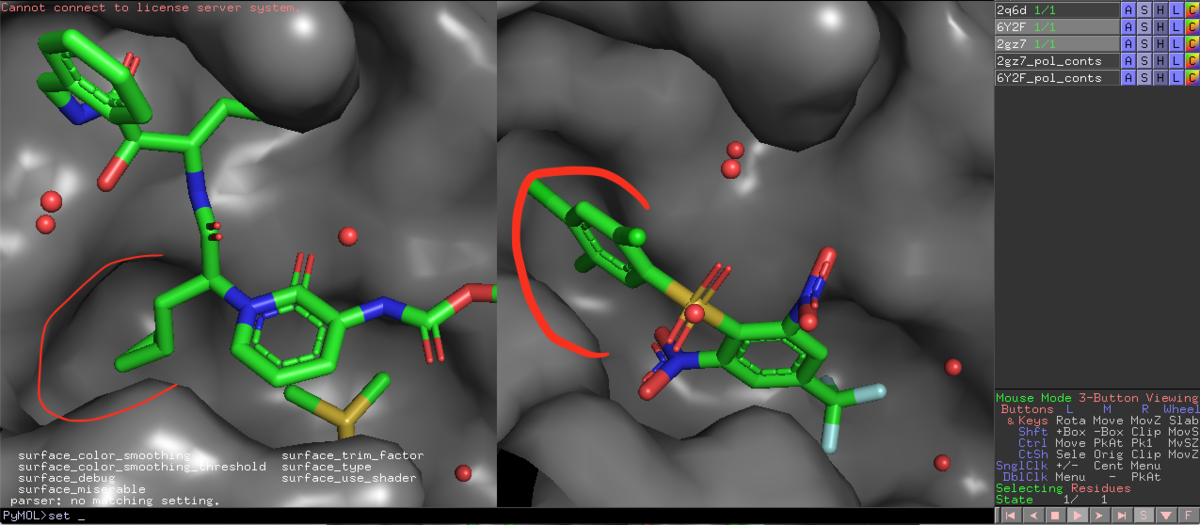
hide surface
脂溶性許容ポケットですが,他に相互作用がなさそうか見ておきましょう.すると...一応ベンゼン環の近くにHis41の側鎖イミダゾールがが平行に配向してますね.左側の6Y2Fと比較しても,フリップしてきてることがわかります.確かこいつは,システインプロテアーゼであるMproの加水分解の活性化を担っていたはずです.
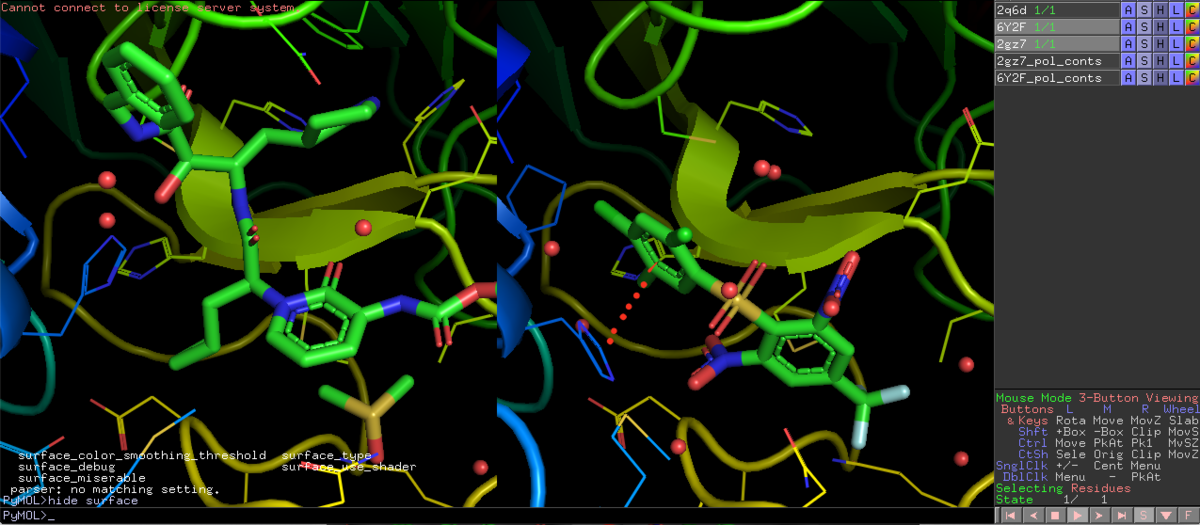
set grid_mode, 0
disable 6Y2F
右下のMouse Modeから3-Button Editingに切り替えて,リガンドと側鎖の距離を見てみましょう.3.8Åなんで,相互作用は有りそうですかね?
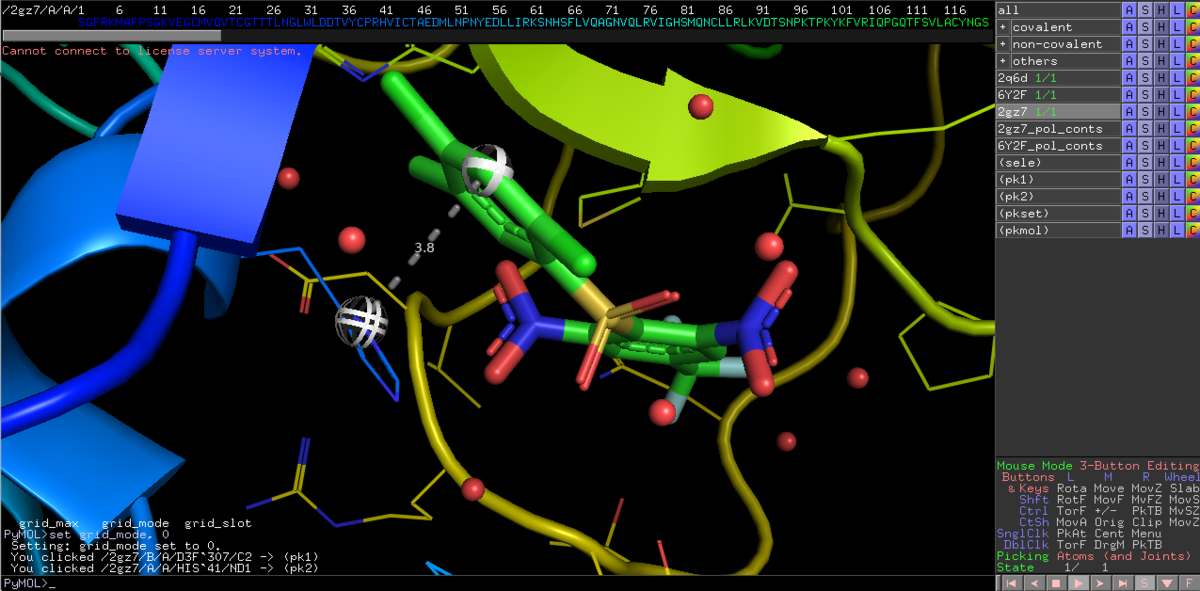
disable 2GZ7
enable 6Y2F
今度は共有結合性ペプチドミミックリガンドと,His41の距離を測ってみましょう.Cys145からの付加反応を受けて,ケトアミドはアルコールとして共結晶が得られています.この立ち上がった水酸基とイミダゾールの距離は2.7Åなので,このHis41が加水分解の触媒として機能しているとみて間違いなさそうです.2GZ7のリガンドは,このHis41を上手く捉えて活性を発現してるってことなんですかね.

ただ,活性値を見ると0.3µMと0.67µMで2GZ7の方が高活性として報告されてるんですよね...ほんまかいな...
共結晶だけでは活性値の推定は中々出来ないので何とも言えませんが,個人的には2GZ7のリガンドの活性は非特異結合か何かが上乗せされてる気がします.
今回のようなプロテアーゼは,基質認識を甘くすることで色んな配列のペプチドに切断出来ることが特徴なんだそうです.つまりここを上手く抑えることが出来れば,virusに必要なタンパクが作られなくなるので効果的と言えますが,一方で色んな基質が結合するなど,選択性の確保も課題になりそうです.上手いこと出来てますね.
enable all
hide cartoon
最後にちょっとだけ,Xchemのフラグメントスクリーニングの結果も見てみましょう.リガンドが集中している黄色で囲った部分がMproが基質を加水分解する部分な訳ですが,othersでgroup化したリガンドが別のところにくっついてます.一つ一つを確認しないと分かりませんが,阻害活性の情報もないですし,あってもアロステリックなので変異が入ったらおしまいです.non-covalentから上手く分子をデザインして,他のシステイプロテアーゼやグルタチオンと選択性を出せれば勝ちってところでしょうか.

今回はこんな感じです.さぁどんな分子をデザインしましょうか?
Dear Pymol Beginners 3 x KNIME 1.3 -リガンドのSDFファイルを書き出してみたり,リガンドの結合様式を比較してみたり-
Grüezi,
Appleで春の学生向けキャンペーンが明日までですね.
新しく出たMacBook AirとiPad Proを順番にコンボで買える日を夢見てましたが,予算の都合上,後者だけになりました笑.オマケのギフトカードで念願のApplePencil2も買うことにします!やっほい!
ひとまず8年選手のMacBook Air2012を使い続けるために,Mojaveをクリーンインストールして...画面が映らない騒動が勃発し...結局あんまり動作は軽くならずに限界を感じつつ...とかやって,ひとまずKNIMEとanaconda, Pymolをインストールし直しました.日本に帰ってからもKNIME遊びは続けられるかなぁ...
と言うわけで今回のWorkFlowでは
- RSC PDBのサイトからPDBとレポートの取得(前回までのマイナーチェンジ)
- レポートからリガンドをSDFとして出力
することが出来ます.
最後に,このWorkFlowの利用例をPymolを交えて紹介してます.
*前回,前々回の重複内容は省きます.
- WorkFlowの全体像
- PDB Connector
- PDB Saver
- Excel Sheet Appender
- RDKit To Molecule
- String Manipulation
- Group Loop Start
- SDF writer
- Variable Loop End
- String Manipulation
- String To URl
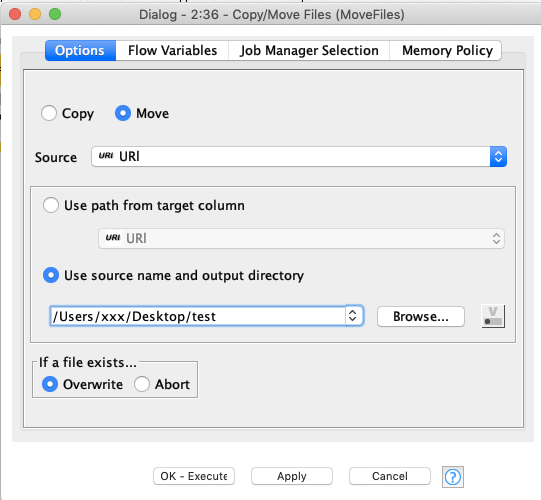
- Copy/Move Files
- 実行結果
- 利用例の紹介
- Pymolで観察
- set grid_mode, 1
- show surface, byres organic around 5
- (set surface_color, gray)
WorkFlowの全体像
上段と中段は前回とほぼ変わりません.中段先頭に置いていたMolecule Type Castは不要とわかったので削除しました.
下段のWorkFlowでは,PDBのレポートに含まれるSMILESからSDFファイルをPDB IDごとに生成しています.残念ながら今回は利用例を紹介するところまではたどり着けませんでしたが,このWorkFlowを使ってどんなことが出来るかを想像するところを宿題ってことにさせてください.(To be continued...)
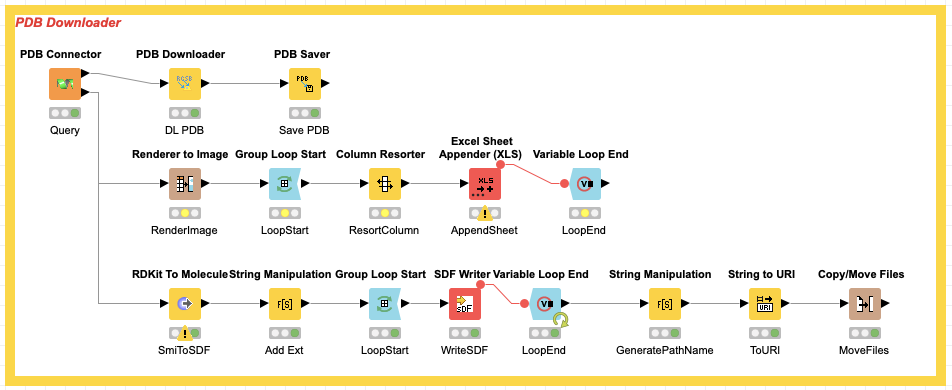
PDB Connector
IDs and Keywordsは前回と同じくSARS-CoV-2です.
PDB Saver
今回はデスクトップ上に架空ユーザー(xxx)のフォルダを指定しました.
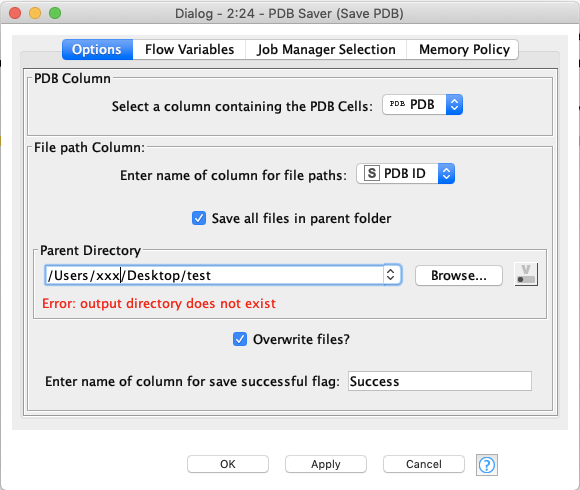
Excel Sheet Appender
今回は上記の架空ディレクトリと同じ場所を指定しています.シート名を変数にして出力するのは前回までと一緒です.

RDKit To Molecule
PDB Connectorから得られるreportのSMILES情報は既にSMILESとして機能してるみたいです.なので,このNodeを使ってSMILESをSDFに変換します.SMILESの方が都合がいいこともあるので,SourceColumnは残してます.Destination FormatはSDFですが,他のファイルも指定できます.

String Manipulation
変換したSDFファイルをリガンドごとに個別に出力したいのですが,そのまま出力するとファイルに拡張子(.sdf)が付いていません.そこで,このNodeで拡張子付きのStringを生成しておきます.ここではPDB IDとしておきます.

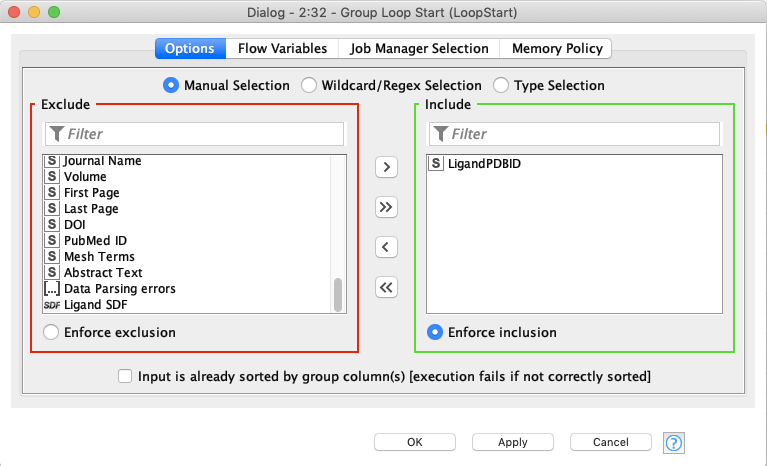
Group Loop Start
上述で生成した拡張子付きのリガンド名ごとにファイルを作りたいので,前項でAppendしたLigandPDBIDというカラムを指定してLoopをスタートします.このカラムについて1行ずつ繰り返し操作を行うってことですね.
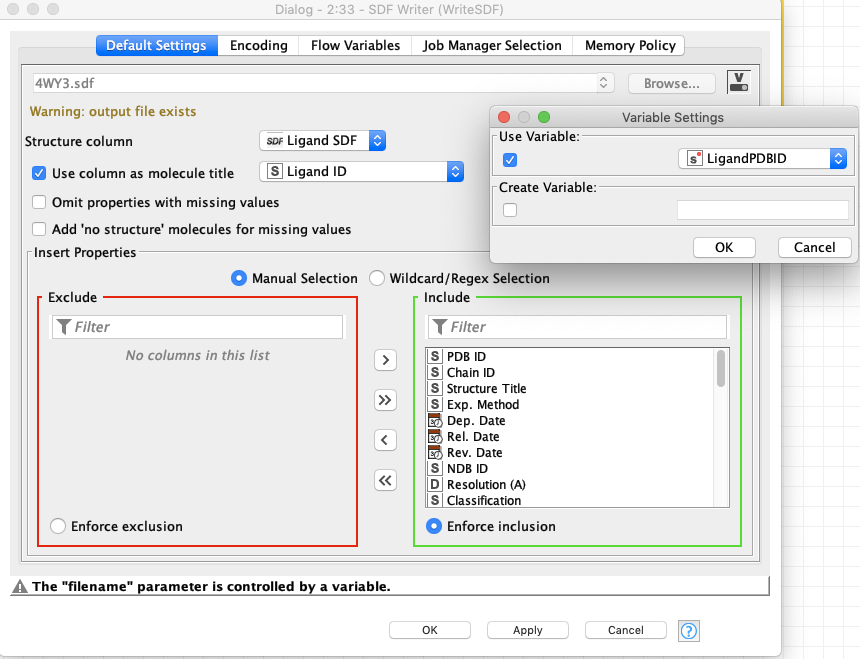
SDF writer
今回は書き出すSDFファイル名が変数になるように設定します.ファイル名の右側にあるvをクリックして,Use VariableからLoopするカラムを指定します.Use Column as molecule titleにチェックしておくと,SDFごとに分子に名前がついて後々便利だったりします.
Variable Loop End
特に設定しません.
String Manipulation
個人的にはここが今回一番トリッキーでした.
前々項のSDF Writerはお気付きの通り,出力先を指定していません.すると,私の場合,デフォルトでは/Applications/KNIME 4.1.0.app/Contents/MacOS と言うディレクトリに保存されていました.なので,このSDFファイルをPDBファイルと同じディレクトリに移動させたいと思います.単純に便利だからです.
このNodeでファイルが出力された場所を指定します.ディレクトリ末尾に/を追記した/Applications/KNIME 4.1.0.app/Contents/MacOS/ にLigandPDBIDと言うファイル名のカラムを追記していきます.

String To URl
前項で生成したStringをURlに変換します.

Copy/Move Files
最後にSDFファイルの出力先からPDFのレポートと同じディレクトにファイルを移動させます.
実行結果
以上を実行するとこんな感じになります.

べらぼうな数が吐き出されるので途中は割愛してますが,リガンドのSDF,レポート,PDBが同じフォルダに集まりました.
前回軽く紹介したPDB ID:6Y2Fなんかは,リガンドがDMSOを含めて2つとされています.

この場合,SDFファイルには2つの分子がリガンドとして出力されています.

利用例の紹介
ついでにさっきのレポートの中から,同じターゲットの別のリガンドを探してみたいと思います.main proteaseで検索をかけると,例えばペプチドアナログ以外にこんなリガンドが見つかりました.今回は出来ませんでしたが,IC50値などの活性値が報告されていると,絞り込みの方法はグッと広がると思います.

*追記
ペプチドアナログ以外を見つけたいとか書いておきながら、思いっきりペプチドアナログを見つけてますね…。深夜の疲労のせいということでご愛敬…。
Pymolで観察
このPDB ID:6W63と,6Y2FをPymolで読み込んでみましょう.

今回はそれぞれをfetchで呼び出した後,preset > ligand site > cartoonで表示しています.
次に6W63のオブジェクトから,align > to molecule > 6Y2Fで重ね合わせます.zoom organicでリガンドに照準を合わせるとこんな感じです.

6W63のリガンドは思いっきりペプチドアナログと同じBinding Siteに来てますね!次のコマンドラインでGrid表示してみましょう.
set grid_mode, 1
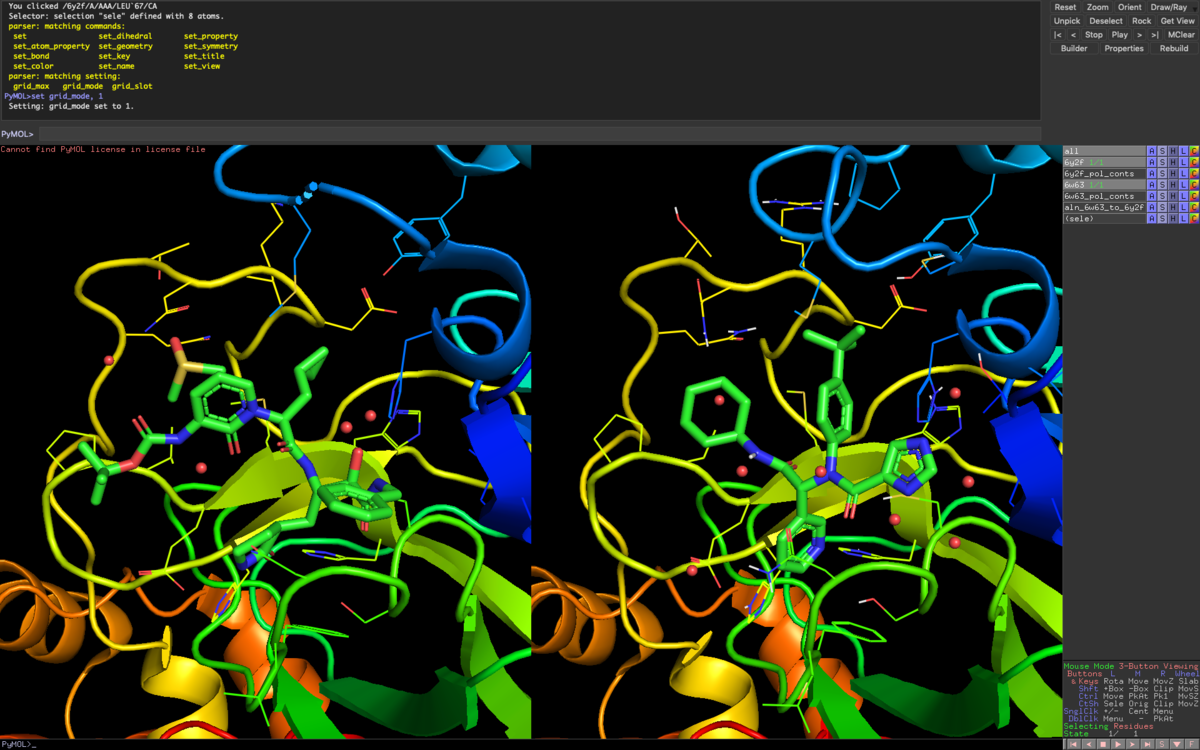
polar contactは一旦非表示にしました.ついでに6W63のnon polar hydrogenも非表示にしてみやすくしてます.次にポケットの比較をしてみます.
show surface, byres organic around 5
(set surface_color, gray)
リガンド周辺部だけSurfaceを表示しました.特に設定をしてない場合は見辛い配色だと個人的に思うので,色を灰色に変えました.

シクロプロピルメチル基が埋めていた(確か)S2ポケットと呼ばれる脂溶性空間にt-Bu基がうまくはまってますね.(下図)

S1ポケットと呼ばれるグルタミンサロゲートが埋めていた部分は3ーピリジルで置換されています.HBDいらないんですね.

件の共有結合部分を観察してみましょう.
6Y2FではC145から思いっきり共有結合しているので,その距離は1.8Åです.

一方,6W63ではC145と共有結合こそ形成していませんが,おそらく該当するであろうアミドとの距離は3.4Åです.プロテアーゼでは通常,コアの反対側にあるヒスチジンからHBDのアシストを受けて加水分解の活性化を受けたりするんですが,今回はそういった残基も特には見当たりませんでした.原子間距離自体は近いので,リガンドの結合にこのアミドは何かしら関与してそうな気はするんですけどね.

他にも細かい相互作用解析はあるんですが,今回はこの辺で切り上げようと思います.
出力したSDFファイルの使い道もWorkFlowが出来たら紹介したいと思います.
Dear Pymol Beginners x KNIME 1.2 -PDBのReportファイルをExcelのシートごとに分けてみたり-
Grüezi,
コロナ騒動がおさまりませんが,私はグラント応募の準備がひと段落したのでKNIME遊びを更新しました.
今回のUpdateはこんな感じです.
- PDB Downloader WorkFlow
- PDB Connector
- Group Loop Start
- Column Resorter
- Excel Sheet Appender (XLS)
- Variable Loop End
前回までにPDB Connetctorで取ってきたPDBと,その付随情報をエクセルで取得するWorkFlowを紹介しました.
今回はLoopを使って,このExcelファイルをもう少し体裁よくします.
Loopを回した結果はこんな感じです.
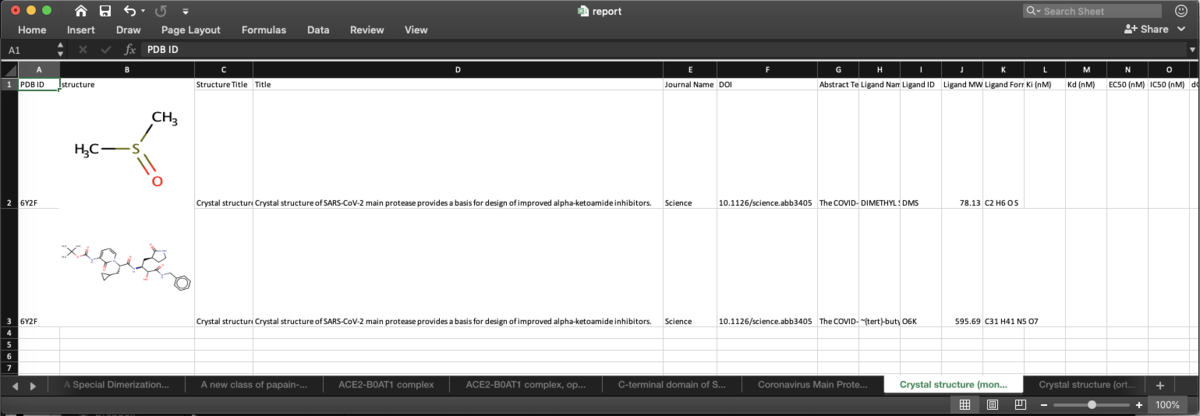
私はケミストなんでリガンドの構造情報が先頭にある方が親近感が湧きます.
阻害剤であればIC50の情報なんかもあると参考になります.
あとはアブストラクトなんかも取得しておくといいかもしれません.
この上から2番目の構造は,Scienceに報告された共有結合性阻害剤で,c&enなんかでも紹介されていました.
ターゲットやキーワードからPDBを検索した時に,リガンドの構造や阻害活性の情報,アブストラクトなんかを俯瞰できると便利かなと思います.今回はやたら検索結果が出てきたので,もう少しうまく絞り込んだ方が良さげですが...
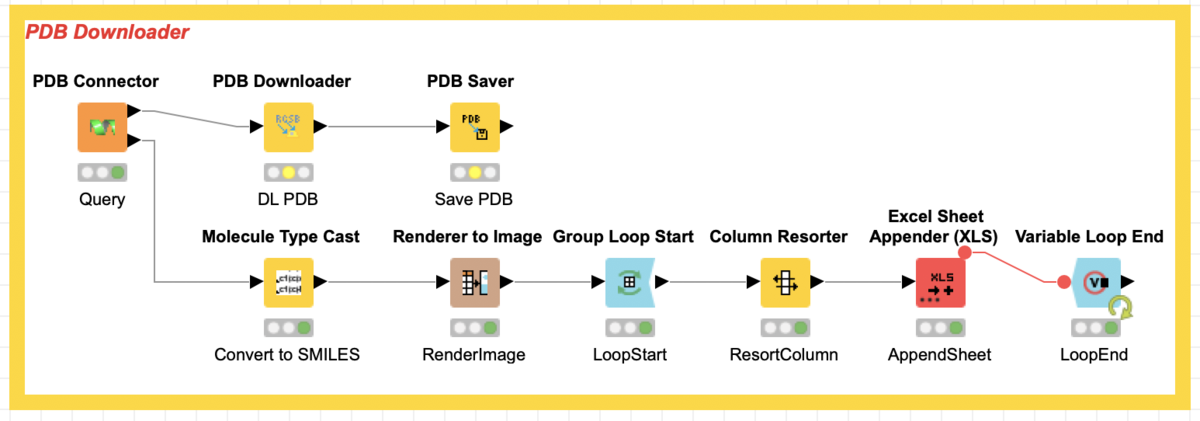
PDB Downloader WorkFlow
まずはWorkFlowをまんま載せるとこんな感じです.
PDB Connector
内容は前回紹介した通りなんで割愛しますが,今回は少し広めのSARS-CoVで検索をかけてみたいと思います.

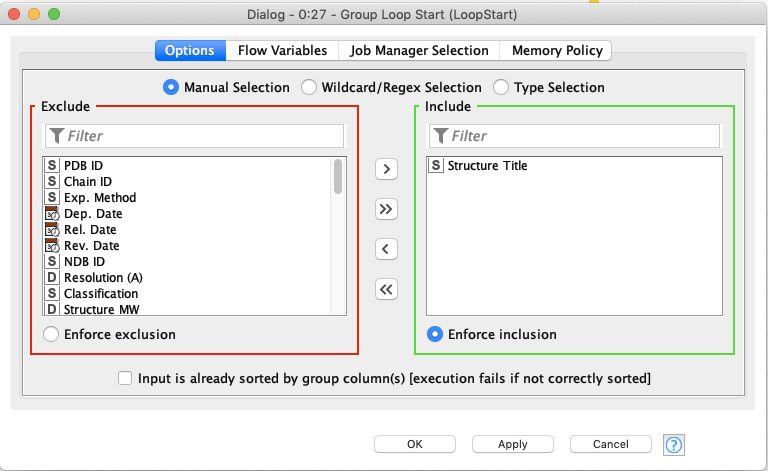
Group Loop Start
一つ前のRenderer to ImageというNodeでは下のような結果が得られます.一つの文献の中で複数のリガンドが報告されることはよくあることですが,このままExcelに吐き出してしまうと情報量が多くて,個人的には見る気が湧きません.
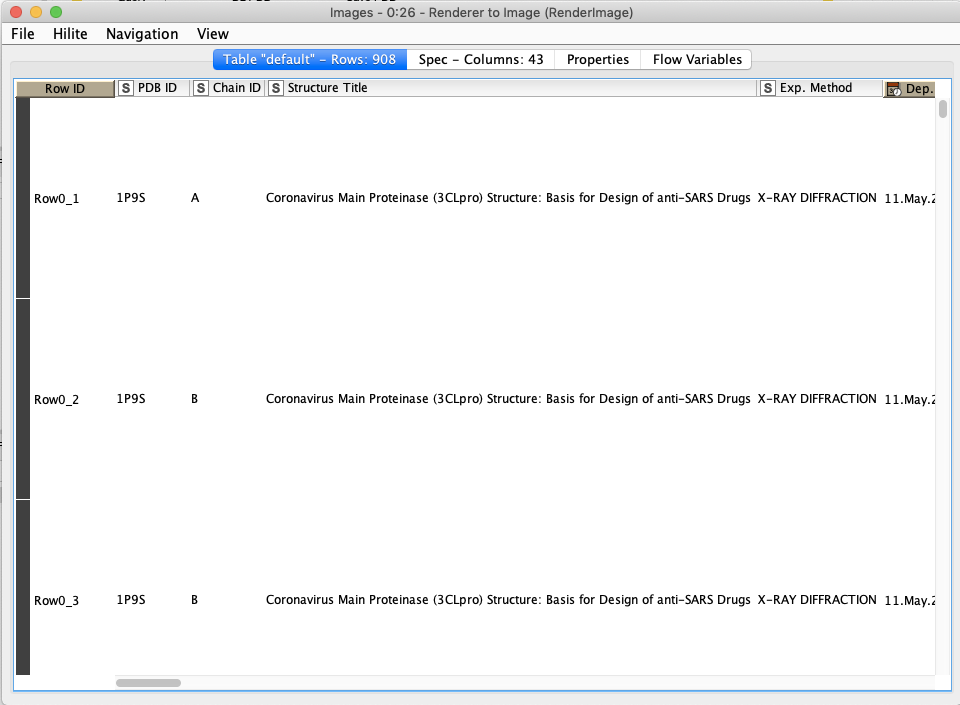
そこで,Group Loop Start Nodeを使って文献ごとにグループ化して,その結果をシートで分けてしまおう,というお話です.
設定からはGroup化したい項目を選択します.複数選択するとより細かくGroupingされます.今回はシートが増えてかえって見づらくなるので,シンプルにStructure Titleだけにしました.
Column Resorter
カラムの並びを好みの順番にします.この辺は好みにあわせて変えるといいと思います.
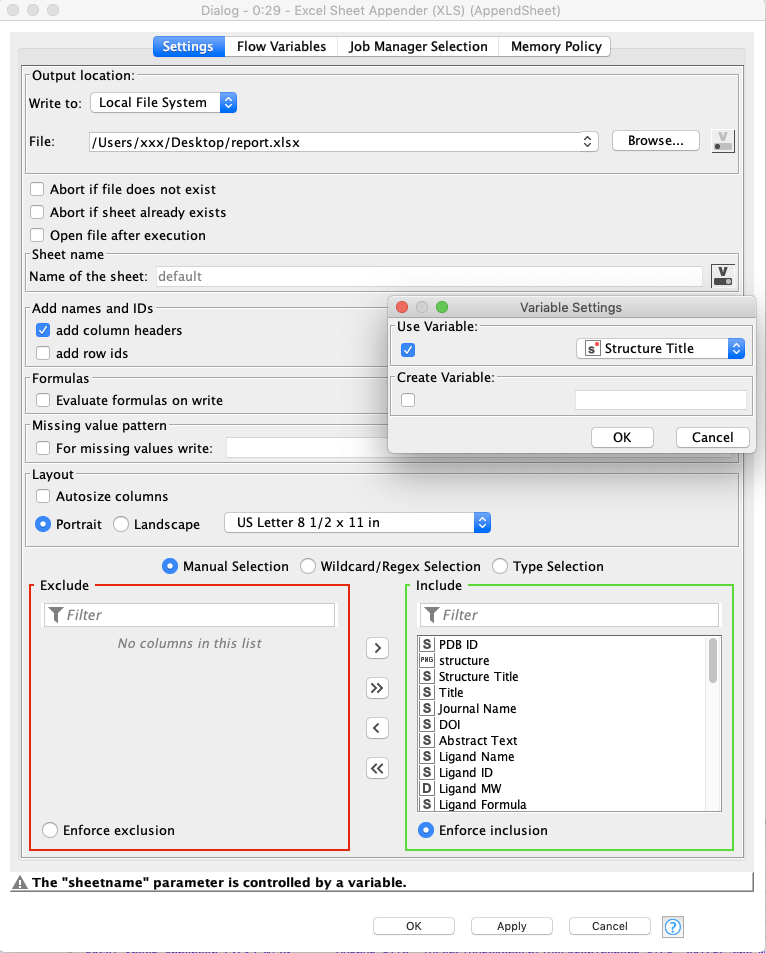
Excel Sheet Appender (XLS)
このNodeでGroupingの結果をエクセルの新規シートとしてAppendします.
Sheet nameの右端にあるvのボタンを押しすことで,前項のGroup Loop Startの結果を変数として扱うことが出来ます.Use Variableにチェックを入れて,どの項目でGroup化するかを選択します.今回はStructure Titleです.
Add names and IDsのadd column headersにもチェックを入れておくと,実行結果のExcelが見やすくなります.
Variable Loop End
最後にここがLoopの終わりだよと宣言するNodeです.特に何も設定してません.
KNIMEを使い始めた頃,「Loop系のnodeは繰り返し処理に使う」というのは知ってはいたんですが,イマイチ使いこなせていませんでした.こういう簡単な処理でLoopを使ってみるのが理解を早めてくれますね.
最後になりますが,今回のWorkFlowはこちらの記事を参考に作成しました.
先人にはいつも大変にお世話になってます.この場を借りて感謝申し上げます.
SDFファイルやPDBファイルも文献ごとにフォルダ分け出来ないかも勉強中です.
KNIMEのVersionあげたら動かなくなったNodeもあって四苦八苦してますが,実験が出来ないので,いい機会だから色々試してみようと思います.
それではWiedersehen.
Dear Pymol Beginner x KNIME -KNIMEを使ってPDBファイルをまとめてDLしてみたり-
Grüezi,
世間ではコロナウィルスが大流行ですが,皆さんいかがお過ごしでしょうか.
さて,この数週間でCOVID-19関連の構造情報が複数公開され,各PDBサイトにも特集が組まれています.
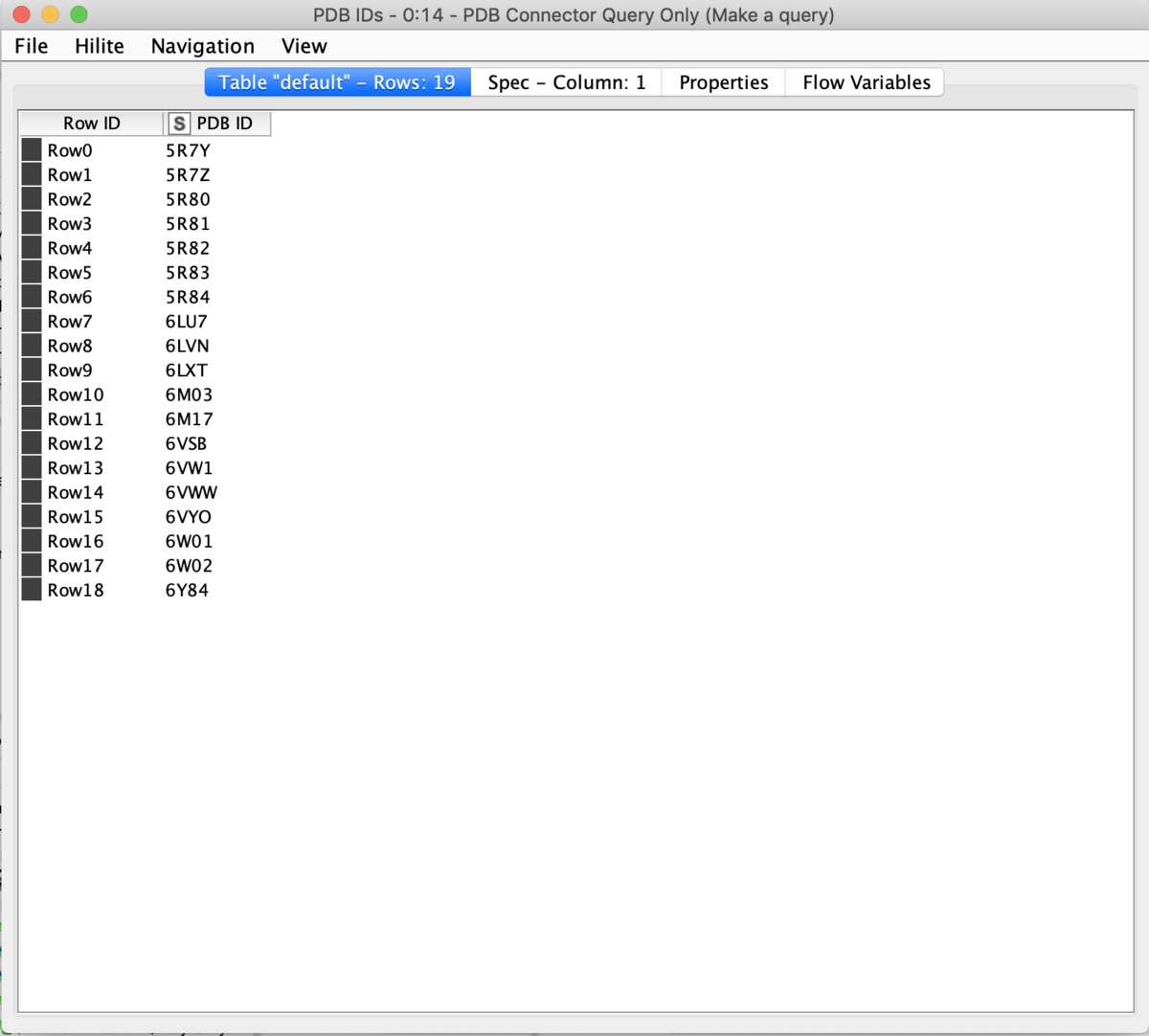
2020/3/14現在でRCSB PDBのサイトには19個の構造情報が公開されています.19個の結晶構造を一変にダウンロードするのは面倒なので,一発で済ます方法を紹介します.*但し,KNIMEユーザーに限る.
*2020/03/18 Molecule Type Cast Nodeは不要だったので削除しました.
本日のコンテンツはこちら.
- KNIMEのWorkFlow
- PDB Connector
- PDB Downloader
- PDB Saver
- Renderer to Image
- Column Resorter
- Excel Writer
KNIMEのWorkFlow
まんま載せるとこんな感じです.

Community Nodes-Vernailsをインストールしておくと,RCSB PDB Toolsというノードが見つかります.それを並べただけのシンプルな構成です.
レポートをLoopで見やすく出来ないかは勉強中です...
PDB Connector
このノードを使うと,RSCB PDBのAdvanced Search(http://www.rcsb.org/pdb/search/advSearch.do)と同じ検索をかけることが出来ます.設定を少し見てみましょう.
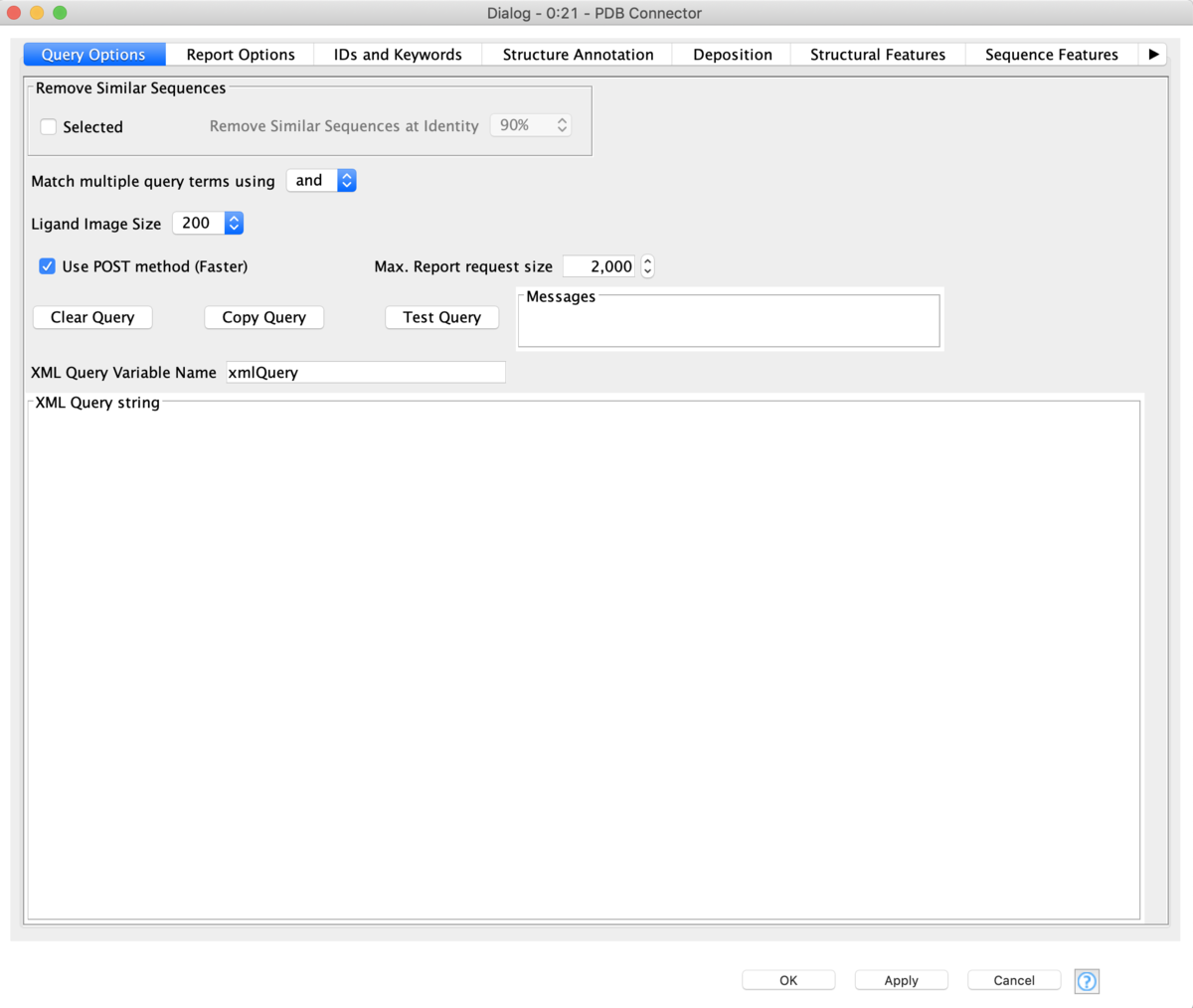
Query Optionsは一番最初に私がハマったポイントです.
Remove Similar SequencesのSelectedからチェックを外してください.そうしないと,同じ報告内の異なるリガンドの共結晶構造ははじかれてしまいます.

Report OptionsでPDB IDからStructure Titleや掲載日など,簡単な情報を取ってきてくれます.結果はノードの下から出力されます.
Select Reportから,上の方にあるCustomizable Tableを選択し,必要な項目をチェックしていきます.とりあえずざっくりとStructure Summary,Ligands,Binding Affinity,Primary Citation辺りをチェックしておきます.後から変更できますし,Row Filterとかでも絞り込めます.基本の設定はここまでです.

IDs and Keywordsから検索したいQueryを作成します.Text Searchをアクティブにするのがシンプルで使いやすいかと思います.今回は話題のcovid-19と入力しておきます.
他にもFilterをかけたい時は他のタブから条件を絞り込んでいけばOKです.解像度や測定方法など,結構細かいことを絞り込めます.
設定を完了して実行すると下記のようにPDB IDとReportが返ってきます.
PDB Downloader
その名の通りでPDBをダウンロードしてきます.この時点で保存はされてません(!)
特に設定不要ですが,ダウンロードしたい項目を増やしたり変更したい時にはいじります.
PDB Saver
こちらもその名の通りで,DownloaderでダウンロードしたPDBをローカルに保存します.保存先のディレクトリに,以下のレポートも保存するといいと思います.
Renderer to Image
SMILESの構造情報は,そのままではExcelで単なる文字列としてしか表示できないので,このNodeでさらにPNGファイルに変換します.

SmilesをMarvinを使ってPng形式で200x200の大きさにして,Structureカラムに出力しますよ,という設定です.
Column Resorter
出力されるReportのカラムを並び替えておきます.
構造情報を前の方に並び替えておく方が,Chemist的にはなんとなくフレンドリーです.
Excel Writer
PDBと同じディレクトリを指定しておくと,色々と作業がしやすくなると思います.
以上,ここまでを一旦流してみると,2020/03/15現在登録されている19個の結晶構造と,そのレポートを一度に取ってこれました.レポートをもう少し見やすくまとめるのにLoopを使えないかなぁ...