ChEMBLのデータを整える
皆さんは長期休暇の宿題は計画通り進める派でしたか?先にやる派?後でまとめて派?
私はというと、宿題とかない小学校でした笑
とは言っても子どもの学校は宿題があるので、どう接するのが正解なのかいつも悩ましいです。
今回はジジババの家に行く前に全て片付けてくれたので大満足の出だしでした。
と言うわけで?次は大人の番です(え?
先日JAKiの紹介したとこでしたが、
JAKiってキナーゼのわりには構造がシンプルでも活性と選択性が出てそうで、ケミストはとっつきやすいのかもですね!
— へい🍅 (@HiGoing) July 3, 2022
大きい買収があったりでほんと開発が活発なんだなーとか思わせるニュースを目にしました。
TYKって結構前のターゲットだった気がしてたんですが、JAKの仲間だったんですね(遅。
kinaseは各社の積み重ねが身を結んだような報告をよく目にします。
ケミストは確かにとっつきやすいのかもなーと思いますが、これからとっつく余地がどれだけあるかが創薬研究では重要かなと思います。
と言う名目で、オープンデータベースのChEMBLを使ってリガンドの外観を捉えてみたいと思います。
- Targetを検索して化合物データをダウンロード
- Import Python Libraries and Load ChEMBL Data File
- Data Cleansing
- 構造式と骨格情報の追加
- 文献数と骨格情報のメモ
- Descriptorを追加
- Summary of Data Table
Targetを検索して化合物データをダウンロード
今回はChEMBLでシンプルにTYK2を検索しました。
8 Targetsの中から一番上のCHEMBL3553を選択します。
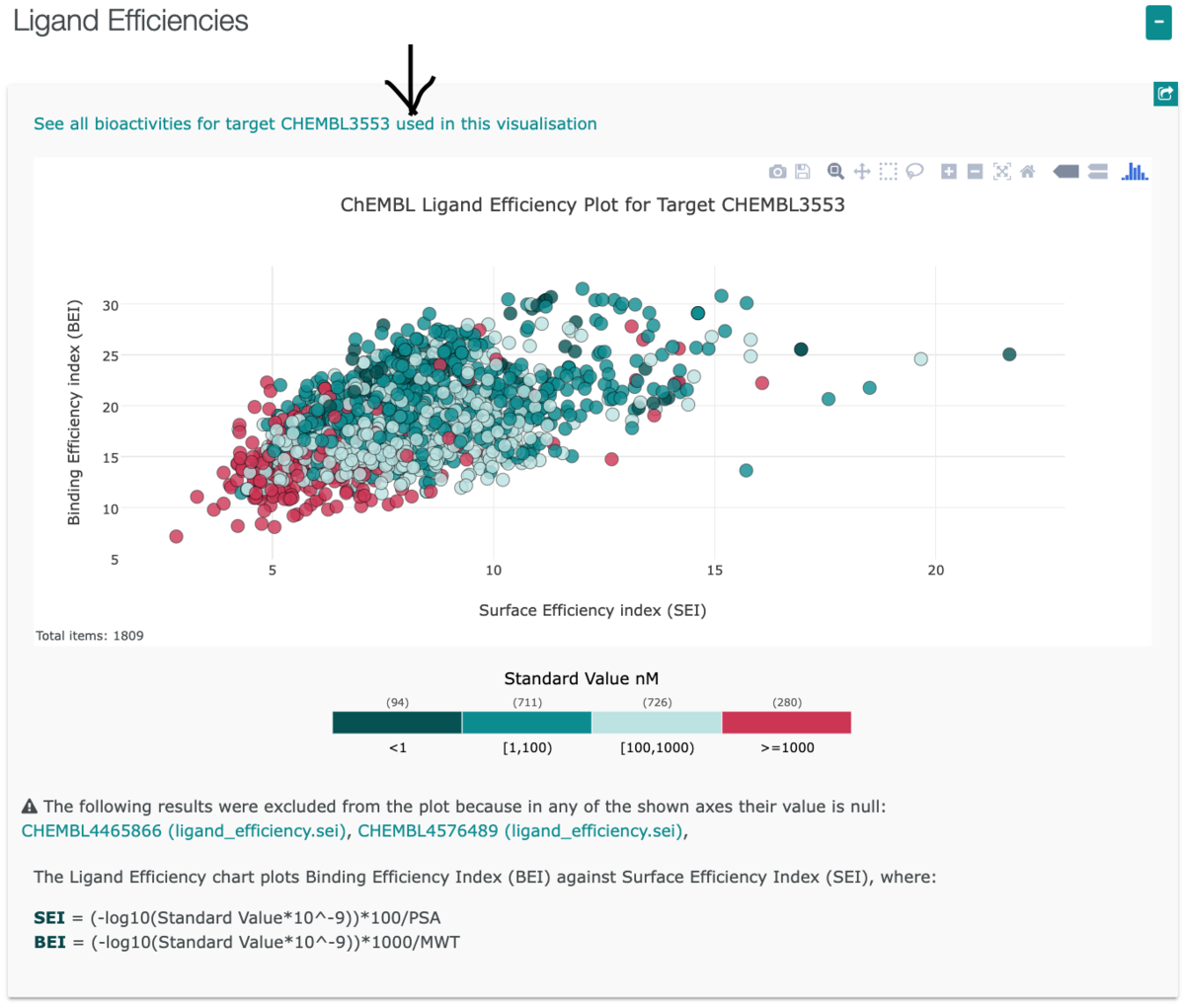
 下の方にあるLigand Efficienciesの
下の方にあるLigand Efficienciesの
See all bioactivities for target CHEMBL3553 used in this visualization
っていう項目をクリックしてBrowse Activities を開きます。
 右の方にあるCSVのボタンをクリックすると化合物と活性情報のcsv fileをダウンロードできるようになります。
右の方にあるCSVのボタンをクリックすると化合物と活性情報のcsv fileをダウンロードできるようになります。
2022.12.27現在1,811 recordsでした。

Import Python Libraries and Load ChEMBL Data File
# import libraries import pandas as pd import platform from rdkit.Chem import AllChem as Chem from rdkit.Chem import Descriptors, Descriptors3D, QED, Draw, PandasTools from rdkit.Chem.Draw import IPythonConsole from rdkit import DataStructs print('python version: ' + platform.python_version()) print(f'rdkit version: {rdBase.rdkitVersion}') # ChEMBLからDLしたファイルの読み込み df = pd.read_csv('./2022TYK2.csv',sep=';',)
# output python version: 3.10.6 rdkit version: 2022.03.5
このままdfでデータを開くとわかりますが、数値データの中にstringのNoneがあったり、Smiles stringの中にNanがあったりでpandasの処理を実行できませんでした。
と言うわけで次項のクレンジングが必要になります。
Data Cleansing
# None(string)を0に変換 df = df.replace('None', '0') # datatypeを変更 df['MW'] = df['Molecular Weight'].astype(float) df['pChEMBL Value'] = df['pChEMBL Value'].astype(float) df['AlogP'] = df['AlogP'].astype(float) df['Ligand Efficiendy SEI'] = df['Ligand Efficiency SEI'].astype(float) df['Ligand Efficiendy BEI'] = df['Ligand Efficiency BEI'].astype(float) df['Ligand Efficiendy LE'] = df['Ligand Efficiency LE'].astype(float) df['Ligand Efficiency LLE'] = df['Ligand Efficiency LLE'].astype(float) df['Document Year'] = df['Document Year'].fillna('2025').astype(int) df['#RO5 Violations'] = df['#RO5 Violations'].astype(int) df['Molecule Max Phase'] = df['Molecule Max Phase'].astype(str) # SmilesカラムのNanを空に変換 df['Smiles'] = df['Smiles'].fillna('')
AlogPとか計算できなかった場合Noneが入ってしまってるらしく、何かしらの値を入れないとvisualizationとかできません。
今回は0でreplaceしました。なので、可視化した時など値が0のものは注意が必要です。
Document Yearが入ってないものも時々あったりするんですが、わからないことがわかればいいので2025でreplaceしました。こちらも可視化した時にそういう理解が必要です。
(他にいい方法があったら教えてください)
構造式と骨格情報の追加
# SMILESからMol fileの追加 PandasTools.AddMoleculeColumnToFrame(df,'Smiles','Molecule_Image',includeFingerprints=True) # Murcko Scaffoldの追加 PandasTools.AddMurckoToFrame(df, molCol='Molecule_Image', MurckoCol='Murcko_SMILES') PandasTools.AddMurckoToFrame(df, molCol='Molecule_Image', MurckoCol='Murcko_generic_SMILES', Generic=True) PandasTools.AddMoleculeColumnToFrame(df, molCol='Murcko_Mol', smilesCol='Murcko_SMILES') PandasTools.AddMoleculeColumnToFrame(df, molCol='Murcko_generic_Mol', smilesCol='Murcko_generic_SMILES') # 立体構造の追加 # df['AddHs'] = ddf['Molecule_Image'].apply(lambda x : AllChem.AddHs(x)) # df['3D'] = ddf['AddHs'].apply(lambda x : AllChem.EmbedMolecule(x, AllChem.ETKDGv3())) # df['3D'] = ddf['AddHs'].apply(lambda x : AllChem.RemoveHs(x)) # df['NPR1'] = ddf['AddHs'].apply(lambda x : Descriptors3D.NPR1(x)) # df['NPR2'] = ddf['AddHs'].apply(lambda x : Descriptors3D.NPR2(x))
SmilesをMol filesにしておくとDataFrameのoutputだったりDraw.MolToImageで構造式を描画できます。
PandasToolsは.apply()いらないから楽でいいですね。
文献数と骨格情報のメモ
# 文献数 print("the number of Document ChEMBL ID is " + str(len(df['Document ChEMBL ID'].unique()))) # the number of Document ChEMBL ID is 134 # 骨格数情報 SMILESじゃないとなぜか反映されない print("the number of Murcko generic scaffold is " + str(len(df['Murcko_generic_SMILES'].unique()))) # the number of Murcko generic scaffold is 419 print("the number of Murcko scaffold is " + str(len(df['Murcko_SMILES'].unique()))) # the number of Murcko scaffold is 651
どれくらい文献があって、SARが展開されてきたか、、、これだけじゃわからないので可視化したいですね。。。
Descriptorを追加
# descriptorの追加 df['TPSA'] = df['Smiles'].apply(lambda x : Descriptors.TPSA(Chem.MolFromSmiles(x))) df['Ct HA'] = df['Smiles'].apply(lambda x : Descriptors.HeavyAtomCount(Chem.MolFromSmiles(x))) df['Ct F'] = df['Smiles'].apply(lambda x : x.count('F')) df['HBD'] = df['Smiles'].apply(lambda x : Descriptors.NumHDonors(Chem.MolFromSmiles(x))) df['HBD_Ro5'] = df['Molecule_Image'].apply(lambda x : rdMolDescriptors.CalcNumLipinskiHBD(x)) df['HBA'] = df['Smiles'].apply(lambda x : Descriptors.NumHAcceptors(Chem.MolFromSmiles(x))) df['HBA_Ro5'] = df['Molecule_Image'].apply(lambda x : rdMolDescriptors.CalcNumLipinskiHBA(x)) df['Ct RB'] = df['Smiles'].apply(lambda x : Descriptors.NumRotatableBonds(Chem.MolFromSmiles(x))) df['Ct Ar'] = df['Smiles'].apply(lambda x : Descriptors.NumAromaticRings(Chem.MolFromSmiles(x))) df['Fsp3'] = df['Smiles'].apply(lambda x : Descriptors.FractionCSP3(Chem.MolFromSmiles(x))) df['QED'] = df['Molecule_Image'].apply(lambda x : QED.qed(x))
RDkitを使ってSmilesからDescriptor追加してみました。気が向いたら使ってみます。
df['new column'] = df['target column'].apply(lambda x : f(x))で計算カラムを追加するやり方はだいたいいつも忘れちゃいます。。。
こちらを参考にしてもらうといいかもです。
Summary of Data Table
df[df['Molecule Name'].notna()].filter(items=[ 'Molecule Name', 'Molecule ChEMBL ID', 'Molecule_Image', 'Molecular Weight', 'pChEMBL Value', 'AlogP', 'Ligand Efficiency LLE', '#RO5 Violations', ]).head(3)
名前がついてるような化合物は上市薬だったりツールだったりで色々情報が多いと期待されるので、どんな感じか要約テーブルで見てみます。
df.filter(items=[column name])でカラムの順番を入れ替えれるのも地味に便利でした。

以上、とりあえずChEMBLからとってきたデータを解析できる状態に加工するところまででした。 KNIMEでも同じことはできるんですが、VScodeとかJupyterの方が早いし見やすいので気に入ってます。
次回に続きます